2020-06-16 理化学研究所,慶應義塾大学薬学部
理化学研究所(理研)生命医科学研究センターメタボローム研究チームの津川裕司研究員(環境資源科学研究センターメタボローム情報研究チーム研究員)、有田誠チームリーダー(慶應義塾大学薬学部教授)らの国際共同研究グループは、生命活動に必須の分子である脂質の構造多様性を明らかにするための革新的なノンターゲットリピドミクス[1]解析技術を開発しました。
本研究成果は、生体を構成する脂質の「質」(リポクオリティ[2])の違いを的確に捉えることを可能にするものであり、生命科学研究のさまざまな分野における複雑な生命現象の理解に貢献すると期待できます。
近年、質量分析法[3]を用いて脂質構造の多様性を捉えようとする研究が盛んに行われています。しかし、質量分析ビックデータは複雑かつ膨大であり、またMS/MSスペクトル[4]から脂質構造を包括的に解明することは困難なことから、その全貌はほとんど明らかになっていませんでした。
今回、共同研究グループは、ヒトおよびマウスの臓器・組織・細胞、腸内細菌叢などの脂質成分を網羅的に捉えるため最先端の質量分析手法によるノンターゲット分析[1]を行いました。得られた質量分析ビッグデータを解析するための情報処理技術(質量分析インフォマティクス)を開発した結果、既存の研究に比べおよそ10倍に上る約8,000種の脂質分子構造の存在が明らかになり、脂質構造の多様性を捉えることが可能になりました。
本研究は、科学雑誌『Nature Biotechnology』オンライン版(6月15日付:日本時間6月16日)に掲載されます。
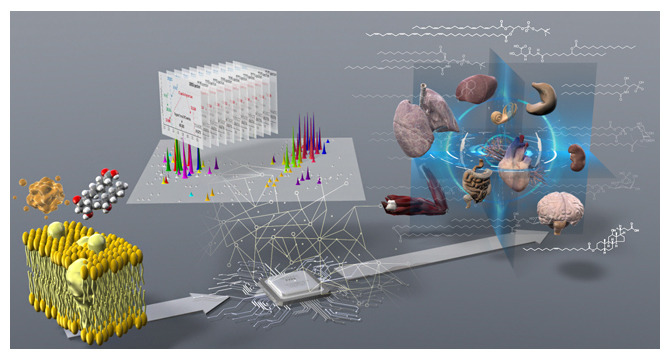
脂質分子種(左)を質量分析・情報科学で解析し(中央)生体組織中のリポクオリティを包括的に捉えた
背景
生命システムのさまざまな機能・役割を担うべく、生体内には多くの種類の脂質分子種が存在し、その多様性がさまざまな生命現象や機能制御に関わっていると考えられています。例えば、細胞膜の主要構成成分であるリン脂質の一つであるホスファチジルコリン(PC)に分類される脂質クラスには、およそ1,500種類の構造多様性があることが知られています(図1)。
これは、PCを構成する脂肪酸の組み合わせの多様性によるものです。脂肪酸には、飽和脂肪酸(パルミチン酸やステアリン酸)、不飽和脂肪酸(オレイン酸など)に加えて、多価不飽和脂肪酸(アラキドン酸、エイコサペンタエン酸、ドコサヘキサエン酸)など多くの種類があります。PCには、最大で二つのアシル基(脂肪酸)が結合しており、その組み合わせによりさまざまな構造をとることが可能です(図1)。
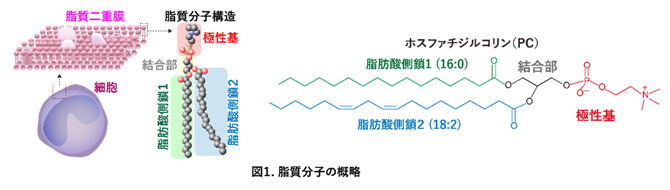
図1 脂質分子の概略
細胞は「脂質二重膜」と呼ばれる脂質の膜で覆われており、その構成成分である脂質分子は極性基と脂肪酸、そしてこれらをつなげる結合部の組み合わせにより、多様性および生命システムを維持している(左図)。細胞膜を構成する主要な脂質クラスの一つであるホスファチジルコリン(PC)にパルミチン酸(炭素数16、二重結合数ゼロ:緑線)およびリノール酸(炭素数18、二重結合数2:青線)が結合した具体的な構造を右図に示す。
また現在、構造的特性により分類されている脂質クラス(サブクラス)には301種類あることが知られており、予測構造を含めて4万種を超える脂質がこの生物界に存在すると見積もられています(図2)。このような各々の脂質構造の質の違い、つまりリポクオリティの多様性に着目し、その多様性やバランスの生物学的意義が解明できれば、これまで未解明であった複雑な生命現象の理解につながると期待されています。
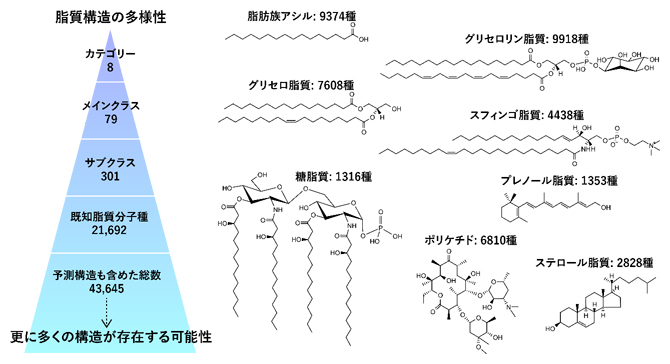
図2 脂質構造の多様性
脂質分子種は大きく分けて8個のカテゴリーに分類され、極性基や機能などにより分類されるサブクラスとしては301脂質クラスが定義されている(左図。脂質研究者により国際的に維持されているLIPID MAPSデータベースより)。右図には、8個のカテゴリーの名称および各カテゴリーに登録されている分子種の数を示す。これまでの生化学的な研究で存在が確認されている脂質は2万種、そして予測構造も含めた総数は4万種を超える構造が登録されているが、これらを包括的に捉える手法はいまだ確立されていない。
一方、近年目覚ましい進歩をとげている質量分析技術を用いて脂質構造の多様性を捉えようと、世界中で研究が盛んに行われています。しかし、質量分析装置により得られるビッグデータは複雑かつ膨大であり、さらには得られたMS/MSスペクトルから脂質構造を包括的に解き明かすことは困難であることから、その多様性の全貌はほとんど明らかになっていませんでした。
そこで、共同研究グループは、このような脂質多様性を広範囲かつ明確に識別し、それぞれのリポクオリティの違いを浮き彫りにできる網羅的脂質分子解析基盤の構築を試みました。
研究手法と成果
脂質の多様性を包括的に捉えるために、液体クロマトグラフィータンデム質量分析(LC-MS/MS)[5]もしくはイオンモビリティータンデム質量分析(IM-MS/MS)[6]が頻用されています。さらに、脂質の分子構造は、脂質特有のMS/MSスペクトルを読むことで解き明かされます(図3)。
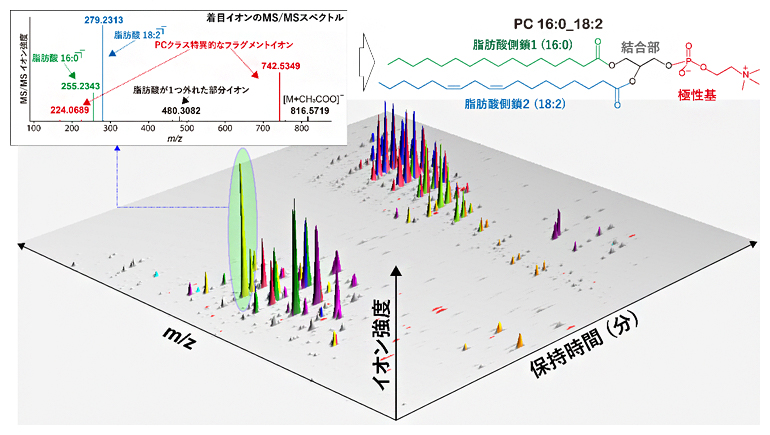
図3 質量分析データの概略と脂質構造とMS/MSスペクトルの関係
実際のLC-MS/MSデータは、脂質分子種の溶出時間(保持時間)と質量情報(m/z)、代謝物の存在量(イオン強度)が含まれる三次元データとなる。そしてピークと呼ばれる山のようなもの一つ一つが、各脂質分子種に対応する。各ピークは、質量分析装置内の衝突室という場所で「マスフラグメンテーション」と呼ばれる手法により断片化され、それがMS/MSスペクトルという形で化合物の断片化構造が観測される(左上図)。これら断片化イオンの情報を読み、脂質代謝物の構造を推定する工程(右上図)を包括的に行うことで、網羅的な脂質分子情報が得られることになる。
共同研究グループはまず、「脂質の分子構造とMS/MSスペクトルの関連性」を定式化し、MS/MSスペクトルから脂質構造を網羅的に推定するためのアルゴリズム開発に着手しました。つまり、各脂質クラスに特有の「マスフラグメンテーションパターン」を読み解き、どのタイプの脂肪酸が含まれていても成り立つフラグメンテーション機構をプログラミング技術で表現しました。最終的には、現在定義されている脂質クラスの全301のうち、未報告の新しい構造を含む合計117脂質クラスを包括的に捉えるためのアルゴリズムを構築しました。この数は、世界で公開されているどのプログラムよりも広い分子種をカバーしており、従来に比べて約2倍の脂質クラスを捉えることが可能です。
また、化合物構造を決定する基準として重要なパラメーターに、LC-MS/MSおよびIM-MS/MSにおける化合物の「溶出時間」があります。溶出時間とは、分析装置内で生体分子が「分離部」に入り、そこから溶出するまでの時間を表します。特に、同じ質量を持つ分子を分離するためのイオンモビリティによる化合物の溶出時間は、化合物自身の物理化学的性質である衝突断面積(CCS)[7]に依存するため、全ての研究機関で共有できる普遍的な物性であり、MS/MSスペクトルだけでは決定できない「異性体」を識別するためにも有用な化学情報です。この溶出時間を機械学習により予測することを試みました。
具体的には、全部で3,000種以上の化合物構造と溶出時間の情報をさまざまな機械学習器(ディープラーニング[8]や勾配ブースティング[9]など)に学習させ、最も高い予測[10]を出力する学習器を選定しました。その結果、勾配ブースティングに基づくXGBoost[9]を用いることとし、117脂質クラスに含まれる約60万種の脂質構造(生物では未発見の推定構造を含む)の溶出時間の予測値を、誤差2.5%前後の予測精度で出力しました。そして、脂質分子の構造推定テストを行ったところ、構築したMS/MSスペクトルのパターン認識、化合物の液体クロマトグラフィーにおける保持時間、イオンモビリティにおけるCCSの情報全てを統合することで、分析データから正しい脂質分子構造を99%前後の精度(FDR[11]が1.5%未満)で出力できることが分かりました。
構築したアルゴリズムを、津川裕司研究員がこれまで開発してきた質量分析データ統合解析プログラムである「MS-DIAL」注1-3)に実装し、「MS-DIAL 4」として、世界に公開しました。MS-DIAL 4を理研メタボローム研究チームで過去5年間に計測してきたさまざまな細胞、マウスおよびヒト由来検体の高精度なノンターゲット質量分析計測データに適用しところ、8,051の脂質分子種の構造多様性を捉えることができました(図4)。この結果は、これまで500~1,000種ほどの分子種しか捉えられなかった既存の研究に比べ、およそ10倍の脂質構造の多様性を捉えるものであり、今後のリポクオリティ研究を支える革新的技術だといえます。
さらに、未報告の新しい脂質クラスの構造を六つ推定し、そのうち一つの構造(生体に豊富に存在するスフィンゴミエリンに、新たにアシル基が一つ付加されたもの)に関しては、化合物の全合成を行い、二重結合の位置やシス/トランス異性体の帰属を行いました。
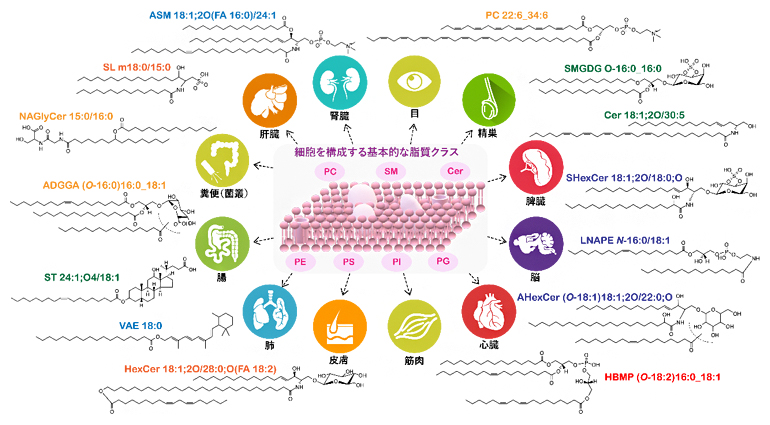
図4 革新的ノンターゲットリピドミクス解析により捉えたリポクオリティの多様性
生体内に豊富に存在し、これまでさまざまな研究が行われてきた基本的な脂質クラス(PCなど)に加え、本研究で捉えた臓器組織特異的な脂質分子種、ならびに本研究により初めて明らかにした脂質クラスの構造(ASM、ADGGA、AHexCerが該当)の概略を示す。
注1)2015年5月5日プレスリリース「生体内の低分子化合物を網羅的に捉える解析プログラムを開発」
注2)2017年11月28日プレスリリース「未開拓の代謝物を次世代メタボローム解析により発見」
注3)2019年3月29日プレスリリース「情報科学で生体内の多様なメタボロームを包括的に解明」
今後の期待
MS-DIAL 4は現時点で、IM-MS/MSの解析プログラムとしては世界初のものであり、捉えられる脂質分子種の数が世界で最も広範囲に、かつ高精度で捉えられるプログラムです。本研究の本質は、117脂質クラスの全8,051脂質分子種が、たった一つの測定手法に基づくノンターゲット分析とMS-DIAL 4による解析により捉えることが可能となったことです。これまで、脂質についてはそれぞれの分子種に測定対象を絞ったターゲット解析[1]による各論的研究が行われてきました。しかし生命は多様な化合物の集合体により構成・維持される複雑なシステムであり、その全体的な理解には本研究のような総論的な研究が欠かせません。
本研究成果は、このような生命システムを脂質分子種から網羅的に捉えることを可能とし、代謝異常が病態の背後に潜む疾患の分子メカニズムの解明に貢献できると考えられます。さらに、ヒトそれぞれが持つリポクオリティを的確に捉えられるため、脂質の多様性の生物学的意義の解明や、脂質分子種の機能発現に関わる遺伝子・タンパク質や標的分子の同定を通じて、ヒトのQOL向上に向けた新たな創薬戦略の確立に貢献すると期待できます。
補足説明
1.ノンターゲットリピドミクス、ノンターゲット解析、ターゲット解析
分析化学では大きく分けて、計測する生体分子の種類を限定する(例えば10種類)「ターゲット解析」と、質量分析に導入された全代謝物を一斉に解析する「ノンターゲット解析」手法が存在する。後者は得られる情報量は多いが、データが複雑になるという難点がある。また、生体中の脂質分子種の総体(リピドーム)を包括的に解析し、複雑な生命システムを解き明かすことを目的とするのが「リピドミクス」という学術分野であり、「ノンターゲットリピドミクス」という言葉は、脂質分子種の総体をノンターゲット解析により捉える手法という意味で使われている。
2.リポクオリティ
Lipid(脂質)とQuality(質)を掛け合わせた言葉である。これは、従来「量」(Quantity)として捉えられることが多かった脂質の「質」(Quality)の違いを見分けることの重要性を表現するため、新学術領域研究「脂質クオリティが解き明かす生命現象」のキーワードとして生み出された言葉である。
3.質量分析法
代謝物を「イオン化」し、そのイオンを検出することにより、原子や分子の「質量」を計測する分析法。そのイオンの存在量から、代謝物の含有量を調べることができる。また質量分析装置内で、代謝物に高エネルギーを付加し、断片化させることで代謝物特異的な「マススペクトル」を取得することで、化学構造の推定も可能である。
4.MS/MSスペクトル
タンデム型質量分析装置では、代謝物由来のイオンそのものを検出するだけでなく、そのイオンを開裂させて生成されるイオン群(断片化イオン、フラグメントイオン)を検出できる。この断片化イオンスペクトルのことをMS/MSスペクトルと呼び、化合物同定では必須の指標として用いられる。
5.液体クロマトグラフィータンデム質量分析(LC-MS/MS)
液体クロマトグラフィーにより分離された化合物をイオン化し、質量分析装置を用いて化合物の検出および定量を行う装置。質量分析装置内でイオンを断片化し(フラグメント化)、その断片化イオン群を測定することを「MS/MSスペクトルを取得する」という。MS/MSスペクトルの情報から、化合物構造を同定・推定することが可能である。
6.イオンモビリティータンデム質量分析(IM-MS/MS)
質量分析の装置内で、同じ質量を持つ分子を分離するための手法を「イオンモビリティ」と呼ぶ。最も有名な方法としては、生体分子イオンをヘリウムなどの不活性ガスが充填されたガス室で飛行させ、「ガス分子と大きく相互作用する分子は遅く、逆にあまり相互作用しない分子は早く溶出する」という原理を用いることで、同じ質量の生体分子を分離できる。また、重要なことは、このような分子の溶出時間は、生体分子と不活性ガスとの衝突断面積(CCS)に依存するため、CCSの理論計算により生体分子の移動度があらかじめ予測可能であり、生体分子の同定に利用できる。また、イオンモビリティにより分離された生体分子は、LC-MS/MSと同様、生体分子特有のMS/MSスペクトルを取得できる。
7.衝突断面積(CCS)
生体分子構造と不活性ガスとの衝突断面積を表す指標であり、よく「分子を回転させたときの三次元表面積」として描写される。CCSの値は、不活性ガスが同じであれば装置ごとに互換性があり、世界の研究者が共有できる物理化学的な値である。そのため、計測で得られた未知の代謝物のCCSとデータベースに登録されている(予測を含む)CCSの情報と照らし合わせることで、未知化合物の構造推定にも応用可能である。CCSはcollision cross sectionの略。
8.ディープラーニング
日本語では深層学習と呼ばれ、人工知能(AI)研究の発展を支える機械学習法の一つ。機械学習研究の目的の一つは、「人間が行う作業をコンピューターができるようにする」ことであるが、今回の研究では「脂質構造の情報から、重要な物理化学的パラメーターである溶出時間をコンピュータに予測してもらう」ことを目的に、本手法を使用した。今回用いた手法は、深層学習に最もよく使われているKerasというフレームワークを用いたディープニューラルネットワークである。ニューラルネットワークとは、人間の神経細胞(ニューロン)の仕組みを模したシステムです。ディープラーニングとは、このニュラルネットワークを多層にして用いることでデータに含まれる特徴を段階的により深く学習することが可能になります。そして、初期入力(脂質構造)と最終出力(溶出時間)の間に潜む最適なニューラルネットワーク階層(入力と出力の細かな組み合わせ)を構築し、高精度の出力(溶出時間)を行うことが可能となる。
9.勾配ブースティング、XGBoost
ディープラーニング同様、機械学習研究で頻用される手法の一つであり、その実装の一つがXGBoostと呼ばれるものである。単独で使うと精度は低いが予測値のばらつきが小さい「弱い学習器」を多数作り、うまく組み合わせることで、ばらつきを小さく保ったまま精度を高める方法(つまり、弱い学習器をまとめることで強い学習器を生成すること)をブースティングと呼ぶ。XGBoostは、「前に構築された弱学習器の結果を考慮し、次の弱学習器を構築する」操作を直列につないでいく学習法である。勾配という言葉は、実装されているブースティングアルゴリズムにおけるパラメーター推定での最適化手法が、勾配降下法という方法論に則っていることに由来する。
10.予測精度
真の値(正解の値)と予測値の差を表す言葉であり、小さければ小さいほど良い指標となる。今回の研究で挑戦した溶出時間(CCS)の予測は、実験誤差として2%の測定誤差を含んでいるため、2.5%という予測精度は現状達成可能な最高予測精度を達成しているといえる。
11.FDR
予測精度を客観的に評価するために頻用される指標。Aと分類されるべきものを正しくAと分類できた和をX、本来Aと分類されるべきでは無いものをAと分類してしまった和をYとしたとき、FDRはX/(X + Y)×100(%)として表される。FDRはfalse discovery rateの略。
国際共同研究グループ
理化学研究所
生命医科学研究センター メタボローム研究チーム
研究員 津川 裕司(つがわ ひろし)
(環境資源科学研究センター メタボローム情報研究チーム 研究員)
チームリーダー 有田 誠(ありた まこと)
(慶應義塾大学薬学部・薬学研究科 教授)
上級研究員(研究当時) 池田 和貴(いけだ かずたか)
(現 かずさDNA研究所ゲノム事業推進部生体分子解析グループ グループリーダー)
大学院生リサーチアソシエート 内野 春希(うちの はるき)
(慶應義塾大学大学院薬学研究科 博士課程D2)
客員研究員 岡橋 伸幸(おかはし のぶゆき)
(大阪大学大学院情報科学研究科 准教授)
環境資源科学研究センター
メタボローム情報研究チーム
テクニカルスタッフI 高橋 みき子(たかはし みきこ)
パートタイマーII 佐藤 綾(さとう あや)
テクニカルスタッフI 山田 豊(やまだ ゆたか)
チームリーダー 有田 正規(ありた まさのり)
(情報・システム研究機構国立遺伝学研究所 教授)
統合メタボロミクス研究グループ
研究員 東 泰弘(ひがし やすひろ)
客員研究員 岡咲 洋三(おかざき ようぞう)
(三重大学生物資源学研究科 准教授)
グループディレクター 斉藤 和季(さいとう かずき)
(環境資源科学研究センター センター長)
ブルカージャパン株式会社 ダルトニクス事業部 アプリケーション部
森 美詞(もり よしふみ)
総合研究大学院大学生命科学研究科遺伝学専攻
博士課程3年多田一風太(ただいっぷうた)
(日本学術振興会特別研究員DC1)
Atens Next Generation Agronomics Laboratory
研究員 パオロ・ボニーニ(Paolo Bonini)
中国科学院 Interdisciplinary Research Center on Biology and Chemistry
博士課程D3 ジベイ・ゾウ(Zhiwei Zhou)
Principal Investigator ジェン・ズ(Zheng-Jiang Zhu)
フロリダ大学 Department of Pathology
研究員 ジェレミー・コーメル(Jeremy Koelmel)
Institute of Physiology of the Czech Academy of Sciences
Department of Translational Metabolism
Associate professorトーマス・チャイカ(Tomas Cajka)
カリフォルニア大学デービス校
West Coast Metabolomics Center
教授 オリバー・フィーン(Oliver Fiehn)
研究支援
本研究は主に、日本学術振興会(JSPS)科学研究費補助金新学術領域研究「脂質クオリティが解き明かす生命現象(領域代表者:有田誠)」、同基盤研究(B)「統合オミクス研究に資する質量分析インフォマティクスによる新規代謝制御機構の解明(研究代表者:津川裕司)」、科学技術振興機構(JST)ライフサイエンスデータベース統合推進事業 統合化推進プログラム「物質循環を考慮したメタボロミクス情報基盤(研究代表者:有田正規)」による支援を受けて行われました。
原論文情報
Hiroshi Tsugawa*, Kazutaka Ikeda, Mikiko Takahashi, Aya Satoh, Yoshifumi Mori, Haruki Uchino, Nobuyuki Okahashi, Yutaka Yamada, Ipputa Tada, Paolo Bonini, Yasuhiro Higashi, Yozo Okazaki, Zhiwei Zhou, Zheng-Jiang Zhu, Jeremy Koelmel, Tomas Cajka, Oliver Fiehn, Kazuki Saito, Masanori Arita & Makoto Arita*, “A lipidome atlas in MS-DIAL 4”, Nature Biotechnology, 10.1038/s41587-020-0531-2
(*corresponding authors)
発表者
理化学研究所
生命医科学研究センター メタボローム研究チーム
研究員 津川 裕司(つがわ ひろし)
(環境資源科学研究センター メタボローム情報研究チーム 研究員)
理化学研究所
生命医科学研究センター メタボローム研究チーム
チームリーダー 有田 誠(ありた まこと)
(慶應義塾大学薬学部・薬学研究科 教授)
報道担当
理化学研究所 広報室 報道担当
慶應義塾広報室(豊田)