2020-12-18 理化学研究所,東京大学大学院理学系研究科
理化学研究所(理研)開拓研究本部新宅マイクロ流体工学理研白眉研究チームの小口祐伴研究員、新宅博文理研白眉研究チームリーダー、東京大学大学院理学系研究科の上村想太郎教授は、「DNAバーコード分子[1]」の配列を1分子単位で識別、空間分布情報を取得する手法を開発しました。
本研究成果は、従来の遺伝子空間分布解析をより高精度にするもので、基礎生物学だけでなく病理解明など医学分野への貢献が期待できます。
これまで、次世代シーケンサー[2]を使った遺伝子発現解析では遺伝子の量(数)を計測するだけでしたが、DNAバーコード分子を活用することで、遺伝子の位置情報を同時に取得できるようになりました。しかし、遺伝子が細胞のどの位置に存在するかを知るには、空間分解能が不十分という難点がありました。
今回、共同研究チームは、全反射蛍光顕微鏡[3]と送液装置を組み合わせることで、DNAポリメラーゼ[4]による「1分子シーケンス反応[5]」を実現する次世代シーケンサーを独自に構築し、DNAバーコード分子の情報(配列および位置)を1分子単位で取得することに成功しました。本手法により、細胞内の遺伝子分布情報を網羅的かつ1分子空間解像度で取得することにつながると考えられます。
本研究は、オンライン科学雑誌『Communications Biology』(12⽉18⽇付︓⽇本 時間12⽉18⽇)に掲載されます。
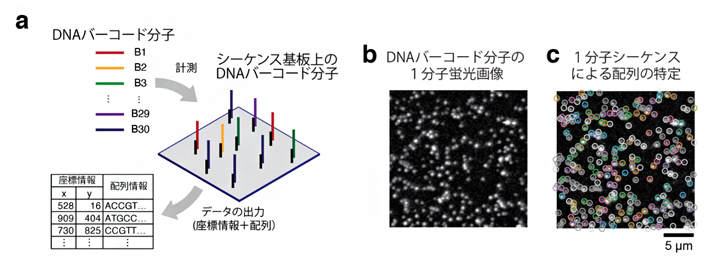
30種類のDNAバーコード分子の空間解析例
背景
私たちの体は、約37兆個の細胞によって構成されています。それぞれの細胞は、全ての遺伝情報が含まれる同一のゲノム配列を持ちますが、この中から特定の遺伝子情報を使うことで、それぞれの細胞に特有の個性(細胞の種類)が生まれると考えられています。近年、次世代シーケンサーの急速な発達により、個々の細胞ごとに遺伝子の種類とその量(数)を調べることが可能になりました。この遺伝子の構成情報は、細胞の正確な分類、新種の細胞の発見などにもつながっています。
また、最近では、単に遺伝子の量を知るだけではなく、遺伝子が細胞のどこにあるかを示す空間分布情報を得ることで、より正確に細胞を分類したり、個々の遺伝子の役割を解明したりすることが試みられています。これまで一般に、次世代シーケンサーを使った遺伝子発現解析は遺伝子の量を計測するだけでしたが、「DNAバーコード分子」を活用することで、遺伝子の位置情報を同時に取得する遺伝子空間分布解析も可能になりました(図1)。
次世代シーケンサーは一度に大量のDNA配列を解析できるため、複数の試料(異なる細胞)を混ぜて測定しますが、それぞれの配列がどの細胞に由来するのかを見分ける必要があります。これを可能にしたのがDNAバーコード分子です。DNAバーコード分子は、10ヌクレオチド(文字)程度の人工的なDNA配列で、それぞれの試料ごとに異なるDNAバーコード分子を付加します。すると、次世代シーケンサーから各々のDNA配列はDNA配列バーコード分子配列を伴い出力されるため、この配列を頼りにどの細胞に由来するのか見分けることができます。このバーコード分子の配列と空間情報を対応させることで、遺伝子空間分布解析が実現します(図1)。
しかし、次世代シーケンサーを活用する既存の遺伝子空間分布解析の空間解像度は、10~100マイクロメートル(μm、1μmは1,000分の1mm)程度です。これはおよそ細胞1個分の大きさに相当することから、特定した遺伝子がどの細胞に属していたかは分かるものの、細胞内での位置までは特定できません。遺伝子は転写後、細胞中の適所に輸送されることで(細胞内局在)機能しますが、このような特徴を捉えることはできません。細胞内での遺伝子位置情報を得るためには、DNAバーコード分子が与える空間解像度を高める必要がありました。
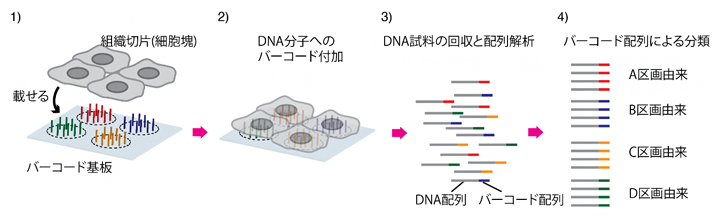
図1 次世代シーケンサーを活用する従来の遺伝子空間分布解析
1)区画ごとに異なるバーコード分子を持つ基板上に、計測試料となる組織切片などを載せる。
2)各々の細胞に含まれる遺伝子から、配列決定用DNA分子を作製する。このとき、配列決定用DNA分子にはバーコード分子が付加される。
3)基板からバーコード分子を付加した配列決定用DNA分子を回収し、次世代シーケンサーによりDNA配列を決定するとともにバーコード配列を取得する。
4)バーコード分子により、どの区画に由来したDNAであったかを識別する。しかし、ここに示す方法でのバーコード分子が識別する区画の大きさ(空間解像度)は、バーコード基板のスポットの大きさで決まり、10~100μm程度である。
研究手法と成果
共同研究チームは、全反射蛍光顕微鏡と送液装置を組み合わせることで、「1分子シーケンス反応」を実現する次世代シーケンサーを独自に構築し、DNAバーコード分子の配列と位置情報を1分子単位で解析することに成功しました。
1分子シーケンス反応の基本原理にはいくつかのタイプがありますが、今回はDNAポリメラーゼによる伸長反応を活用する方式を採用しました。DNAポリメラーゼはDNAの複製を担う非常に重要な酵素です。通常、DNAポリメラーゼは鋳型鎖の配列を認識しつつ、これに対応したヌクレオチド(DNAを構成する4文字:ATGC)をつなぎ合わせ、鋳型鎖配列を合成していきます。このヌクレオチドを蛍光色素で標識すると、ポリメラーゼが鋳型鎖を伸長する様子を全反射蛍光顕微鏡で観察できるようになります。しかし、同時に大量のDNA分子配列を正確に決定するためには、各々の分子で起こる伸長反応の制御が必要で、これは特殊な蛍光ヌクレオチドを活用することで実現します(図2)。
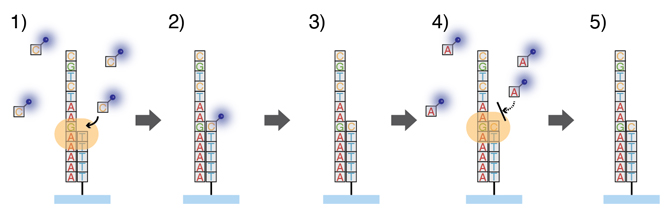
図2 DNAポリメラーゼによる1分子シーケンス反応
本研究で活用した1分子シーケンス反応。通常のDNAポリメラーゼが伸長反応の基質として用いる天然のヌクレオチド(ATGC)とは異なり、蛍光色素と反応制御部位を持つ特殊な蛍光ヌクレオチドを用いる。また、一度の伸長反応では1種類の特殊ヌクレオチドを入れる。
1)特殊蛍光ヌクレオチドの反応制御部位により、一度の伸長反応によってDNAポリメラーゼは一つのヌクレオチドのみを結合させる(1文字分)。
2)余剰の特殊蛍光ヌクレオチドを洗い流した後、蛍光顕微鏡で観察する。
3)特殊蛍光ヌクレオチド反応制御部位を切り離すと、また1)-2)と同じ反応を繰り返すことが可能になる。
4)5)対応する塩基でない場合は、ヌクレオチド取り込みは起こらない。これら一連の反応サイクルを繰り返し、取り込まれた特殊ヌクレオチドを逐次観察することで、DNAバーコード分子の配列を1分子単位に決定できる。
まず、この独自のシーケンサーを用い、配列が分かっている30種類のDNAバーコード分子の識別を試みました(図3a)。このDNAバーコード分子を鋳型配列として、特殊蛍光ヌクレオチドのDNAポリメラーゼによる取り込み反応を可視化し、DNAバーコード分子の位置(観察視野における座標情報)を1分子単位で特定しました(図3b)。さらに、この1分子シーケンス反応を繰り返し実施することで、それぞれの分子配列を特定し、期待通り30種類のバーコード分子を検出することに成功しました(図3c, d)。
また、これらのバーコード分子はシーケンス基板上に、既存技術のようにあらかじめ区画と配列を対応づける必要はなく、かつ、パターン化して並べるというような煩雑な操作も必要としません。
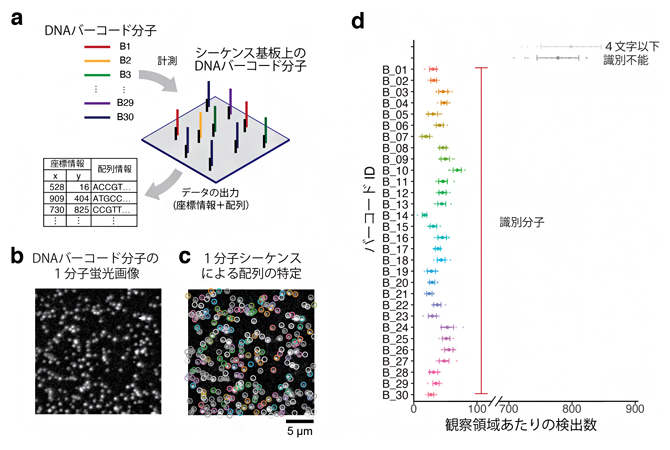
図3 30種類のDNAバーコード分子の空間解析例
a)DNAバーコード1分子空間解析の概略図。配列既知の30種類のDNAバーコード分子を用意し、独自に開発したシーケンサーに試料を投入する。シーケンサーにて、1分子単位に配列を解読する。ガラス基板上での位置情報を取得する。本研究で開発したDNA配列の決定精度は1文字あたり95%である。各々のDNA配列がどのバーコードに対応するか、配列探索アルゴリズムを用いて解析する。
b)特殊蛍光ヌクレオチドの取り込みにより可視化されたDNAバーコード分子の蛍光画像。黒い背景に対し、白の点状のものが1分子単位のDNAバーコードである。
c)1分子シーケンス反応による配列の特定。b)の画像において、30種類のうちどのDNAバーコード分子か、識別できたものを丸で囲っている。色はDNAバーコード分子の種類を示し、その種類(バーコードID)と色の対応関係はd)に示している。
d)観察視野あたりの検出DNAバーコード分子の数。30種類のDNAバーコード分子を漏れなく検出していることが分かる。ただし、1分子シーケンス反応が途中で停止してしまう分子もある。4文字以下で停止したものは解析から除外した。
さらに、DNAバーコード分子として、抗体に結合したバーコード分子の特定も可能であることを示しました。これまでタンパク質の検出には、蛍光標識された抗体が用いられてきました。抗体の種類(タンパク質の種類)と蛍光色素の色を対応させることで、一度に複数のタンパク質を可視化できます。しかし、蛍光顕微鏡で活用できる色の数には限界があり、現状、4~5種類程度にとどまっています。この蛍光色素をDNAバーコード分子に置き換えることで、一度に同時に検出できるタンパク質の種類が飛躍的に向上することが期待できます。このように本手法は、遺伝子の検出だけではなく、DNAバーコード分子標識抗体を活用したタンパク質空間分布解析の実現にもつながると考えられます。
今後の期待
次世代シーケンサーとDNAバーコード分子による従来の遺伝子空間分布解析は、筋萎縮性側索硬化症(ALS)の脳組織切片やがん組織切片の解析に活用されています。本研究で開発した1分子空間解像度を持つDNA分子バーコード識別技術は、この遺伝子空間分布解析をより高精度にすることから、このような病理解明などの医学分野への貢献はもとより、遺伝子の局在性と機能の関連などの理解を深める基礎生物学への貢献も期待できます。
補足説明
1.DNAバーコード分子
それぞれ異なる配列を持つDNA分子(DNAバーコード分子)を、目的の核酸分子(DNAやRNA)の一つ一つに付加することで、核酸分子の数をデジタル的に正確に計測できる。
2.次世代シークエンサー
サンガー法を利用した蛍光キャピラリーシークエンサーである「第一世代シークエンサー」と対比させて使われる用語。機種によるが、一度に1,000万~100億個のDNAの配列を決定できる。以前は、ヒトのゲノム配列を決定するのに14年程度かかったが、次世代シークエンサーを用いると数日で決定できる。
3.全反射蛍光顕微鏡
励起光を当てることで、蛍光色素は蛍光を発する。この励起光を、励起光の観察試料を載せるカバーガラスとの境界面で全反射するように入射すると、この境界面から数百ナノメートルが照明される。したがって、この厚みの範囲でのみ励起光が当たった試料からのみ蛍光が発生する。このため、背景に光ノイズの少ない非常に暗い状態で観察することで、蛍光分子1個を観察することも可能となる。
4.DNAポリメラーゼ
細胞分裂の際、染色体を複製するという生命過程に非常に大事な役割を担う酵素の一つ。一本鎖DNAを鋳型として、相補的なDNA配列を合成する酵素。
5.1分子シーケンス反応
DNAポリメラーゼのDNAの複製を活用し、DNAの配列を1分子のDNA単位で決定する方法。ただし、通常の複製反応で活用する天然のヌクレオチドではなく、特殊蛍光ヌクレオチドを代用することで、DNAポリメラーゼによる取り込み反応を1分子単位に可視化し、塩基配列を特定する。
研究支援
本研究は、内閣府革新的研究開発推進プログラム(ImPACT)「セレンディピティの計画的創出による新価値創造(プログラムマネージャー:合田圭介)」、日本学術振興会(JSPS)科学研究費補助金基盤研究(C)「1細胞非増幅シーケンスを可能にするライブラリ調製法の開発(研究代表者:小口祐伴)」、科学技術振興機構(JST)戦略的創造研究推進事業 個人型研究(さきがけ):「多細胞システムにおける細胞間相互作用とそのダイナミクス研究領域(研究総括:高橋淑子)」における研究課題「細胞間相互作用の理解に資するゲノムワイド1分子遺伝子空間分布解析(研究者:小口祐伴)」の一環として行われました。
原論文情報
Yusuke Oguchi, Hirofumi Shintaku, Sotaro Uemura, “Development of a sequencing system for spatial decoding of DNA barcode molecules at single-molecule resolution.”, Communications Biology, 10.1038/s42003-020-01499-8
発表者
理化学研究所
開拓研究本部 新宅マイクロ流体工学理研白眉研究チーム
研究員 小口 祐伴(おぐち ゆうすけ)
理研白眉研究チームリーダー 新宅 博文(しんたく ひろふみ)
東京大学大学院 理学系研究科 生物科学専攻
教授 上村 想太郎(うえむら そうたろう)
報道担当
理化学研究所 広報室 報道担当