2021-02-18 理化学研究所
理化学研究所(理研)計算科学研究センター粒子系生物物理研究チームの杉田有治チームリーダー、小林千草技師、開拓研究本部杉田理論分子科学研究室の森貴治専任研究員、ジョン・ジェウン専任技師らの研究チームは、スーパーコンピュータ「富岳」[1]と「Oakforest-PACS」[2]を用いて新型コロナウイルスSARS-CoV-2の表面に存在する「スパイクタンパク質」のシミュレーションを行い、ウイルスがヒト細胞に侵入する際に起こるスパイクタンパク質の構造変化において、スパイクタンパク質表面を修飾している糖鎖[3]が重要な役割を果たしていることを発見しました。
本研究成果は、新型コロナウイルス感染症 COVID-19に対する感染予防や治療に向けた医薬品の分子設計に貢献すると期待できます。
新型コロナウイルスのスパイクタンパク質の受容体結合ドメイン(RBD)には、「ダウン型構造」と「アップ型構造」が存在し、RBDがヒト細胞表面のACE2受容体[4]に結合して感染する際はアップ型構造をとっていることが知られています。
今回、研究チームは「富岳」と「Oakforest-PACS」を用いて、スパイクタンパク質の分子動力学シミュレーション[5]を行いました。その結果、ダウン型構造とアップ型構造の両方において、スパイクタンパク質の表面を修飾している糖鎖が”補強役”となってRBDを安定化していることを発見し、RBD間の静電的な反発が駆動力となり、アップ型への構造変化が誘起されるという分子メカニズムを提案しました。
本研究は、科学雑誌『Biophysical Journal』オンライン版(2月13日付)に掲載されました。
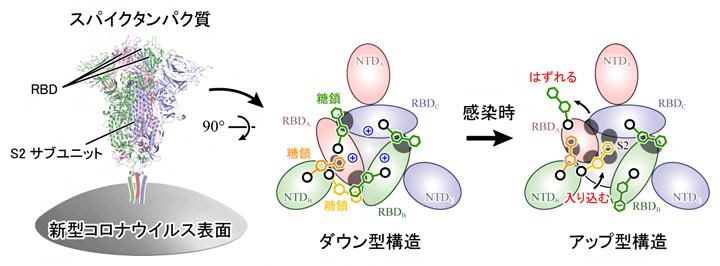
新型コロナウイルス表面に存在するスパイクタンパク質の構造変化機構
背景
2019年に発生した新型コロナウイルスSARS-CoV-2は、新型コロナウイルス感染症 COVID-19として「パンデミック」と呼ばれる世界的大流行を引き起こし、2021年2月現在でも感染拡大が収束しないことから、感染予防および治療に対する有効な医薬品の早期開発が望まれています。医薬品の効率的な開発には、ウイルス感染の詳細なメカニズムを知る必要があり、感染に関与するタンパク質に対してさまざまな解析が世界的に進められています。
新型コロナウイルスがヒト細胞に侵入する初期段階では、ウイルスの表面に存在するタンパク質(スパイクタンパク質)がヒト細胞表面のアンジオテンシン変換酵素II(ACE2受容体)に結合して吸着し、ウイルスが侵入することで感染に至ります(図1A)。スパイクタンパク質は3本のポリペプチド鎖[6]から構成され、各ポリペプチド鎖はN端ドメイン(NTD)、受容体結合ドメイン(RBD)、S2ドメインから構成されます(図1B左)。生化学実験により、スパイクタンパク質表面の多くのアミノ酸が、糖鎖によって修飾されていることも分かってきました。スパイクタンパク質に限らず、多くのタンパク質の表面は翻訳後修飾[7]によって糖鎖が付加されており、タンパク質がお互いを認識する際にその糖鎖が用いられていると考えられています。また、ウイルスの場合は、抗体による攻撃から逃れるためにスパイクタンパク質が糖鎖で覆われているともいわれています。
X線結晶構造解析[8]やクライオ電子顕微鏡[9]を用いた単粒子解析[10]により、新型コロナウイルスのスパイクタンパク質は「ダウン型」および「アップ型」の構造をとることが分かってきました(図1B)。アップ型構造では、RBDがS2サブユニットから離れるように移動します(図1B赤色部分)。スパイクタンパク質がACE2受容体に結合していないときは、ダウン型とアップ型を行き来しており(平衡状態)、ACE2受容体に結合するとアップ型がより安定になることも分かってきました。
このような構造解析実験によって、スパイクタンパク質の立体構造に対する理解が大きく進んできましたが、一方で、揺らぎの大きい糖鎖については詳細な分子構造が分からず、実際、新型コロナウイルスがヒト細胞に侵入する過程において、糖鎖がどのような役割を果たしているのかは分かっていませんでした。
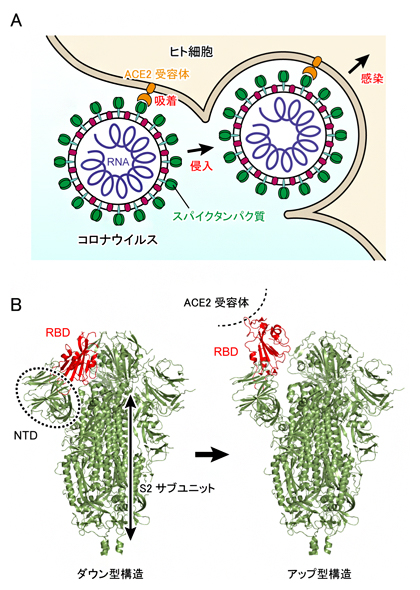
図1 新型コロナウイルスSARS-CoV-2の感染メカニズム
(a)ウイルスがヒト細胞に感染する初期段階において、ウイルス表面にあるスパイクタンパク質(緑色)がACE2受容体(橙色)に結合する。その後、ヒト細胞の膜と融合を経て、ウイルス内のRNA(紫色)がヒト細胞内に取り込まれる。
(b)クライオ電子顕微鏡を用いた単粒子解析により明らかになったスパイクタンパク質の立体構造。左はダウン型構造、右はアップ型構造で、アップ型構造はACE2受容体に結合したときにより安定化される。結合部位RBDは赤色で示されており、ダウン型からアップ型構造になるときに、S2サブユニットから離れるように上方に移動する。NTDとS2サブユニットはRBDに近接しているが、大きな構造変化は起こさない。
研究手法と成果
研究チームは、スパイクタンパク質の詳細な分子構造を調べるために、スーパーコンピュータ「富岳」と「Oakforest-PACS」を用いて、ダウン型構造とアップ型構造に対する分子動力学シミュレーションを行いました(図2A)。分子動力学シミュレーションとは、コンピュータの中に仮想的に分子システムを構築し、原子1個1個に対してニュートンの運動方程式 F=maを解くことで、安定な分子構造を理論的に予測したり、分子の動きを可視化したりする方法です(図2B)。分子システムが巨大な場合、演算量が膨大となるため、スーパーコンピュータを用いた高速な計算が必要となります。
研究チームは、2015年に分子動力学計算ソフトウェアGENESISを独自に開発しました注1,2)。GENESISは「富岳」コデザイン開発のターゲットアプリの一つに選ばれ、ジョン・ジェウン専任技師および小林千草技師らが中心となって「富岳」に最適化し、「京」の100倍以上のアプリケーション実行性能を達成しました。今回は、GENESISを用いて、スパイクタンパク質の1マイクロ秒(100万分の1秒)の分子動力学計算を行い、RBDの構造変化前後におけるアミノ酸-アミノ酸および糖鎖-アミノ酸間相互作用を網羅的に解析しました。
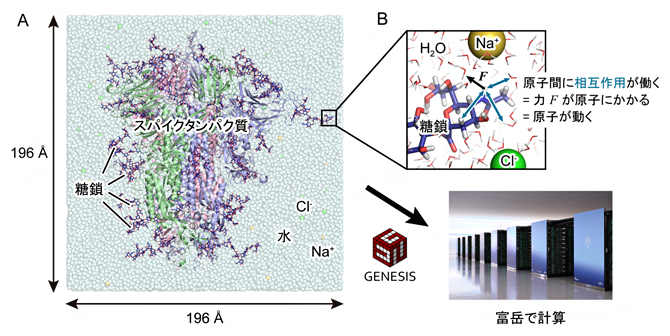
図2 GENESISと「富岳」を用いたSARS-CoV-2スパイクタンパク質のシミュレーション
(a)コンピュータの中に仮想的に構築した水溶液中に存在するダウン型構造のスパイクタンパク質。系の原子数は約76万個で、水分子(H2O)を約23万個含む。タンパク質をリボンモデル、水分子とイオンを球モデル(水色:水分子、黄色:ナトリウムイオン Na+、緑色:塩化物イオン Cl–)、糖鎖を青色のスティックモデルで表示している。本研究で用いたシステムでは、糖鎖が合計で60カ所アミノ酸を修飾している。システムの大きさは、一辺が約196オングストローム(Å、1Åは100億分の1メートル)である。
(b)原子1個1個に対してニュートンの運動方程式 F=maを適用し、数フェムト秒(1フェムト秒は1000兆分の1秒)の時間刻みで原子を動かす。注目する原子と周囲の原子との間の相互作用(図中青色矢印)を計算し、相互作用エネルギーの原子位置に関する1階微分から原子にかかる力 Fを計算する。mには原子の質量を用いる。本研究では、1マイクロ秒(100万分の1秒)の分子運動を追跡するために、2.5フェムト秒の時間刻みで4億ステップの計算を行った。
その結果、スパイクタンパク質の165番目と234番目、343番目の三つのアスパラギン[11]N165、N234、N343を修飾する各糖鎖が、RBDの構造安定化に重要な役割を果たしていることを発見しました。ダウン型ではN343とN165の糖鎖がRBDを上から覆うようにそれぞれRBD-RBD間(図3A上)とRBD-NTD間をつないでいます(図3B上)。アップ型に変化する際は、N343の糖鎖がはずれ(図3A下)、次いでN234の糖鎖がアップ型に変化した際に生じるRBD-S2ドメイン間の空洞(図3C)に入り込み、上部に移動したRBDを下から補強するようにS2 ドメインやRBDと強い水素結合を形成することが分かりました(図3B下)。
さらに、ダウン型構造に対して静電ポテンシャル[12]を解析したところ、三つのRBDの境界面は378番目のリシン(K378、図3A中央青色)や407番目のアルギニン(R407)などによって広い範囲にわたり、正に帯電していることが分かりました。
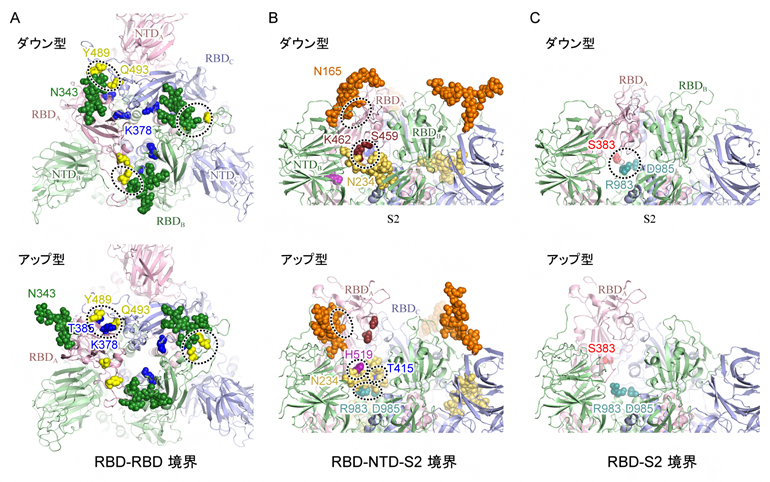
図3 スパイクタンパク質中のアミノ酸-アミノ酸およびアミノ酸-糖鎖相互作用
上段はダウン型構造、下段はアップ型構造を表し、Aはスパイクタンパク質を上から眺めた図、BとCは側面から上部を見た図。主要な相互作用に関与するアミノ酸および糖鎖を球モデルで示し、シミュレーション中で観察された強い相互作用を点線で囲っている。AはRBD-RBD境界、BはRBD-NTD-S2サブユニット境界、CはRBD-S2サブユニット境界に焦点を絞っている。
これらの結果から、アップ型への構造変化は、RBD間の静電的な反発が駆動力となって起こること、さらにダウン型構造を安定化する糖鎖がはずれ、RBDが移動した後に別の糖鎖が入り込むことが示唆されました(図4)。本研究により、研究チームは新型コロナウイルス感染における詳細な分子メカニズムを解明しただけでなく、糖鎖によるタンパク質の動的構造の安定化という新しい生物学的知見を得ることに成功しました。
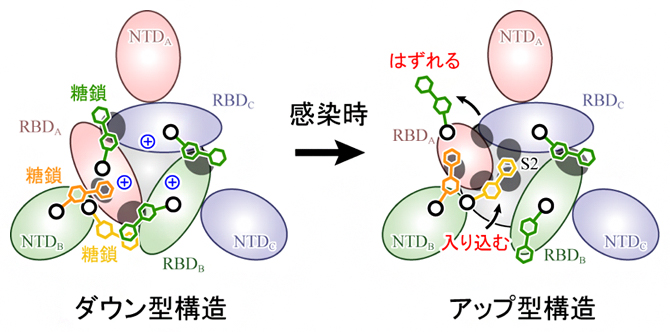
図4 スパイクタンパク質の構造変化のメカニズム
スパイクタンパク質を上から見た図。ダウン型からアップ型への構造変化は、RBD間の正電荷(中央青色+印)による静電的な反発が駆動力となって起こり、ダウン型構造を安定化する糖鎖(緑色)がはずれ、RBDが移動した後に別の糖鎖(黄色)が入り込み、アップ型を安定化する。
注1)2015年5月8日プレスリリース「超並列分子動力学計算ソフトウェア「GENESIS」を開発」
今後の期待
パンデミックを早期に収束させるためには、感染予防および治療のための医薬品開発が急務です。スパイクタンパク質とACE2受容体との結合を阻害する中和抗体[13]医薬品の開発では、ウイルスに結合した不完全な抗体を介した「抗体依存性感染増強」[14]のリスクもあり、抗体や薬剤の分子設計戦略において、「ダウン型を安定化させる」あるいは「アップ型への変化を阻害する」といった、スパイクタンパク質の構造変化を考慮した多様な設計が有用と考えます。今回、研究チームの発見により、スパイクタンパク質の構造変化で補強役となる糖鎖に着目し、例えば「ダウン型のRBD-RBD間の糖鎖を外さないようにする」、あるいは「アップ型になる際に入り込む糖鎖を妨害する」といった新たな戦略の創出が期待できます。
ウイルスには変異しやすい性質、すなわち、一部のアミノ酸が別のアミノ酸に置き換わり、これまでとは異なる感染力を獲得しやすいという特徴があります。2020年12月にイギリスで発見された変異種には17カ所の変異があり、感染力が従来よりも高く、さらなる社会問題となっています。これまでに世界各国でスパイクタンパク質に対する分子動力学シミュレーションが行われ、「富岳」を用いた本研究のトラジェクトリーデータ[15]もウェブサイトで公開されています注3)。シミュレーションで得られたタンパク質の詳細な構造情報は医薬品開発のためのヒントになります。「富岳」や「Oakforest-PACS」を利用することで高速な解析が可能であるため、突如現れる変異種などの問題に対しても、スーパーコンピュータを用いて迅速に対応することで、計算科学技術が社会に大きく貢献すると期待できます。
注3)新型コロナウイルスのシミュレーション研究に関するMolSSIのホームページ(英語)
補足説明
1.スーパーコンピュータ「富岳」
「京」の後継機。社会的・科学的課題の解決で日本の成長に貢献し、世界をリードする成果を生み出すことを目的とし、電力性能、計算性能、ユーザーの利便性・使い勝手の良さ、画期的な成果創出、ビッグデータやAI(人工知能)の加速機能の総合力において世界最高レベルのスーパーコンピュータ。
15万8976個の中央演算装置(CPU)を搭載し、1秒間に約44京2010兆回の計算が可能。2020年6月と11月に世界のスパコンランキング「TOP500」「HPCG」「HPL-AI」「Graph500」で2期連続の世界一位を獲得した。
2.スーパーコンピュータ「Oakforest-PACS」
東京大学情報基盤センターと筑波大学計算科学研究センターが共同運営する、最先端共同HPC基盤施設(JCAHPC: Joint Center for Advanced High Performance Computing)の共同利用スーパーコンピュータシステム。インテルXeon PhiプロセッサとインテルOmni-Pathアーキテクチャを搭載した、国内最大規模の超並列クラスタ型スーパーコンピュータである。
3.糖鎖
グルコース、ガラクトースなどの単糖がグリコシド結合を介して長く連なった化合物。多くのタンパク質の表面は、小胞体やゴルジ体内で酵素の働きにより糖鎖が付加される。糖鎖の修飾を受けたタンパク質は、糖タンパク質と呼ばれ、糖鎖はタンパク質の安定性やウイルスの認識などに重要な役割を果たす。
4.ACE2受容体(アンジオテンシン変換酵素II)
ヒトの細胞膜に存在する膜タンパク質の一つで、心臓、肺、腎臓などの臓器や、舌などの口腔内粘膜に発現している。ACE2は本来、血圧を調整する役割を担っており、生理活性ペプチドホルモンであるアンジオテンシンIIと結合してアンジオテンシン(1-7)を生成する酵素であるが、コロナウイルスのスパイクタンパク質と結合してウイルス感染の入り口にもなってしまう。
5.分子動力学シミュレーション
コンピュータを用いた分子シミュレーション法の一つ。原子間相互作用をフックの法則やクーロンの法則などから計算し、分子系の運動をニュートン方程式 F=maに基づいて数値的に解くことで、分子の動きを理論予測し解析する方法。
6.ポリペプチド鎖
アミノ酸がペプチド結合を介して長く連なった生体高分子化合物。天然には20種類のアミノ酸が存在し、それぞれ異なる化学的性質を持っている。例えば、セリン、スレオニン、アスパラギンは親水性、バリン、イソロイシンは疎水性、アスパラギン酸、グルタミン酸は負電荷、リシン、アルギニンは正電荷を持っている。このようなアミノ酸が連なることで、特定の立体構造を形成する。特に細胞内で機能を発現するポリペプチドはタンパク質と呼ばれる。
7.翻訳後修飾
リボソームによりタンパク質が合成(遺伝情報が翻訳)された後、小胞体やゴルジ体内で別の酵素によって、さらに糖鎖やアセチル基、リン酸基などが特定のアミノ酸に付加されること。
8.X線結晶構造解析
タンパク質の結晶を作製し、その結晶にX線を照射して得られる回折データを解析することにより、タンパク質の内部の原子の立体的な配置を調べる方法。この方法によって、タンパク質の立体構造や内部構造を知ることができる。
9.クライオ電子顕微鏡
タンパク質を含む溶液を極低温(液体窒素温度)にまで急速に冷却し、試料を観察する透過型電子顕微鏡。近年、試料調製法の改良や、電子直接検出器の開発、解析ソフトの進歩により、近原子分解能の性能が得られるようになった。2017年、タンパク質立体構造解析への応用に貢献したとして、クライオ電子顕微鏡を開発したジャック・デュボシェ、ヨアヒム・フランク、リチャード・ヘンダーソンの3氏にノーベル化学賞が授与されている。
10.単粒子解析
クライオ電子顕微鏡によって観察された溶液中にランダムに配向したタンパク質の多数の投影像から立体像を再構築する手法。
11.アスパラギン
アミノ酸の一つで、化学式はC4H8N2O3で表され、一文字表記でNと略される。糖鎖の翻訳後修飾を受ける場合、アスパラギン側鎖の窒素原子に糖鎖が付加される(N-グリコシル化)。
12.静電ポテンシャル
静電場の中の任意の点において、+1クーロンの電荷が持つ位置エネルギー。タンパク質を構成する原子の点電荷によって作られる静電場から分子表面の静電ポテンシャルを解析することで、分子の形状と静電的相互作用に基づいたタンパク質の構造安定性や構造変化を理解できる。
13.中和抗体
ウイルスの受容体結合部位を認識し、結合することで感染を阻害(中和)する抗体。コロナウイルスの場合、中和抗体がRBDに結合することでACE2受容体との結合を阻害し、感染を防止する。
14.抗体依存性感染増強
過去の感染やワクチンの接種などによって獲得された不完全な抗体(中和能力はないが吸着力のある抗体)がウイルスに結合すると、免疫細胞への吸着および侵入が促進されて、ウイルスが分解されずに増殖が引き起こされる現象。
15.トラジェクトリーデータ
分子動力学シミュレーションによって得られる原子の座標データのこと。トラジェクトリーは「軌跡」を意味する。シミュレーション中に一定の時間間隔で原子のXYZ座標の値をファイルに出力し、専用のソフトウェアで読み込むことで、分子が動く様子を可視化できる。
研究チーム
理化学研究所
計算科学研究センター 粒子系生物物理研究チーム
チームリーダー 杉田 有治(すぎた ゆうじ)
(開拓研究本部 杉田理論分子科学研究室 主任研究員)
(生命機能科学研究センター 分子機能シミュレーション研究チーム チームリーダー)
技師 小林 千草(こばやし ちぐさ)
(現 運用技術部門 利用環境技術ユニット 技師)
開拓研究本部 杉田理論分子科学研究室
専任研究員 森 貴治(もり たかはる)
専任技師 ジョン・ジェウン(Jaewoon Jung)
特別研究員 ドカイニッシュ・ヒシャム(Hisham Dokainish)
生命機能科学研究センター 分子機能シミュレーション研究チーム
上級研究員(研究当時) 李 秀栄(り すよん)
(現 国立研究開発法人医薬基盤・健康・栄養研究所)
研究支援
本研究は、「富岳」の優先的な試行的利用課題「「富岳」を用いた新型コロナウイルス表面のタンパク質動的構造予測(課題代表者:杉田有治)」および新型コロナウイルス感染症対応HPCI臨時公募課題「新型コロナウィルス表面のタンパク質動的構造予測(課題代表者:杉田有治)(課題番号:hp200153)」を通じて、スーパーコンピュータ「富岳」および最先端共同HPC基盤施設(JCAHPC)が提供するスーパーコンピュータ「Oakforest-PACS」の計算資源の提供を受け、実施しました。
原論文情報
Takaharu Mori, Jaewoon Jung, Chigusa Kobayashi, Hisham M. Dokainish, Suyong Re, and Yuji Sugita, “Elucidation of interactions regulating conformational stability and dynamics of SARS-CoV-2 S-protein”, Biophysical Journal, 10.1016/j.bpj.2021.01.012
発表者
理化学研究所
計算科学研究センター 粒子系生物物理研究チーム
チームリーダー 杉田 有治(すぎた ゆうじ)
開拓研究本部 杉田理論分子科学研究室
専任研究員 森 貴治(もり たかはる)
報道担当
理化学研究所 広報室 報道担当