2023-10-02 アメリカ合衆国・コーネル大学
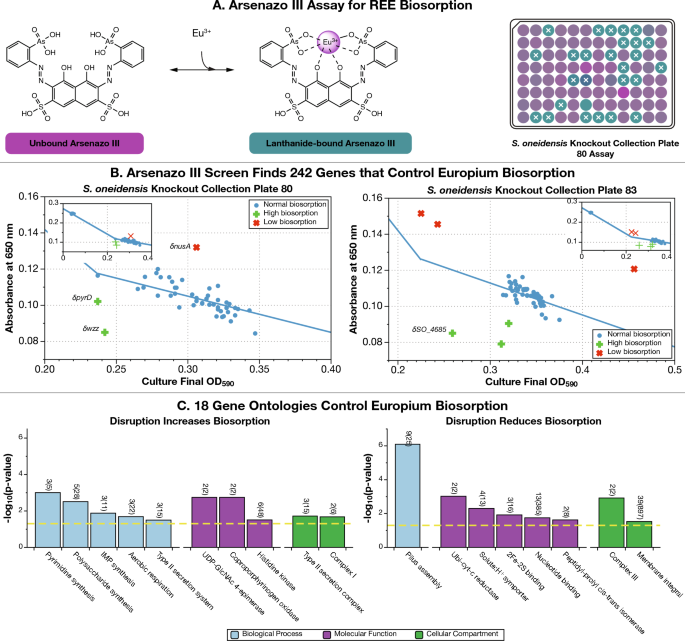
- コーネル大学が、微生物の Shewanella oneidensis (シュワネラ・オネイデンシス)による希土類元素(REE)のバイオ吸着に関与するゲノムを解析。
- 現行の化学的 REE 精製方法の問題は、高コストで環境に有害な有機溶剤や強力な化学物質の使用に大きく依存すること。本研究の成果は、微生物を利用して REE を選択的に吸着・精製する環境に優しい代替手法を提供する。
- S.oneidensis は、ランタノイドとして知られる周期表の 6 段目にある F ブロック元素のユウロピウムを特に好む。同微生物のゲノムの 3,373 の変異体のスクリーニングにより、このことに関与している 42 遺伝子を特定した。これらを操作して他の REE を好むように調整することができる。
- 同微生物で発見された突然変異遺伝子は、S.oneidensis の天然の変種に比べ、REE 精製プロセス時間を従来の 1/3 に短縮し、グリーンな精製手法向上のロードマップを提供する。
- REE のバイオ吸着において、従来より細胞接着やバイオフィルム形成に関わる膜組成を制御する主要な遺伝子を特定し、S.oneidensis の REE 吸着を司るメカニズムを促進する。
- 現在、REE の精製処理は、精製工場を建設する際の厳格な環境規制と高額なインフラコストのため現在外国で実施されている。
- 微生物による REE バイオ吸着プロセスでは、環境に有害な溶剤や土地、多大な資本を不要とし、カラムの中の微生物に栄養分を繰り返し与えることで REE を分離できる。技術や防衛のアプリケーションに向けた米国における REE の安定供給を支援するもの。
- バイオ吸着技術はまだ開発中であるが、2028 年までのパイロット精製システムの構築を目指す。
- 本研究は、Cornell Atkinson、Burroughs Welcome Fund、米国エネルギー省(DOE) エネルギー高等研究計画局 (ARPA-E)および Mary Fernando Conrad ’83 and Tony Conrad が資金を提供した。
- URL: https://news.cornell.edu/stories/2023/10/metal-loving-microbes-savor-green-way-refine-rare-earth
関連情報
Scientific Reports 掲載論文(フルテキスト)
Genomic characterization of rare earth binding by Shewanella oneidensis
URL: https://www.nature.com/articles/s41598-023-42742-6
Abstract
Rare earth elements (REE) are essential ingredients of sustainable energy technologies, but separation of individual REE is one of the hardest problems in chemistry today. Biosorption, where molecules adsorb to the surface of biological materials, offers a sustainable alternative to environmentally harmful solvent extractions currently used for separation of rare earth elements (REE). The REE-biosorption capability of some microorganisms allows for REE separations that, under specialized conditions, are already competitive with solvent extractions, suggesting that genetic engineering could allow it to leapfrog existing technologies. To identify targets for genomic improvement we screened 3,373 mutants from the whole genome knockout collection of the known REE-biosorbing microorganism Shewanella oneidensis MR-1. We found 130 genes that increased biosorption of the middle REE europium, and 112 that reduced it. We verified biosorption changes from the screen for a mixed solution of three REE (La, Eu, Yb) using Inductively Coupled Plasma Mass Spectrometry (ICP-MS) in solution conditions with a range of ionic strengths and REE concentrations. We identified 18 gene ontologies and 13 gene operons that make up key systems that affect biosorption. We found, among other things, that disruptions of a key regulatory component of the arc system (hptA), which regulates cellular response to anoxic environments and polysaccharide biosynthesis related genes (wbpQ, wbnJ, SO_3183) consistently increase biosorption across all our solution conditions. Our largest total biosorption change comes from our SO_4685, a capsular polysaccharide (CPS) synthesis gene, disruption of which results in an up to 79% increase in biosorption; and nusA, a transcriptional termination/anti-termination protein, disruption of which results in an up to 35% decrease in biosorption. Knockouts of glnA, pyrD, and SO_3183 produce small but significant increases (≈ 1%) in relative biosorption affinity for ytterbium over lanthanum in multiple solution conditions tested, while many other genes we explored have more complex binding affinity changes. Modeling suggests that while these changes to lanthanide biosorption selectivity are small, they could already reduce the length of repeated enrichment process by up to 27%. This broad exploratory study begins to elucidate how genetics affect REE-biosorption by S. oneidensis, suggests new areas of investigation for better mechanistic understanding of the membrane chemistry involved in REE binding, and offer potential targets for improving biosorption and separation of REE by genetic engineering.

