2024-07-10 東京大学
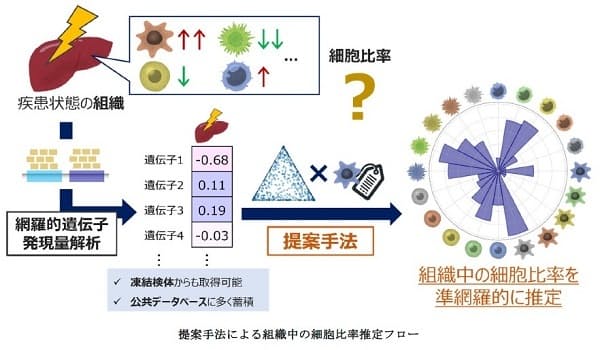
東京大学大学院薬学系研究科の東一織大学院生、水野忠快助教らによる研究グループは、組織の網羅的遺伝子発現量(トランスクリプトーム)データから当該組織中の細胞種の比率を準網羅的に推定するアルゴリズムを開発しました。
組織中の細胞比率は、免疫細胞の浸潤などにより変化するため、疾患の病型など、個体の状態を表現する重要な情報です。一方、組織中の細胞比率を実測する方法論は、多くの場合、新鮮組織を対象としており、ビッグデータの蓄積や臨床現場での取得は困難でした。本研究グループは、遺伝子を言葉として扱い、同じ言葉として与えられるマーカー遺伝子名を効果的に活用することで、凍結組織などからも取得可能であり、また公共データベース(公共DB)にも豊富に蓄積されているトランスクリプトームデータより、多くの細胞種の比率を推定する方法論を開発しました。本研究成果は7月10日、国際科学誌「Briefings in Bioinformatics」に掲載されました。
論文情報
Iori Azuma, Tadahaya Mizuno*, Hiroyuki Kusuhara, “” GLDADec: marker-gene guided LDA modelling for bulk gene expression deconvolution “,” Briefings in Bioinformatics: 2024年7月10日, doi:10.1093/bib/bbae315.
論文へのリンク (掲載誌)