カリフォルニア大学リバーサイド校の研究により、SARS-CoV-2やその他のコロナウイルスに対抗する効果的な薬剤の設計に役立つ可能性があります。 UC Riverside study could inform the design of effective drugs to fight SARS-CoV-2 and other coronaviruses
2022-10-03 カリフォルニア大学リバーサイド校(UCR)
Viruses誌に掲載された論文の中で、SARS-CoV-2の構成要素からの組み立てと形成についての全体像を提示している。
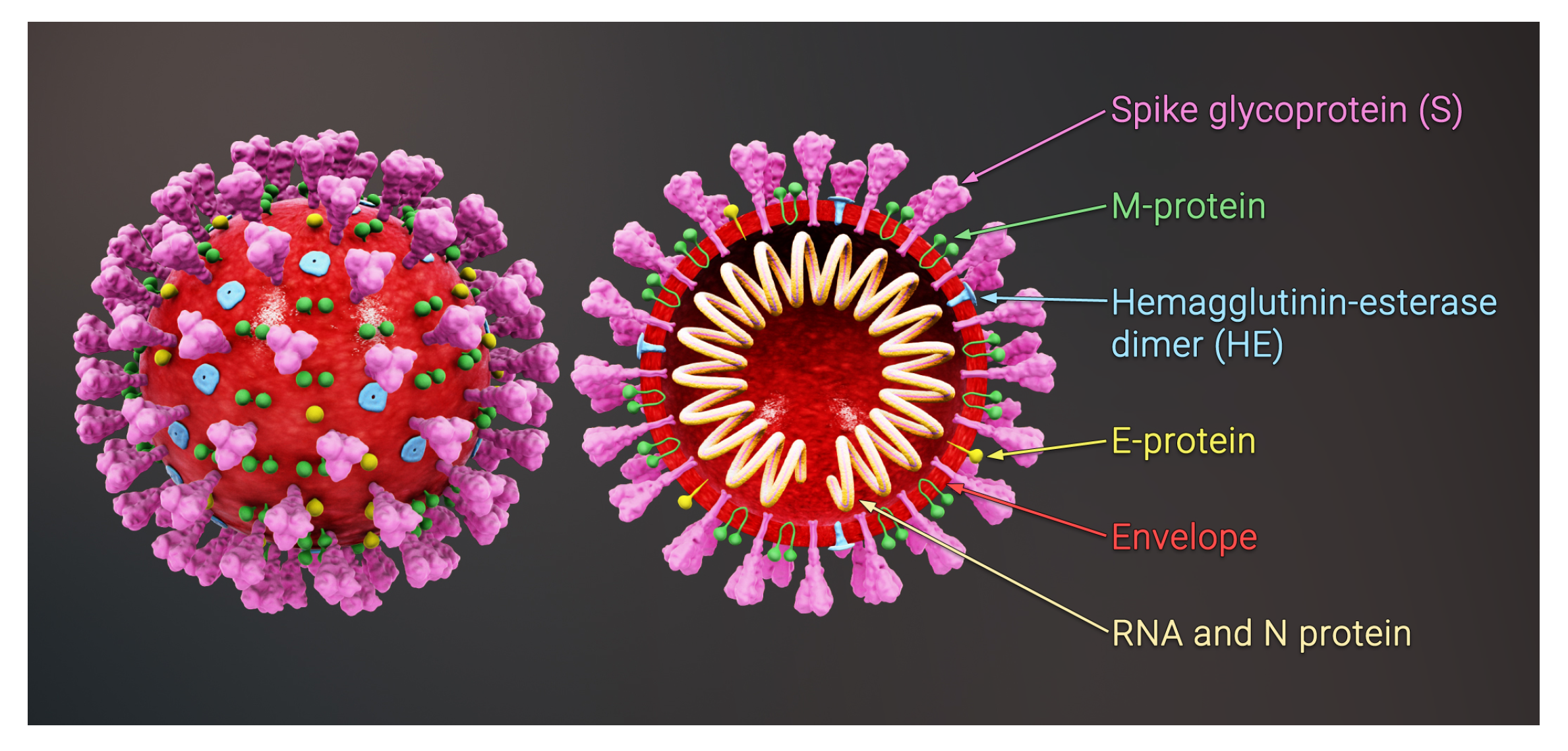
SARS-CoV-2には4つの構造タンパク質がある。エンベロープ(E)、膜(M)、ヌクレオキャプシド(N)、スパイク(S)である。構造タンパク質M、E、Nは、ウイルスを保護し、宿主細胞への侵入を容易にするウイルスの最外層であるエンベロープの組み立てと形成に必須である。このプロセスは、コロナウイルスに脂質エンベロープを提供する複雑な膜システムである小胞体ゴルジ体中間部(ERGIC)の膜で起こる。
Nタンパク質はRNAを凝縮し、タンパク質とRNAの両方を含む分子の集合体であるコンパクト・リボ核タンパク質複合体を形成する。の複合体は、脂質膜に埋め込まれたMタンパク質と相互作用する。膜の一部が湾曲し始める「出芽」によって、ウイルス形成が完了する。
タンパク質のオリゴマー化、構造タンパク質によるRNAの凝縮、細胞膜とタンパク質の相互作用のメカニズムが解明された。
<関連情報>
- https://news.ucr.edu/articles/2022/10/03/coronavirus-formation-successfully-modeled
- https://www.mdpi.com/1999-4915/14/10/2089
SARS-CoV-2アセンブリの生物物理学的モデリング。ゲノムの凝縮と出芽 Biophysical Modeling of SARS-CoV-2 Assembly: Genome Condensation and Budding
Siyu Li and Roya Zandi
Viruses Published: 20 September 2022
DOI:https://doi.org/10.3390/v14102089
Abstract
The COVID-19 pandemic caused by the Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2) has spurred unprecedented and concerted worldwide research to curtail and eradicate this pathogen. SARS-CoV-2 has four structural proteins: Envelope (E), Membrane (M), Nucleocapsid (N), and Spike (S), which self-assemble along with its RNA into the infectious virus by budding from intracellular lipid membranes. In this paper, we develop a model to explore the mechanisms of RNA condensation by structural proteins, protein oligomerization and cellular membrane–protein interactions that control the budding process and the ultimate virus structure. Using molecular dynamics simulations, we have deciphered how the positively charged N proteins interact and condense the very long genomic RNA resulting in its packaging by a lipid envelope decorated with structural proteins inside a host cell. Furthermore, considering the length of RNA and the size of the virus, we find that the intrinsic curvature of M proteins is essential for virus budding. While most current research has focused on the S protein, which is responsible for viral entry, and it has been motivated by the need to develop efficacious vaccines, the development of resistance through mutations in this crucial protein makes it essential to elucidate the details of the viral life cycle to identify other drug targets for future therapy. Our simulations will provide insight into the viral life cycle through the assembly of viral particles de novo and potentially identify therapeutic targets for future drug development.