2023-01-03 ジョージア工科大学
eLifeに掲載された新しい論文では、生物科学部とコンピュータサイエンス部の研究者が、AF2Complexが手を貸すことができるかもしれないことを示しました。
個々のタンパク質の詳細な立体構造を予測できる機械学習ツールであるDeepMindのAlphaFold 2のモデルを基にしたAF2Complex(AlphaFold 2 Complexの略)は、複数のタンパク質の物理的相互作用を予測するために設計された深層学習ツールである。この予測により、AF2Complexは、どのタンパク質が互いに作用して機能的な複合体を形成する可能性が高いかを、これまでにないほど詳細に計算できるようになりました。
最新の研究では、研究者達は、この「計算顕微鏡」を使って、複雑なタンパク質合成・輸送経路を調べ、経路内のタンパク質が、最終的に新しく合成されたタンパク質をバクテリアの内部から外膜に輸送するためにどのように相互作用するかを明らかにし、実験が見逃しているかもしれないプレイヤーを特定しようと試みています。この経路の解明により、抗生物質や治療法の新しい標的が見つかるかもしれません。また、AF2Complexを利用して、この種の生物学研究全体を計算機で高速化するための基礎となる可能性もあります。
ロンドンにある人工知能研究所DeepMindが開発したAlphaFold 2は、タンパク質の構成要素であるアミノ酸のみを用いて、タンパク質の立体構造を正確に予測することができる深層学習ツールである。AF2Complexは、これらの構造を用いて、タンパク質が相互作用して機能的な複合体を形成する可能性、それぞれの構造のどの部分が相互作用部位になりそうか、さらにはどのタンパク質複合体がペアになってスーパーコンプレックスと呼ばれるさらに大きな機能グループを形成しそうなのかを予測することができます。
研究チームは、オークリッジ国立研究所のスーパーコンピュータ「Summit」を使って、AF2Complexの機能を検証した。OMPの合成と輸送に重要であることが知られているいくつかのタンパク質を、大腸菌の細胞外皮に存在する既知のタンパク質すべてである約1,500種類のタンパク質と比較し、このツールが最も相互作用しやすいと計算したペアと、その中でスーパーコンプレックスを形成しやすいペアを確認したのである。
研究チームは、AF2Complexの予測が正しいかどうかを判断するために、ツールの予測値と既知の実験データとを比較した。「Skolnick助教は、「心強いことに、計算機によるスクリーニングでヒットしたタンパク質のなかには、既知の相互作用パートナーが見いだされました」と述べている。相互作用が知られているタンパク質ペアであっても、AF2Complexは、過去の実験データを説明する相互作用の構造的詳細を強調することができ、ツールの精度にさらなる信頼性を与えることができました。
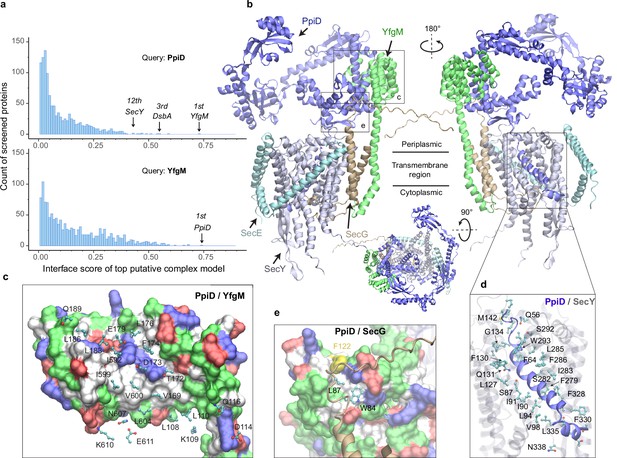
既知の相互作用に加え、AF2Complexはいくつかの未知のペアを予測しました。これらの未知のペアをさらに詳しく調べたところ、ペアのどの部分が相互作用してより大きな機能性タンパク質群を形成するのか、これまで実験者が理解できなかった複合体の活性構成が明らかになり、OMPが合成され輸送されるメカニズムに関する新しい可能性が見えてきました。
AF2Complexはオープンソースのツールとして公開されており、ここからダウンロードすることができます。
<関連情報>
- https://research.gatech.edu/af2complex-computational-microscope-predicts-protein-interactions-potential-paths-new-antibiotics
- https://elifesciences.org/articles/82885
- https://github.com/FreshAirTonight/af2complex
ディープラーニングによるバクテリアの外膜タンパク質生合成のためのスーパータンパク質複合体の解明 Deep learning-driven insights into super protein complexes for outer membrane protein biogenesis in bacteria
Mu Gao,Davi Nakajima An,Jeffrey Skolnick
eLife Published: Dec 28, 2022
DOI:https://doi.org/10.7554/eLife.82885
Abstract
To reach their final destinations, outer membrane proteins (OMPs) of gram-negative bacteria undertake an eventful journey beginning in the cytosol. Multiple molecular machines, chaperones, proteases, and other enzymes facilitate the translocation and assembly of OMPs. These helpers usually associate, often transiently, forming large protein assemblies. They are not well understood due to experimental challenges in capturing and characterizing protein-protein interactions (PPIs), especially transient ones. Using AF2Complex, we introduce a high-throughput, deep learning pipeline to identify PPIs within the Escherichia coli cell envelope and apply it to several proteins from an OMP biogenesis pathway. Among the top confident hits obtained from screening ~1500 envelope proteins, we find not only expected interactions but also unexpected ones with profound implications. Subsequently, we predict atomic structures for these protein complexes. These structures, typically of high confidence, explain experimental observations and lead to mechanistic hypotheses for how a chaperone assists a nascent, precursor OMP emerging from a translocon, how another chaperone prevents it from aggregating and docks to a β-barrel assembly port, and how a protease performs quality control. This work presents a general strategy for investigating biological pathways by using structural insights gained from deep learning-based predictions.