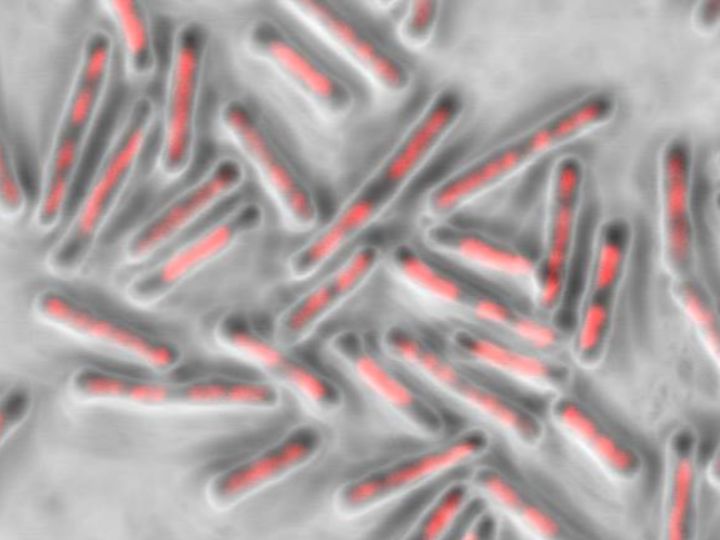
カリフォルニア大学サンディエゴ校の科学者が開発したツールは、新しい抗体医薬の開発を加速させる可能性があります。 Tools developed by UC San Diego scientists could accelerate the development of new antibody drugs.
2023-01-31 カリフォルニア大学サンディエゴ校(UCSD)
◆2023年1月28日にNature Communicationsに掲載されたこの研究で、研究者らはこのアプローチを用いて、既存の抗体医薬よりも17倍も強く主要ながんターゲットに結合する新しい抗体を同定しました。著者らは、このパイプラインにより、がんや、COVID-19、関節リウマチなどの他の疾患に対する新薬の発見が加速される可能性があると述べている。
◆医薬品として成功するためには、抗体はそのターゲットにしっかりと結合していなければならない。このような抗体を見つけるために、研究者は通常、既知の抗体のアミノ酸配列から始め、細菌や酵母の細胞を用いて、その配列の変異体を含む一連の新しい抗体を作り出す。そして、これらの変異体が、標的抗原と結合する能力を評価する。このサイクルは、結合力の強い最終候補が現れるまで繰り返される。
◆このように長い時間とコストがかかるにもかかわらず、出来上がった抗体の多くは臨床試験で効果を発揮することができない。今回の研究では、カリフォルニア大学サンディエゴ校の科学者たちが、こうした取り組みを加速・効率化するために、最先端の機械学習アルゴリズムを設計しました。
◆このアプローチは、研究者が約50万個の抗体配列からなる初期ライブラリを作成し、特定のタンパク質標的に対する親和性をスクリーニングするところから始まります。しかし、このプロセスを何度も繰り返すのではなく、データセットをベイジアンニューラルネットワークに送り込み、その情報を解析して、他の配列の結合親和性を予測するのである。
◆このパイプラインを検証するため、プロジェクト科学者で本研究の共同筆頭著者であるJonathan Parkinson博士とRyan Hard博士は、がんで高発現し、いくつかの市販抗がん剤の標的となっているタンパク質、プログラム死リガンド1(PD-L1)に対する抗体を設計することに着手しました。このアプローチにより、研究者らは、米国食品医薬品局(FDA)から臨床使用が承認されている野生型抗体であるアテゾリズマブ(商品名テセントリク)よりも17倍PD-L1への結合が良好な新規抗体を同定しました。
◆研究者らは現在、この方法を用いて、SARS-CoV-2など、他の抗原に対する有望な抗体を同定している。また、安定性、溶解性、選択性など、臨床試験の成功に重要な他の抗体特性についてアミノ酸配列を解析するAIモデルも追加開発している。
<関連情報>
- https://today.ucsd.edu/story/artificial-intelligence-aids-discovery-of-super-tight-binding-antibodies
- https://www.nature.com/articles/s41467-023-36028-8
RESP AI モデルにより、タイトバインディング抗体の同定が加速される The RESP AI model accelerates the identification of tight-binding antibodies
Jonathan Parkinson,Ryan Hard & Wei Wang
Nature Communications Published:28 January 2023
DOI:https://doi.org/10.1038/s41467-023-36028-8
Abstract
High-affinity antibodies are often identified through directed evolution, which may require many iterations of mutagenesis and selection to find an optimal candidate. Deep learning techniques hold the potential to accelerate this process but the existing methods cannot provide the confidence interval or uncertainty needed to assess the reliability of the predictions. Here we present a pipeline called RESP for efficient identification of high affinity antibodies. We develop a learned representation trained on over 3 million human B-cell receptor sequences to encode antibody sequences. We then develop a variational Bayesian neural network to perform ordinal regression on a set of the directed evolution sequences binned by off-rate and quantify their likelihood to be tight binders against an antigen. Importantly, this model can assess sequences not present in the directed evolution library and thus greatly expand the search space to uncover the best sequences for experimental evaluation. We demonstrate the power of this pipeline by achieving a 17-fold improvement in the KD of the PD-L1 antibody Atezolizumab and this success illustrates the potential of RESP in facilitating general antibody development.