低分子を同定する新しいツールは、診断、創薬、基礎研究などに役立つ。 A new tool to identify small molecules offers benefits for diagnostics, drug discovery and fundamental research.
2022-12-20 フィンランド・アールト大学
代謝物と呼ばれる何千種類もの低分子は、エネルギーを輸送し、細胞情報を人体全体に伝達しています。代謝物は非常に小さいため、血液サンプル分析では互いに区別することが困難です。しかし、これらの分子を特定することは、運動、栄養、アルコールの使用、代謝異常が健康にどのように影響するかを理解する上で重要です。
代謝物の同定は、通常、液体クロマトグラフィー質量分析法と呼ばれる分離技術で質量と保持時間を分析することによって行われます。この技術では、まずサンプルをカラムに通すことで代謝物を分離し、その結果、測定装置での流速(保持時間)が異なります。次に質量分析計を用いて、質量に応じて代謝物を分類し、同定作業を微調整します。また、タンデム質量分析法と呼ばれる技術により、代謝物を細かく分解して成分を分析することもできる。
このたび、Rousu教授の研究グループは、低分子を同定するための新しい機械学習モデルを開発した。これは最近『Nature Machine Intelligence』誌に掲載された。
この新しいオープンソースのモデルは、研究コミュニティ全体に、低分子についての豊かな見方を提供します。糖尿病などの代謝異常や癌を特定する方法の研究にも役立つでしょう」と、Rousuは言う。
この新しいアプローチは、従来の方法が直面していた問題の1つをエレガントに回避している。分子の保持時間は研究室によって異なるため、研究室間でデータを比較することができないのだ。アールト大学の博士課程に在籍するEric Bachは、博士課程での研究中に、この問題を解決する代替策を考え出した。
私たちの研究から、絶対的な保持時間は変化しても、保持順序は異なるラボによる測定でも安定していることがわかりました」とBach氏は説明する。このため、代謝物に関する一般に公開されているすべてのデータを史上初めて統合し、機械学習モデルに送り込むことができたのです』。
世界中の数十の研究室からのデータを取り込むことで、機械学習モデルは、立体化学的変異体として知られる鏡像分子を識別するのに十分な精度を持つようになったのです」。これまで、識別ツールは立体化学的変異体を見分けることができなかったので、この新しい能力は、創薬などの分野で新しい道を開くと期待されています。
<関連情報>
- https://www.aalto.fi/en/news/scientists-use-machine-learning-to-gain-unprecedented-view-of-small-molecules
- https://www.nature.com/articles/s42256-022-00577-2
液体クロマトグラフィーの保持順とタンデム質量分析データを用いた低分子化合物の共同構造アノテーション Joint structural annotation of small molecules using liquid chromatography retention order and tandem mass spectrometry data
Eric Bach,Emma L. Schymanski &Juho Rousu
Nature Machine Intelligence Published:19 December 2022
DOI:https://doi.org/10.1038/s42256-022-00577-2
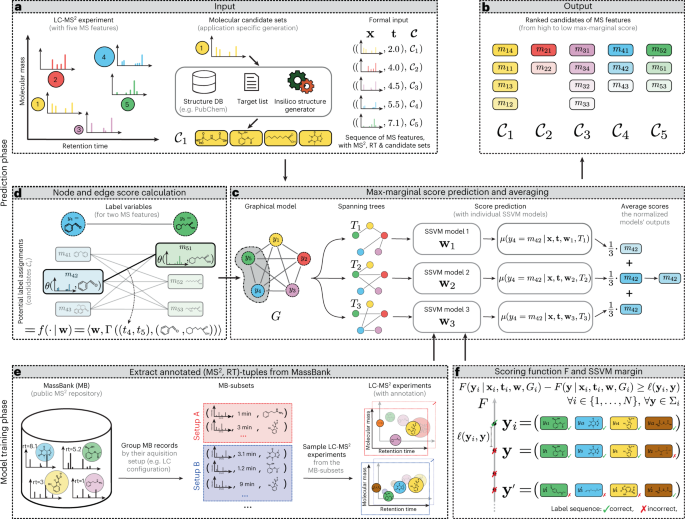
Abstract
Structural annotation of small molecules in biological samples remains a key bottleneck in untargeted metabolomics, despite rapid progress in predictive methods and tools during the past decade. Liquid chromatography–tandem mass spectrometry, one of the most widely used analysis platforms, can detect thousands of molecules in a sample, the vast majority of which remain unidentified even with best-of-class methods. Here we present LC-MS2Struct, a machine learning framework for structural annotation of small-molecule data arising from liquid chromatography–tandem mass spectrometry (LC-MS2) measurements. LC-MS2Struct jointly predicts the annotations for a set of mass spectrometry features in a sample, using a novel structured prediction model trained to optimally combine the output of state-of-the-art MS2 scorers and observed retention orders. We evaluate our method on a dataset covering all publicly available reversed-phase LC-MS2 data in the MassBank reference database, including 4,327 molecules measured using 18 different LC conditions from 16 contributors, greatly expanding the chemical analytical space covered in previous multi-MS2 scorer evaluations. LC-MS2Struct obtains significantly higher annotation accuracy than earlier methods and improves the annotation accuracy of state-of-the-art MS2 scorers by up to 106%. The use of stereochemistry-aware molecular fingerprints improves prediction performance, which highlights limitations in existing approaches and has strong implications for future computational LC-MS2 developments.