2025-11-21 ウィスコンシン大学マディソン校
<関連情報>
- https://news.wisc.edu/uw-researchers-turn-to-the-tiny-copepod-for-a-big-discovery-showing-that-gene-location-influences-natural-selection/
- https://www.nature.com/articles/s41467-025-65292-z
侵略的コペポーダ類種複合体におけるゲノム構造の進化 Genome architecture evolution in an invasive copepod species complex
Zhenyong Du,Johannes Wirtz,Yifei Joye Zhou,Anna Jenstead,Taylor Opgenorth,Angelise Puls,Cullan Meyer,Gregory W. Gelembiuk & Carol Eunmi Lee
Nature Communications Published:21 November 2025
DOI:https://doi.org/10.1038/s41467-025-65292-z
Abstract
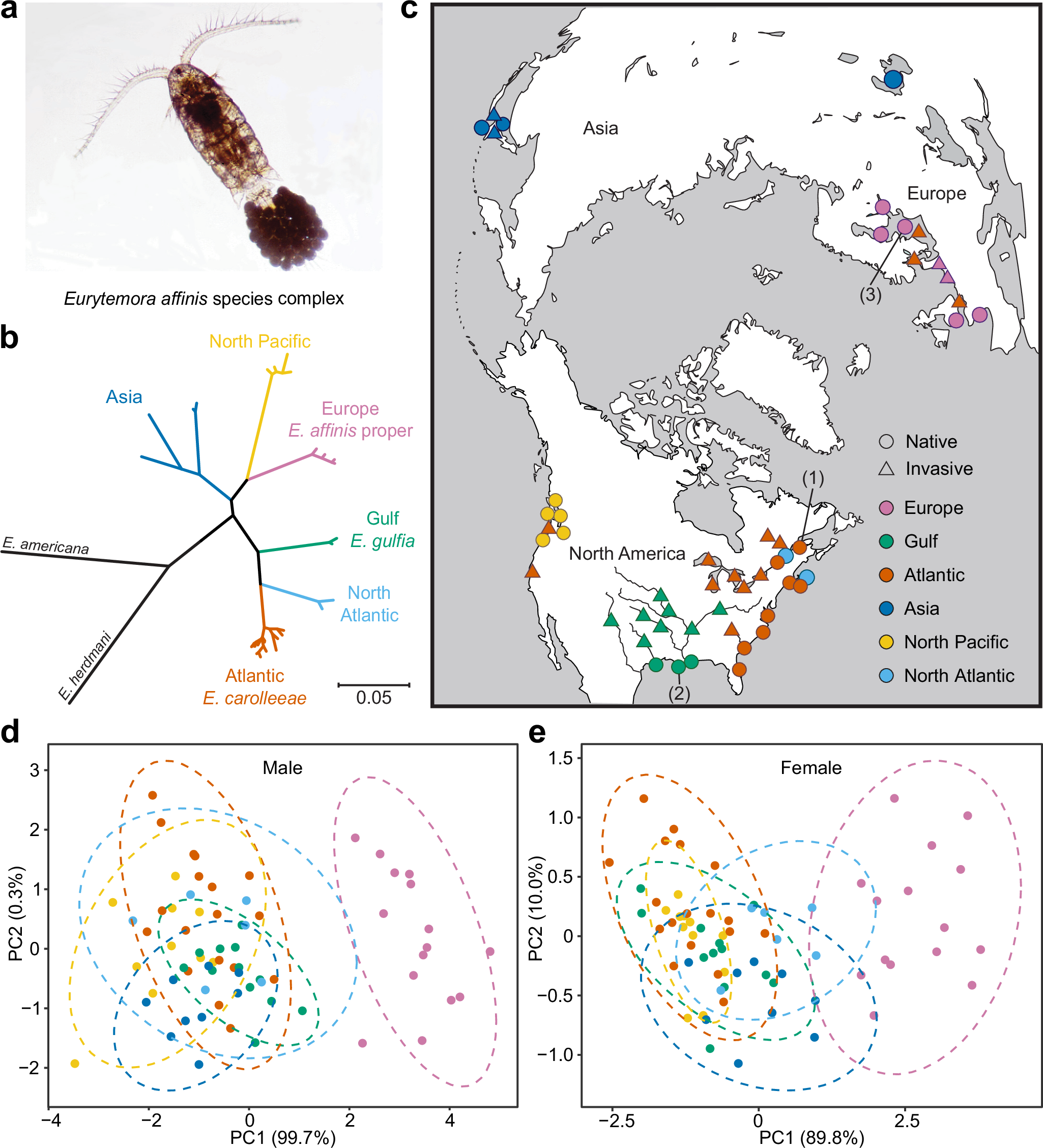
Chromosomal fusions are hypothesized to facilitate evolutionary adaptation, but empirical evidence has been scarce. Here, we analyze chromosome-level genome sequences of three sibling species within the copepod Eurytemora affinis species complex, known for its remarkable ability to rapidly colonize new habitats. Genomes of this species complex show expansions of ion transport-related gene families, likely related to adaptation to various environmental salinities. Among three genetically distinct sibling species, we discover notable patterns of chromosomal evolution, with chromosomal fusions observed in two different sibling species. As a result of these chromosomal fusions, functionally linked ion transport-related genes located near the telomeres become joined near the newly formed centromeres, where recombination is low. Notably, for the highly invasive E. carolleeae and to a lesser extent for E. gulfia, the ancient chromosomal fusion sites, especially the centromeres, are significantly enriched with contemporary signatures of selection between saline and freshwater populations. This study uncovers intriguing patterns of genome architecture evolution with potentially important implications for mechanisms of adaptive evolution in response to rapid environmental change.