(Accelerating biological systems design for sustainable biomanufacturing)
2020/6/15 アメリカ合衆国・ノースウェスタン大学
・ ノースウェスタン大学が、バイオマニュファクチャリングによるサステナブルな製品を数週間で作製する高速プロトタイピングシステム、「in vitro Prototyping and Rapid Optimization of Biosynthetic Enzymes (iPROBE)」を開発。
・ 気候変動、人口増加やエネルギー安全保障等の世界的課題の深刻化に伴い、サステナブルな資源で生産する低コストのバイオ燃料やバイオ製品への需要が高まっている。
・ これに対応するため、産業バイオテクノロジーでは、分子を化学物質に転換する酵素セットを活用した微生物セルファクトリーを利用しているが、セルの経路の設計、構築、最適化のサイクルが複雑で時間がかかることから、ダイナミックな需要の変化への対処が困難となっている。
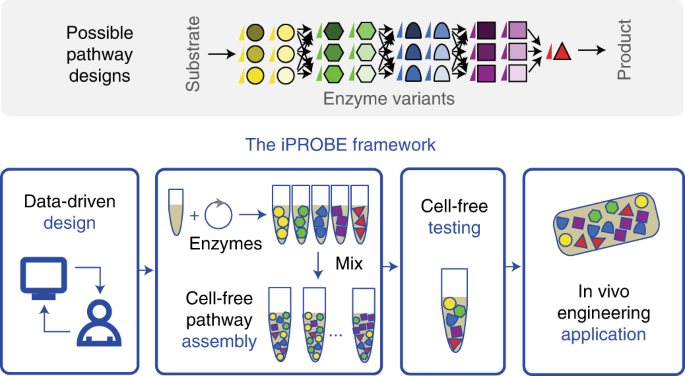
・ 今回開発した iPROBE プラットフォームは、セルフリーのシステムで最適なバイオ合成経路を発見する迅速で効果的な設計・構築・試験のフレームワークを提供し、様々な化学物質生成に最適な酵素セットの特定と大規模な物質製造を支援する。従来では 6~12 ヶ月を要したプロセスを約 2 週間で完了させ、クリーンエネルギーから消費者向製品に至るまで、多くの産業や課題に影響を及ぼす可能性がある。
・ バイオマニュファクチャリングでは、タンパク質酵素を組合せて個々の分子を変換し、グルコースやCO2 等から新しい化学物質を作製する。酵素を特定する試験では、DNA にエンコードした酵素をプラスミド分子に配置後、生きた細胞に挿入するが、最適な酵素グループを特定するためには酵素セット毎にこのようなプロセスを繰り返す必要がある。
・ セルフリーの iPROBE プラットフォームでは、タンパク質合成を利用して試験管で物質を転換する生合成酵素を強化することで、微生物エンジニアリングでの制限を回避。併用する Lockheed Martin 社開発の設計アルゴリズムが、経路となる酵素の割合を迅速に精査し、個々の酵素を任意の多段階経路に変換して高性能な酵素を選別・特定する。このような各種酵素のミックス・アンド・マッチ分析により、数百件もの生合成経路の試験が可能に。
・ 同プラットフォームの実証では、CO の代謝でエタノールを自然に生成するクロストリジウム・オートエタノゲナムの 2 種類の有機化合物である、3-ヒドロキシ酪酸(3-HB)とブタノールの最適な生合成経路を特定。その後、クロストリジウム株によるサステナブルな燃料生成を専門とする Lanzatech 社の協力で、特定した経路が生成する 3-HB が 20 倍増加したことを発見。また、リモネンを生成する酵素経路の構築や、E.coli セルを使用しないスチレンの高収率生成も実証した。
・ 本研究は、米国エネルギー省(DOE)科学局の生物環境科学室(BER)が支援した。
URL: https://www.mccormick.northwestern.edu/news/articles/2020/06/accelerating-biologicalsystems-design-for-sustainable-biomanufacturing.html
<NEDO海外技術情報より>
(関連情報)
Nature Chemical Biology 掲載論文(アブストラクトのみ:全文は有料)
In vitro prototyping and rapid optimization of biosynthetic enzymes for cell design
URL: https://www.nature.com/articles/s41589-020-0559-0
Metabolic Engineering 掲載論文(アブストラクトのみ:全文は有料)
Cell-free prototyping of limonene biosynthesis using cell-free protein synthesis
URL: https://www.sciencedirect.com/science/article/abs/pii/S109671762030094X?via%3Dihub
Metabolic Engineering 掲載論文(アブストラクトのみ:全文は有料)
Cell-free styrene biosynthesis at high titers
URL: https://www.sciencedirect.com/science/article/abs/pii/S1096717620300975
Abstract
The design and optimization of biosynthetic pathways for industrially relevant, non-model organisms is challenging due to transformation idiosyncrasies, reduced numbers of validated genetic parts and a lack of high-throughput workflows. Here we describe a platform for in vitro prototyping and rapid optimization of biosynthetic enzymes (iPROBE) to accelerate this process. In iPROBE, cell lysates are enriched with biosynthetic enzymes by cell-free protein synthesis and then metabolic pathways are assembled in a mix-and-match fashion to assess pathway performance. We demonstrate iPROBE by screening 54 different cell-free pathways for 3-hydroxybutyrate production and optimizing a six-step butanol pathway across 205 permutations using data-driven design. Observing a strong correlation (r = 0.79) between cell-free and cellular performance, we then scaled up our highest-performing pathway, which improved in vivo 3-HB production in Clostridium by 20-fold to 14.63 ± 0.48 g l-1. We expect iPROBE to accelerate design–build–test cycles for industrial biotechnology.