2024-08-09 東京大学医科学研究所
発表のポイント
- 日本発のゲノム編集技術CRISPR-Cas3を用いて、マウスやラットの受精卵において高効率に大規模なゲノム編集を行うことに成功しました
- 導入方法を最適化することでCRISPR-Cas3による数キロベースにおよぶゲノム欠失やゲノム導入を高効率で誘導できるようになった一方、意図しない変異は検出されませんでした
- 本成果によりCRISPR-Cas3を用いた様々な生物でのゲノム操作が可能になり、安全性の高い国産ゲノム編集技術として医療や産業への活用が期待されます
CRISPR-Cas3によって作出された遺伝子改変マウス
概要
東京大学医科学研究所先進動物ゲノム研究分野の吉見一人准教授、真下知士教授らと、筑波大学医学医療系、公益財団法人実中研動物資源技術センターおよび実験動物応用研究部による研究グループは、国産ゲノム編集技術であるCRISPR-Cas3(注1)を用いてマウスやラット受精卵で効率的に大規模なゲノム編集を行う新しい手法を開発しました。
この手法では、オフターゲット変異(注2)なしに、狙った場所に数キロベース規模の大規模欠失を40%~70%と高効率で導入することができます。また、Nanopore装置を用いたロングリードシーケンス(注3)による多重遺伝子型判定法を確立し、一度に複数の大規模欠失を検出することも可能にしました。さらに、簡単な導入方法であるエレクトロポレーション法(注4)による受精卵への導入、標的配列の塩基の置き換えや、遺伝子配列を導入するノックインにも成功し、非常に汎用的な遺伝子改変動物の作製方法を構築しました。
本研究成果は、遺伝子クラスターや非コード領域などの大きなゲノム領域の機能解析を容易にし、疾患モデル動物の作製や遺伝子機能の理解に大きく貢献すると期待されます。加えて、様々な生物でのゲノム操作への応用も可能になり、安全性の高い国産ゲノム編集技術として医療や産業への活用が期待されます。
本研究成果は2024年8月8日、米国科学雑誌「Cell Reports Methods」オンライン版で公開されました。
発表内容
〈研究の背景〉
ヒト疾患のメカニズム解明に向けて、様々な遺伝子やゲノム調節領域などを操作した遺伝子改変動物の利用は欠かせません。CRISPR-Cas9をはじめとしたゲノム編集技術の登場により、遺伝子改変動物の作製効率は大幅に向上し、また実験手技も簡素化され、多くの研究者が利用するようになりました。その一方で、従来のゲノム編集ツールは主にDNAの二本鎖切断を通じて小さな変異を導入するため、大規模なゲノム領域の編集が難しいといった課題がありました。複数の標的ガイドRNAを用いた大規模ゲノム編集法などは知られていますが、オフターゲット領域への影響、ゲノム編集効率の低下、逆位や転座などのエラーが発生するリスクが課題となっており、新しいゲノム編集技術の開発、利用が望まれています。
近年、本研究グループは、クラス1に属する大腸菌由来CRISPR-Cas3がヒト細胞でゲノム編集できることを見出し、日本発のゲノム編集技術として確立しました。CRISPR-Cas3は、CRISPR-Cas9とは異なって大きくゲノムを削る特徴を持ち、さらに認識標的配列も長くオフターゲットへの影響も極めて低いと考えられています。そのため、安全性が高く確実に遺伝子を破壊できる国産ゲノム編集技術として医療応用、産業応用が期待されています。一方で、受精卵を含む生体への利用はこれまで報告されていませんでした。
このような背景から、本研究では、ゲノム編集技術CRISPR-Cas3を用いて、マウスやラットの受精卵において高効率に大規模なゲノム編集を行うことを目指しました。
〈研究の内容〉
CRISPR-Cas3による高効率ゲノム編集を実現させるため、Cap1修飾mRNAとcrRNA末端修飾を組み合わせました。Cap1構造がmRNAの安定性と翻訳効率を向上させ、CRISPR-Cas3構成因子の合成を促進しました。また、crRNAの両末端を2′-O-メチルとホスホロチオエート結合で修飾することで、細胞内での安定性を向上させました。これらを組み合わせ、マウスおよびラットの受精卵にマイクロインジェクション法により直接導入した結果、高効率(40%~70%)なゲノム編集を実現しました(図1)。実際に、マウスのチロシナーゼ(Tyr)遺伝子やラットのインターロイキン2受容体γ鎖(Il2rg)遺伝子を標的としたところ、数キロベース規模の大規模欠失が導入されていることが確認されました。Tyr欠失個体ではアルビノやモザイク状の毛色が、Il2rg欠失個体では胸腺の縮小および免疫細胞の消失が観察され、遺伝子機能も欠損していることが確認されました。さらに汎用性を向上させるため、エレクトロポレーション法による受精卵へのCRISPR-Cas3 mRNAの導入も試みました。その結果、マイクロインジェクション法に比べるとゲノム編集効率は低下する傾向がみられましたが、初心者でも実施可能な同手法が利用できることを示しました。
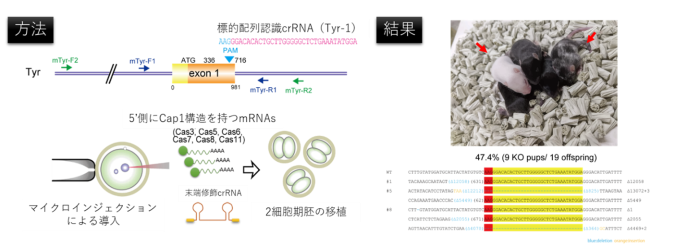
図1 CRISPR-Cas3によって作出されたTyr遺伝子KOマウスの作製
標的配列のデザインとマイクロインジェクション法によってRNAを導入し、二細胞期胚を移植した(左)。
得られたKO個体と標的部位のシーケンス結果(右)。
本研究を行う過程で、色々なパターンを示す大規模なゲノム改変を正確に検出することが難しい、といった課題が生じました。そこで、筑波大学の共同研究チームが開発してきたナノポアシーケンシングを用いた新しい多重遺伝子型判定法(DAJINシステム)を改良し、CRISPR-Cas3の大規模ゲノム改変を検出できる系をDAJIN2として確立しました(図2)。この方法により、従来のPCRベースの方法では困難だったキロベース単位の大規模な欠失だけでなく、欠失と同時に生じた挿入変異も正確かつ効率的に検出することができるようになりました。
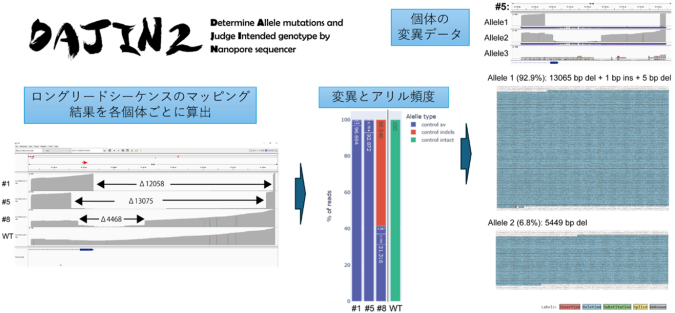
図2 DAJIN2を用いたCas3に誘導される大規模欠失パターンの解析自動化
各個体の変異パターンについて、その頻度と変異パターン配列を自動で抽出できる。
最後に、狙ったゲノム配列の導入、書き換えが可能であることを示すため、ドナープラスミドを用いたノックイン法の確立も試みました。マウスのRosa26遺伝子座にCAG-GFP-polyA-mCherryカセットを22.2%(2/9匹)の効率で挿入する、また、NOGマウス系統のc-Kit遺伝子において単一アミノ酸置換(V831C)を導入することにも成功し、遺伝子置換や一塩基置換などの精密な遺伝子改変も可能であることを示しました(図3)。
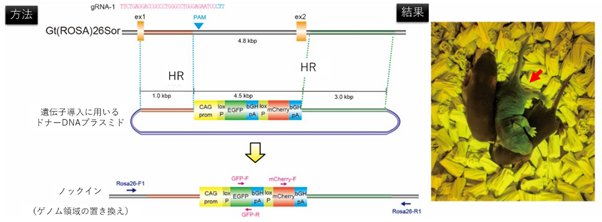
図3 Rosa26領域を蛍光配列に置換したノックインマウスの作製
ドナープラスミドと標的配列の設計(左)とGFP蛍光を示すノックインマウス個体(右)。
〈今後の展望〉
本研究では、国産ゲノム編集技術CRISPR-Cas3を用いた遺伝子改変動物作製方法を確立しました。本法は1つの標的認識crRNAで大きな欠失を導入することができるため、複数の遺伝子が隣接したクラスター遺伝子の機能解析や、エンハンサーやサイレンサーなどの非コード調節領域の機能解析が簡便になります。CRISPR-Cas3システムを用いて大規模ゲノム領域を操作した動物を作製することで、疾患メカニズムの理解や治療法の開発に貢献することができます。加えて、本法はマウスやラットに限らず様々な生物種に応用可能な技術です。国産ゲノム編集技術を用いた様々な生物でのゲノム操作は、生命現象を解明するような基礎研究はもちろんのこと、工業におけるバイオ生産の効率化、農水産業における品種改良、医学分野における遺伝子治療や新規薬剤開発など、幅広い分野における活用が期待されます。
〈関連のプレスリリース〉
「国産ゲノム編集技術CRISPR-Cas3が二本鎖DNAを切断する仕組みを解明」(2022/08/29)
https://www.ims.u-tokyo.ac.jp/imsut/jp/about/press/page_00191.html
「国産ゲノム編集技術CRISPR-Cas3を用いたCOVID-19迅速診断法の開発」(2022/01/30)
https://www.ims.u-tokyo.ac.jp/imsut/jp/about/press/page_00009.html
発表者・研究者等情報
東京大学医科学研究所 実験動物研究施設 先進動物ゲノム研究分野
真下 知士 教授
吉見 一人 准教授
筑波大学医学医療系/トランスボーダー医学研究センター(生命科学動物資源センター)
高橋 智 教授
久野 朗広 研究員
公益財団法人 実中研
動物資源技術センター
高橋 利一 センター長
後藤 元人 室長
実験動物応用研究部
伊藤 亮治 室長
論文情報
雑誌名:Cell Reports Methods
題 名:Genome editing using type I-E CRISPR-Cas3 in mice and rat zygotes
著者名:Kazuto Yoshimi*, Akihiro Kuno, Yuko Yamauchi, Kosuke Hattori, Hiromi Taniguchi, Kouya Mikamo, Ryuya Iida, Saeko Ishida, Motohito Goto, Kohei Takeshita, Ryoji Ito, Riichi Takahashi, Satoru Takahashi, Tomoji Mashimo*
(*Corresponding author)
DOI: 10.1016/j.crmeth.2024.100833
研究助成
本研究は、日本学術振興会(JSPS)科研費(課題番号:18H03974, 19KK0401, 22K19238, 23H00367, 24K02010, 22H04922 (AdAMS))、国立研究開発法人科学技術振興機構COI-NEXT (JPMJPF2010)、 日本医療研究開発機構(AMED)(24bm12230009)からの支援を受けて実施されました。
用語解説
(注1)CRISPR-Cas3:
多くの細菌は、獲得性免疫に似た「CRISPR-Cas(クリスパー-キャス)システム」と呼ばれる防御システムを備えています。CRISPR-Cas3は、CRISPRシステムの中でクラス1に属するCRISPRシステムに属しており、複数タンパク質の複合体でDNAを人工的に切断する国産ゲノム編集ツールとして、2019年に報告しました。
(注2)オフターゲット変異:
ゲノム編集技術において、狙った特定の配列以外の場所に、意図しないDNA配列の変異が起こること。オフターゲット変異を最小限に抑えることが安全性の高いゲノム編集技術として重要とされています。
(注3)ロングリードシーケンス:
DNAやRNAの塩基配列を、従来に比べて長い断片を一度に解析する技術。本研究ではナノスケールの穴(ナノポア)を通過させることで高速に解読する技術Nanoporeシーケンシング法を利用しました。
(注4)エレクトロポレーション法:
電気パルスを用いて細胞膜に一時的な穴を開け、外部からDNAやRNAなどを細胞内に導入する方法。直接針を用いて核酸などを細胞内に導入するマイクロインジェクション法と異なり、手技が簡便で、汎用性が高い。
問合せ先
〈研究に関する問合せ〉
国立大学法人東京大学医科学研究所 実験動物研究施設 先進動物ゲノム研究分野
教授 真下 知士(ましも ともじ)
〈報道に関する問合せ〉
国立大学法人東京大学医科学研究所 プロジェクトコーディネーター室(広報)
国立大学法人筑波大学広報局
公益財団法人実中研 広報室