2025-01-27 理化学研究所
理化学研究所(理研)バイオリソース研究センター(BRC)植物-微生物共生研究開発チームの小泉 敬彦 客員研究員、市橋 泰範 チームリーダーらの共同研究グループは、オミクスデータ[1]に基づく未観測データの高精度かつ頑健な定量推定アルゴリズムを開発しました。
ある事象に関連した挙動を示す生体分子は、その事象を推定するための「バイオマーカー」として利用できます。網羅的な生体分子情報であるオミクスデータには膨大な数のバイオマーカー情報が潜在的に含まれます。しかし、オミクスデータを有効活用するには、膨大な変数やノイズの多さなど複数の技術的な問題が未解決のまま残されていました。
今回、共同研究グループは、確率論的なアプローチによって従来の推定手法が抱える技術的問題を克服し、推定精度の向上を実現しました。本研究の成果は、有用で膨大なオミクスデータを最大限に活用する技術として、医療の高品質化や農業および環境管理技術のスマート化に貢献すると期待できます。
本研究は、科学雑誌『Scientific Reports』(1月27日付:日本時間1月27日)に掲載されました。
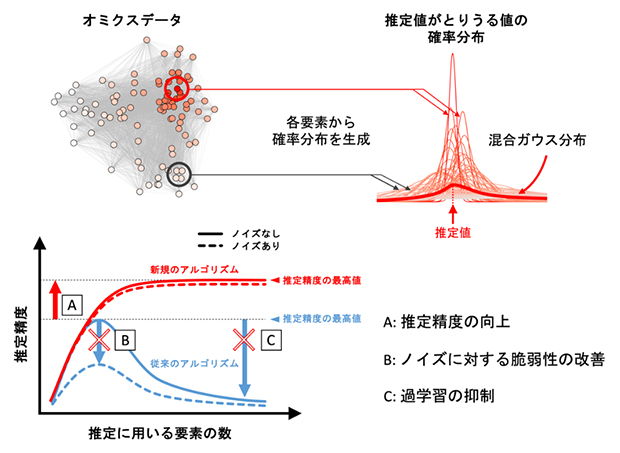
遺伝子に関するオミクスデータで構築する「バイオセンサー」
背景
生体分子は、生物の生理状態を反映した挙動を示します。例えば医学分野では、特定のタンパク質やマイクロRNA[2]が、視認できない疾患の有無や症状の進行度を推定するバイオマーカーとして広く利用されています。ただし、従来のバイオマーカーとして利用される生体分子は、過去の知見によって着目する事象との関連が明確なものに限られ、一つの事象につき少数のバイオマーカーが特定されるに過ぎませんでした。しかし、少数のバイオマーカーでは小さな測定誤差が推定結果に大きく影響します。
近年の塩基配列解析における技術革新により、網羅的な生体分子情報である「オミクスデータ」を容易に取得できるようになりました。オミクスデータには、これまで見逃されてきたような、潜在的にバイオマーカーとして機能し得る生体分子の情報が豊富に含まれています。これらの要素を最大限に活用することで、推定の精度をより向上させることが可能になると見込まれます。
通常、ある要素を説明変数(予測変数)として定量値を推定するには、教師データ[3]を基に回帰式[4]を組み立てます。説明変数には複数の要素を選択できますが、その数が多過ぎると教師データを過剰に学習してしまう「過学習」という現象に陥ります。これでは膨大な数の有用情報を含むオミクスデータの強みを生かせません。この現象を回避する数々の推定アルゴリズムが考案されていますが、いずれも過学習を原因とした新規のデータでの推定精度の低下、個体差や測定誤差により生じるデータの「ノイズ」による影響を抑えるには限界がありました。推定に関わるパラメータを調節することで多少はこれらの影響を軽減できますが、データを扱う個人の技量による部分が大きく「恣意(しい)的」になりやすい問題があります。
研究手法と成果
多数の説明変数を用いて推定する手法は、これまで回帰式の枠組みに基づいて発展してきました。しかし、前述したようにこうした手法には数々の問題が残されています。その点を踏まえて共同研究グループは、新たに確率論[5]的な枠組みの定量推定アルゴリズム「OmicSense」を開発しました。どんな物事も多かれ少なかれ一定の確率で「ばらつき」ます。このばらつきは、対象とする事象をより多く観測するほど正確に求めることができます。推定においては、このばらつきが小さいほど手がかりとして有用であると考えられます。この考えを軸とした推定手法により、前述した三つの問題(過学習、ノイズ、恣意性)を同時に克服することに成功しました。
従来の代表的な推定手法に比べて、OmicSenseによる推定値は常に誤差が小さい値を取りました(図1A)。また、過学習による推定誤差の増加は最小限に抑えられました(図1B)。さらに、教師データにノイズを付加した場合も推定誤差の増加が最小限に抑えられました(図1C)。これらにより、OmicSenseは過学習に陥ることのない、ノイズに対して頑健な推定アルゴリズムであることが実証されました。オミクスデータの取得過程においていずれの要素にもノイズは混在し、それを取り除くことは実質的に不可能です。そのためOmicSenseはオミクスデータを扱うには最適な定量推定アルゴリズムであるといえます。また、OmicSenseは特別なパラメータ調整の手順を踏まないため、恣意性を排除することも達成できました。
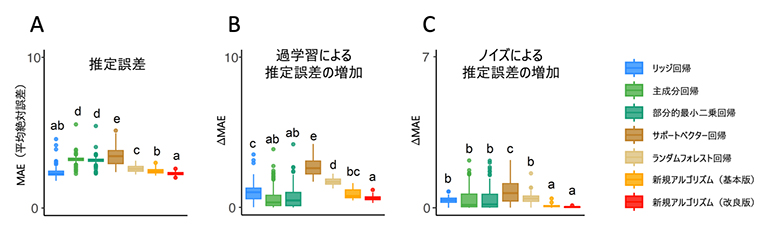
図1 アルゴリズム間での推定精度の比較
異なるアルゴリズムの間で、(A)推定誤差、(B)過学習による推定誤差の増加、(C)ノイズによる推定誤差の増加をそれぞれ比較した。新規アルゴリズム(OmicSense)の改良版では、説明変数と推定量の間に非線形性を考慮している。異なる小文字アルファベットは多重比較(3標本以上の比較において順位を付けることを目的にしている)により5%水準で統計的に有意な違いが認められたことを示す。
OmicSenseは既存のバイオマーカーに加えて、オミクスデータから潜在的なバイオマーカーを検出します。植物の遺伝子発現に関するオミクスデータを例に検証し、OmicSenseが遺伝子共発現ネットワーク[6]上における関連遺伝子の周辺遺伝子を推定要素として選択していることが確かめられました(図2)。
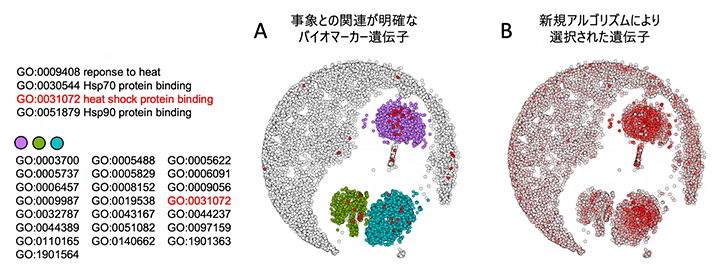
図2 遺伝子に関するオミクスデータで構築するバイオセンサー
発現パターンの類似性に基づいて可視化した遺伝子のネットワーク。GOは遺伝子オントロジー(分類体系)を表し、識別番号によって遺伝子の機能を分類する。
(A)既存のバイオマーカー遺伝子の選択。赤円は温度に関連する機能遺伝子を示す。
(B)新規アルゴリズム(OmicSense)は、既存のバイオマーカー遺伝子に関連の強い遺伝子を優先しながらはるかに多数の遺伝子(赤円)を選択する。
加えて、OmicSenseは細胞単位の遺伝子発現、組織における代謝産物、環境中の微生物叢(びせいぶつそう)など、幅広いスケールで得られるオミクスデータに対しても適用できることが示されました(図3)。このことは、学問領域の垣根を越えてオミクスデータを活用する場面が広がる可能性を示しています。
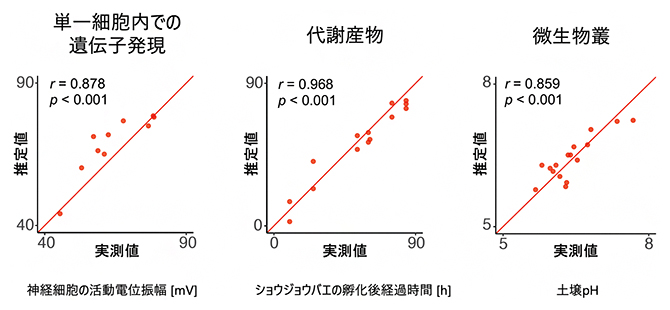
図3 OmicSenseの各種オミクスデータへの適用
対象の異なるオミクスデータにOmicSenseを適用して推定を行った。「単一細胞内での遺伝子発現」「組織における代謝産物」「環境中の微生物叢」のいずれにおいても実測値と推定値との間に高い相関(r>0.8)が認められた。p値:0.05(5%水準)より小さい値であると有意であることを示す。
さらに共同研究グループは、オープンソースの統計解析ソフトウェアRにおいてOmicSenseを実装するパッケージ(Rパッケージ)を作成して公開しました。これにより、誰でもOmicSenseの技術を利用することができます(図4)。
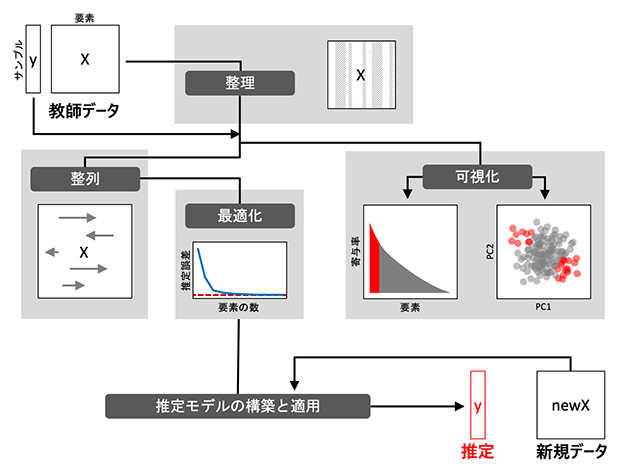
図4 OmicSenseを実装するRパッケージ
オープンソースの統計解析ソフトRを使用してOmicSenseに基づく推定を実行することができる。本パッケージを使用することで、必要に応じて推定の中間プロセスを確認し、データを可視化することができる。
今後の期待
本研究では、確率論的な枠組みによるアルゴリズムによってオミクスデータの強みを生かす定量推定を実現しました。これにより、網羅的な生体分子情報であるオミクスデータに潜む無数のバイオマーカーを統合し、バイオセンシング技術として利用できる「バイオセンサー」を構築することが可能になります。さらに、本アルゴリズムを実装するRパッケージを作成・公開したことにより、オミクスデータ活用の基盤を整えました。
バイオマーカー技術は、今や医療だけでなく、ヘルスケア、農業、環境分野にも広がりを見せています。このビッグデータの時代にオミクスデータをバイオセンサーとして活用する試みは、貴重なデータを最大限に生かし、医療の質の向上や農業・環境管理技術のスマート化に貢献するものと期待されます。
補足説明
1.オミクスデータ
生物学の分野において、特定の種類の生体分子(遺伝子や代謝産物など)に関して網羅的に解析することで得られる情報。
2.マイクロRNA
タンパク質に翻訳されない短いRNA分子。遺伝子の発現調節に関与することから、医学分野では疾患に関連するマイクロRNAがバイオマーカーとして利用されている。
3.教師データ
正解となる数値を伴ったデータであり、正解が導かれるように訓練することで推定モデルが構築される。
4.回帰式
ある変数を使って別の変数を推定するための数学的モデル。
5.確率論
ランダムな現象や不確実性を定量的に扱う数学の分野。
6.遺伝子共発現ネットワーク
遺伝子の発現パターンに基づいて構築されるネットワーク。遺伝子共発現ネットワークを解析することで、特定の生物学的プロセスや機能に関連する遺伝子群を特定することができる。
共同研究グループ
理化学研究所 バイオリソース研究センター
植物-微生物共生研究開発チーム
客員研究員 小泉 敬彦(コイズミ・タカヒコ)
(学術振興会特別研究員PD(研究当時)、現 東京農業大学 生命科学部 助教)
チームリーダー 市橋 泰範(イチハシ・ヤスノリ)
(環境資源科学研究センター ホロビオント・レジリエンス研究チーム チームリーダー)
テクニカルスタッフ(研究当時)熊石 妃恵(クマイシ・キエ)
統合情報開発室
研究員 鈴木 健大(スズキ・ケンタ)
(横浜国立大学 総合学術高等研究院 客員准教授)
日本大学文理学部
准教授 井上 みずき(イノウエ・ミズキ)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業特別研究員奨励費「樹木-菌根菌の共生バイオセンサーによる森林の機能的コミュニティ構造の解明(研究代表者:小泉敬彦)」、同若手研究「トランスクリプトームを用いた外生菌根菌の共生パフォーマンス評価手法の確立(研究代表者:小泉敬彦)」、内閣府ムーンショット型農林水産研究開発事業「土壌微生物叢アトラスに基づいた環境制御による循環型協生農業プラットフォーム構築(研究代表者:竹山春子、栽培マネジメントチーム代表者:市橋泰範)」、理研TRIPユースケースフィールドオミクスによる助成を受けて行われました。
原論文情報
Takahiko Koizumi, Kenta Suzuki, Inoue Mizuki, Kie Kumaishi, and Yasunori Ichihashi, “A quantitative prediction method utilizing whole omics data for biosensing”, Scientific Reports, 10.1038/s41598-024-84323-1
発表者
理化学研究所
バイオリソース研究センター 植物-微生物共生研究開発チーム
客員研究員 小泉 敬彦(コイズミ・タカヒコ)
チームリーダー 市橋 泰範(イチハシ・ヤスノリ)
報道担当
理化学研究所 広報室 報道担当