渡航禁止は、深刻度の推定に依存するため、変種を発生源で封じ込めるのに、ほとんど効果がない。 Travel bans, relying on estimates of severity are largely ineffective at containing variants at their source
2022-10-11 ジョージア大学 (UGA)
PNAS Nexusに掲載されたこの研究では、新しい変異体の重大性を最初に評価する標準的な方法では、その拡散を阻止するには時間がかかりすぎることが判明した。
COVID-19の場合、いくつかの新しい変異体、特に懸念される変異体は、その遺伝子コードに明確な違いがある。つまり、研究者は遺伝暗号を利用して、新たに問題となる変異体を検出することができる。
研究者らは、出現した変異型の影響をシミュレートする数学的モデルを開発し、集団内での初期伝播と、その後の他地域からの旅行者による拡散を追跡した。
研究者らは、ウイルスに感染していないことが確認されるまで、新たに入国したすべての人を隔離する程度の渡航禁止措置では、亜種が地理的に広がることを防ぐことはできないことも明らかにした。せいぜい、ある集団に流入する人の量を99%削減するような渡航禁止令では、亜種到着を約1カ月遅らせるだけであることが、この研究によって明らかになった。
<関連情報>
- https://news.uga.edu/genetic-sequencing-could-contain-covid-19-variants/
- https://academic.oup.com/pnasnexus/article/1/4/pgac159/6672588?login=false
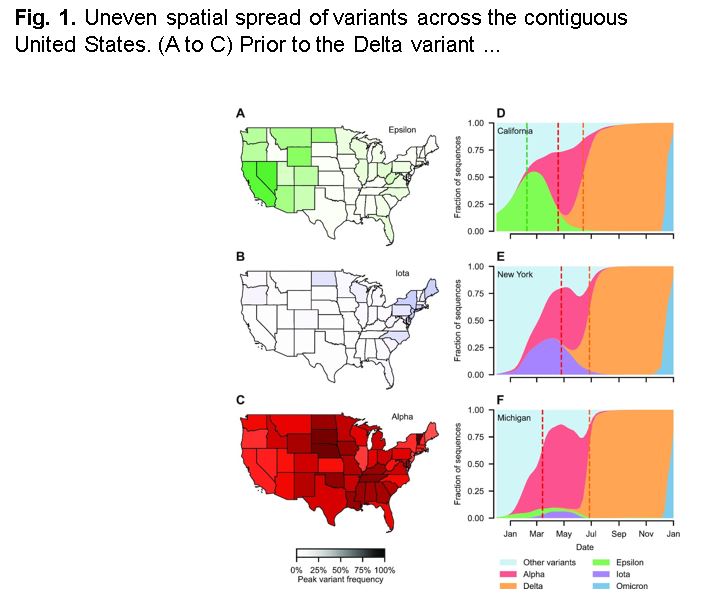
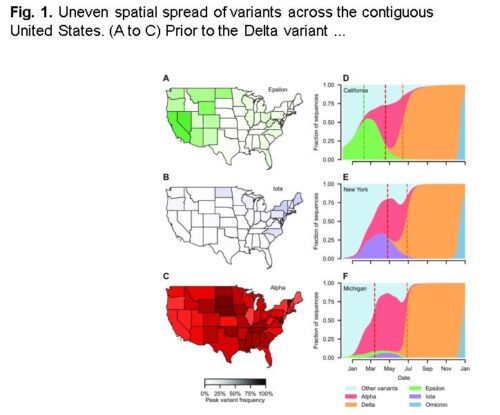
高密度シークエンスにより、SARS-CoV-2の新規変異体を感染源から検出することが可能になった。 Containing novel SARS-CoV-2 variants at source is possible with high-intensity sequencing
Tobias S Brett, Pejman Rohani
PNAS Nexus Published:19 August 2022
DOI:https://doi.org/10.1093/pnasnexus/pgac159
Abstract
Throughout the COVID-19 pandemic, control of transmission has been repeatedly thwarted by the emergence of variants of concern (VOC) and their geographic spread. Key questions remain regarding effective means of minimizing the impact of VOC, in particular the feasibility of containing them at source, in light of global interconnectedness. By analysing a stochastic transmission model of COVID-19, we identify the appropriate monitoring requirements that make containment at source feasible. Specifically, precise risk assessment informed primarily by epidemiological indicators (e.g. accumulated hospitalization or mortality reports), is unlikely prior to VOC escape. Consequently, decision makers will need to make containment decisions without confident severity estimates. In contrast, successfully identifying and containing variants via genomic surveillance is realistic, provided sequence processing and dissemination is prompt.
Significance Statement
As SARS-CoV-2 continues to circulate, the stochastic emergence of novel variants will pose a threat for the foreseeable future. Minimizing their impact requires identifying and containing each variant at source to halt onward circulation. Our results highlight that, due to the lag between exposure and death, identifying a novel variant using mortality data is unlikely before substantial spatial spread. On the other hand, our results suggest that by the start of 2022 multiple countries were sequencing sufficient infections to make successfully identifying and containing variants via genomic surveillance realistic, contingent on fast sequence processing, dissemination, and analysis times. As a consequence, while genomic surveillance makes early variant identification possible, it is not feasible to precisely quantify the threat posed before a proportionate containment response is needed.