2022-11-29 ローレンスリバモア国立研究所(LLNL)
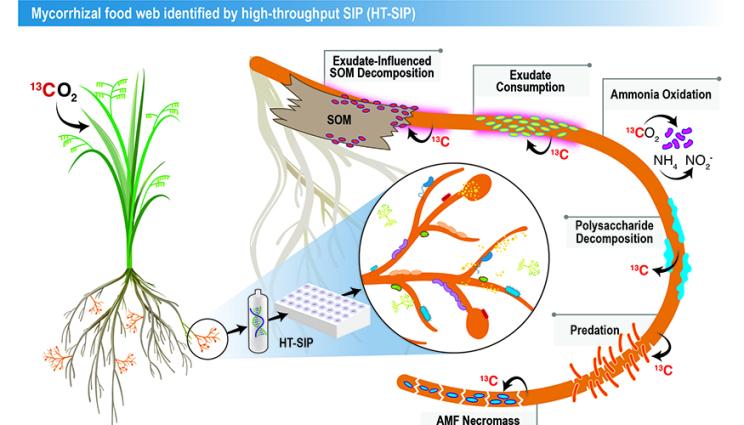
The researchers used the new high-throughput stable isotope probing (HT-SIP) pipeline and metagenomics to get the first look at the active microbiome surrounding a beneficial plant symbiont, arbuscular mycorrhizal fungi (AMF).
LLNLの科学者は、安定同位体プロービングのプロセスのいくつかのステップを自動化する新しい技術 – ハイスループットSIP – を開発し、ラボでの培養を必要とせず、現実的な条件での微生物の活性を調査できるようにしました。
SIPでは、安定同位体をバイオマスに取り込むことで、活性の高い微生物を同定します。SIP法は、複雑な生物群集の活性微生物とその生理的形質(基質利用、細胞生化学、代謝、成長、死亡率)を自然条件下で同定できるため、微生物生態学で最も強力な手法の一つです。
“私たちの半自動化アプローチは、作業者の時間を減らし、再現性を向上させます。”と、LLNL科学者のエリン・ヌッチオはは語っています。「我々は現在、このアプローチで1000以上のサンプルを処理しており、その中には非常に研究が進んでいない土壌のマイクロハビタットからのものも含まれています。
そのようなマイクロハビタットの一つが、菌根菌の組織のすぐ周りの土壌です。菌根菌は、全陸上植物の72%と共生関係を形成する菌類の一種です。この菌根菌は、植物の炭素と引き換えに、窒素、リン、水などの必須資源を宿主に供給している。Nuccioさんは、エネルギー省(DOE)の助成を受けた初期キャリア研究のために、このユビキタス菌と土壌マイクロバイオームとの相互作用について研究しています。
今回の概念実証研究では、土壌中の菌根菌によって刺激される相互作用の「食物網」を示しました。
<関連情報>
- https://www.llnl.gov/news/new-method-unearths-improved-understanding-soil-microbial-interactions
- https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-022-01391-z
HT-SIP:半自動安定同位体探査パイプラインによるアーバスキュラー菌根菌の下層圏におけるクロスドメイン相互作用の同定。 HT-SIP: a semi-automated stable isotope probing pipeline identifies cross-kingdom interactions in the hyphosphere of arbuscular mycorrhizal fungi
Erin E. Nuccio, Steven J. Blazewicz, Marissa Lafler, Ashley N. Campbell, Anne Kakouridis, Jeffrey A. Kimbrel, Jessica Wollard, Dariia Vyshenska, Robert Riley, Andy Tomatsu, Rachel Hestrin, Rex R. Malmstrom, Mary Firestone & Jennifer Pett-Ridge
Microbiome volume 10, Article number: 199 (2022)Published: 25 November 2022
Abstract
Background
Linking the identity of wild microbes with their ecophysiological traits and environmental functions is a key ambition for microbial ecologists. Of many techniques that strive for this goal, Stable-isotope probing—SIP—remains among the most comprehensive for studying whole microbial communities in situ. In DNA-SIP, actively growing microorganisms that take up an isotopically heavy substrate build heavier DNA, which can be partitioned by density into multiple fractions and sequenced. However, SIP is relatively low throughput and requires significant hands-on labor. We designed and tested a semi-automated, high-throughput SIP (HT-SIP) pipeline to support well-replicated, temporally resolved amplicon and metagenomics experiments. We applied this pipeline to a soil microhabitat with significant ecological importance—the hyphosphere zone surrounding arbuscular mycorrhizal fungal (AMF) hyphae. AMF form symbiotic relationships with most plant species and play key roles in terrestrial nutrient and carbon cycling.
Results
Our HT-SIP pipeline for fractionation, cleanup, and nucleic acid quantification of density gradients requires one-sixth of the hands-on labor compared to manual SIP and allows 16 samples to be processed simultaneously. Automated density fractionation increased the reproducibility of SIP gradients compared to manual fractionation, and we show adding a non-ionic detergent to the gradient buffer improved SIP DNA recovery. We applied HT-SIP to 13C-AMF hyphosphere DNA from a 13CO2 plant labeling study and created metagenome-assembled genomes (MAGs) using high-resolution SIP metagenomics (14 metagenomes per gradient). SIP confirmed the AMF Rhizophagus intraradices and associated MAGs were highly enriched (10–33 atom% 13C), even though the soils’ overall enrichment was low (1.8 atom% 13C). We assembled 212 13C-hyphosphere MAGs; the hyphosphere taxa that assimilated the most AMF-derived 13C were from the phyla Myxococcota, Fibrobacterota, Verrucomicrobiota, and the ammonia-oxidizing archaeon genus Nitrososphaera.
Conclusions
Our semi-automated HT-SIP approach decreases operator time and improves reproducibility by targeting the most labor-intensive steps of SIP—fraction collection and cleanup. We illustrate this approach in a unique and understudied soil microhabitat—generating MAGs of actively growing microbes living in the AMF hyphosphere (without plant roots). The MAGs’ phylogenetic composition and gene content suggest predation, decomposition, and ammonia oxidation may be key processes in hyphosphere nutrient cycling.