2024-03-27 理化学研究所,東京大学
理化学研究所(理研)環境資源科学研究センター 植物免疫研究グループの白須 賢 グループディレクター、増田 幸子 研究員、生命医科学研究センター 共生微生物叢研究チームの須田 亙 チームリーダー、東京大学 大学院新領域創成科学研究科の岩崎 渉 教授、木口 悠也 特任助教、按田 瑞恵 特任助教、同大学院農学生命科学研究科の佐々木 和浩 助教(研究当時)らの共同研究グループは、ロングリードメタゲノム解析[1]により植物共生微生物叢の遺伝情報を網羅的に調べ、その分子基盤を解明しました。
本研究成果は、ゲノム情報を用いた新規有用微生物の単離などにつながると期待されます。
植物には多くの微生物が共生しており、その中には窒素やリンを植物に供給するものや、他の植物病原菌の生育を抑えるものなど、植物の生育に重要なものがあることが知られています。しかし、これらの機能を発揮するのに重要な、微生物の遺伝情報についてはほとんど分かっていませんでした。今回、共同研究グループは、ロングリードシークエンサーを用いてイネ地上部の共生微生物叢のメタゲノム解析を行いました。得られた16S rRNA遺伝子[2]配列のうち約4分の3は新種のものでした。また、新規の完全長染色体、プラスミド[3]、バクテリオファージ[4]などの配列を得ました。新規のプラスミド上には、植物との相互作用に関わる4型分泌装置[5]などを見いだしました。また、難培養性細菌の完全長の染色体を得るなど、植物共生微生物叢の分子基盤を解明しました。
本研究は、科学雑誌『Communications Biology』オンライン版(3月27日付:日本時間3月27日)に掲載されました。
本研究の概要
背景
植物にはさまざまな微生物が共生していることが知られています。これまでに単離された共生微生物の中には、植物に窒素やリンを供給して生育を促進する微生物や、他の植物病原菌の生育を抑える微生物などが明らかになっています。しかし、自然環境中の99%以上の微生物はまだ単離されていないと考えられています。また、植物に共生している微生物は、人間と同じく「社会(叢:むら)」を形成しており、これを植物共生微生物叢(しょくぶつきょうせいびせいぶつそう)といいます。叢を構成する微生物は、叢の形成や維持のため、それぞれの役割を担っているはずですが、それは個々の菌の解析だけでは明らかにできません。植物共生微生物叢は、植物の生育に重要であるにもかかわらず、その機能に重要な情報、つまり叢を構成する微生物の種類、各微生物が持つ遺伝子の種類、それらの遺伝子を持つ染色体やプラスミドなどの情報の多くは未解明でした。
近年のシークエンス技術の発展により、微生物を単離することなく、網羅的に植物に共生している微生物ゲノム配列を読み取るメタゲノム解析が可能になりました。その結果、植物に共生している微生物の種類や遺伝子などが大まかに明らかになってきましたが、微生物種の同定には至っていません。なぜなら、これまでのメタゲノム解析では、ショートリードシークエンサー[6](解析対象のDNAを細かく断片化するため一度の読み取り長が数百塩基対と短い)が用いられています。微生物の種を同定するためには16S rRNA遺伝子の全長(約1,500塩基対)をシークエンスする必要がありますが、ショートリードでは一部の配列しかシークエンスできません。また、読み取り長を超える長い繰り返し配列やゲノム上に複数のコピー数がある配列、染色体とプラスミドにおける一部遺伝情報の重複を区別して、実際の配列に並べ替える(アセンブリ[7])ことが困難だったため、各微生物種や遺伝子情報の詳細は分からないままでした。
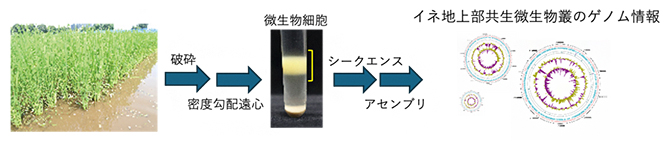
一方、最先端技術であるロングリードシークエンサーでは10万塩基対以上の長いDNA配列を得ることができます。実際に、ロングリードシークエンサーは、単離培養された微生物などのゲノム決定に用いられつつあり、上記のようなショートリードではアセンブリ困難な配列を克服し、完全な染色体やプラスミドなどを再構築することが可能となってきています。そのため、植物共生微生物叢においても、ロングリードシークエンサーを用いたメタゲノム解析を行うことで、長年不明であった、叢を構成する微生物種、染色体やプラスミド上の遺伝子情報などを得られると考えました。しかし、ロングリードシークエンサーでシークエンスするためには分解・断片化されていない微生物ゲノムを抽出する必要があります。また、植物共生微生物叢を構成する微生物の細胞数は、葉1cm2当たり106~108といわれており、いまだに種数は不明です。このような多種多様な微生物が生息する植物共生微生物叢から、未分解の微生物ゲノムを網羅的に抽出する方法、得られた配列を微生物ごとに分けるアセンブリ方法などは確立されていませんでした。今回、共同研究グループは、上記の問題を解決してロングリードシークエンサーを用いたメタゲノム解析を行い、植物共生微生物叢の基本的な分子基盤を明らかにしました。
研究手法と成果
本研究では、東京大学大学院農学生命科学研究科附属生態調和農学機構内の圃場で栽培した水稲の地上部に共生している微生物叢を解析対象としました。水稲の地上部を、直接ゲノムDNAの抽出に用いると、体積の大きい植物由来のゲノムDNAが非常に優勢となり、微生物叢由来のゲノムDNAはほとんど得られません。そのため、まず、植物に共生している微生物叢を植物から分離し、その後微生物叢からゲノムDNAを抽出する必要があります。そこで、サンプリングした水稲の地上部を破砕後、密度勾配遠心によって植物から微生物細胞を分離しました。一般的に微生物からゲノムDNAを抽出する場合は物理的に細胞を破砕してゲノムDNAを抽出しますが、物理的破砕を行うことでゲノムDNAは断片化します。そこで本研究では分離した微生物細胞を、酵素を用いて温和に破砕することで、未分解の微生物ゲノムを網羅的に抽出することに成功しました。抽出したゲノムをロングリードシークエンサー(PacBio Sequel Ⅱ)でシークエンスし、得られた約1.8億塩基対のアセンブリを行い、約26,000個のコンティグ[7]を構築しました。100万塩基対以上の染色体レベルでのコンティグが187個、完全長のコンティグが142個得られ、ショートリードのメタゲノム解析では難しい染色体レベルでのアセンブリに成功しました。これらのコンティグから669個の完全長16S rRNA遺伝子の配列を得ることができました。そのうち約4分の3の配列が一般的に公開されている配列との配列類似性が低い(97%未満)ことが明らかになり、これらは新種であると考えられました(図1)。
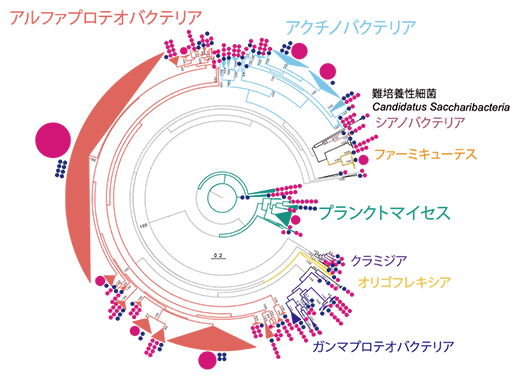
図1 16S rRNA遺伝子配列に基づいた系統樹
ピンクで示したものが今回見つかった新たな配列。ロングリードメタゲノム解析により多種多様な16S rRNA配列が明らかになった。
一般的に、自然環境中に存在する微生物の種と割合(群集構造)を調べるには、ポリメラーゼ連鎖反応(PCR)により16S rRNA遺伝子の一部(1,500塩基対のうち約300塩基対)を増幅させてシークエンスし、得られた16S rRNA遺伝子の数からその環境中での相対存在比を計算します。しかし、(1)PCRでの増幅条件が微生物によって異なる、(2)複数の16S rRNA遺伝子を持つ微生物は相対存在比が多く見積もられる、(3)微生物種を同定するためには16S rRNA遺伝子の全長をシークエンスする必要があり、一部の配列からは種まで同定するのが難しい、などの問題がありました。
一方、ロングリードメタゲノム解析では、PCRは行わずに抽出したゲノムをそのままシークエンスします。また、本研究では、染色体レベルでアセンブリできたため、正確に微生物種まで同定でき、また各微生物が持つ16S rRNA遺伝子数が分かりました。これにより、ロングリードメタゲノムを用いることで、イネ地上部の微生物群集構造を、種レベルの精密さで正確に明らかにすることができました。完全長のコンティグ142個のうち、プラスミドが100個、バクテリオファージが29個あり、ほとんどが新規の配列でした。プラスミドやバクテリオファージは微生物間をさまざまに移動します。移動先の微生物では、新しい遺伝子を獲得する、または既知の遺伝子の機能を失うなど、元々の形質に変化が起きます。共同研究グループは、新規プラスミド上に、植物との相互作用に関わる4型分泌装置を見いだしました(図2)。4型分泌装置は植物病原性微生物と非病原性の共生微生物の両方が共生に利用していることが明らかになっており、4型分泌装置を持つ微生物の種類は、植物と共生微生物叢の共生関係に大きな影響を与えます。得られた遺伝子の配列を既知のデータベースで検索すると、4型分泌装置を持つプラスミドはさまざまな微生物種を宿主とすることが示唆されました。従って、イネ地上部の共生微生物叢では、プラスミドを介して4型分泌装置が広く分布し、宿主植物との共生に利用されていると考えられました。
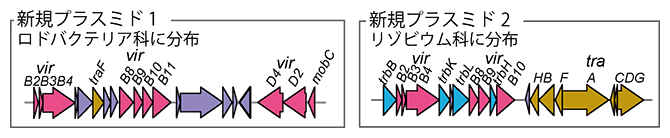
図2 新規のプラスミド上の4型分泌装置(一部プラスミドを抜粋)
これらの4型分泌装置は植物共生微生物叢で広く分布していると予想される。
また、本研究では、培養が難しい微生物Candidatus Saccharibacteriaの完全長染色体配列を得ることができました。得られた配列から代謝経路を予測したところ、他の難培養性微生物と同様に、微生物の生育に必須なTCAサイクルやビタミン合成系などを持たないことが明らかになりました(図3)。
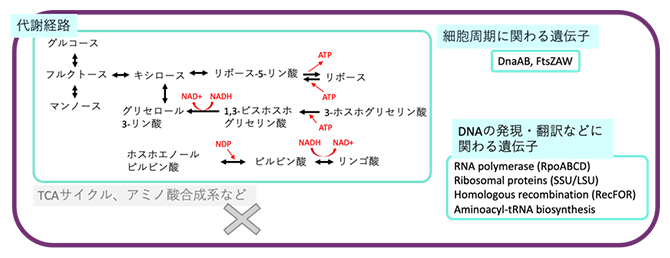
図3 ゲノム情報から予想される難培養性微生物の代謝経路
微生物Candidatus Saccharibacteriaの完全長染色体配列から代謝経路を予測した。TCAサイクルやアミノ酸合成系などは持たないと考えられる。
以上のように、共同研究グループはロングリードメタゲノム解析によって植物共生微生物叢の多様な微生物ゲノムの配列を解読し、その多くが新規の配列であることを明らかにしました。難培養性微生物の完全長染色体情報を明らかにするなど、ロングリードメタゲノム解析の有用性も示しました。
今後の期待
解明した植物共生微生物叢のゲノム情報を基に、新規の有用微生物の単離ができると考えられます。また、植物共生微生物叢以外の土壌や水系などの自然環境サンプルにも、ロングリードメタゲノムを用いることでいろいろな新規の配列が得られることが期待されます。
本研究成果は、国際連合が定めた17項目の「持続可能な開発目標(SDGs)[8]」のうち、「9.産業と技術革新の基盤を作ろう」、「15.陸の豊かさも守ろう」に貢献するものです。
補足説明
1.ロングリードメタゲノム解析
数万塩基対以上の長い核酸配列を大量にシークエンス(DNAを構成する塩基の順序を決定する技術)できるロングリードシークエンサーを用いて、特定環境中の核酸配列を網羅的に解析すること(メタゲノム解析)。長い塩基配列が得られるためアセンブリ([7]参照)がしやすくなり、染色体レベルでの繰り返し配列などのアセンブリが可能となってきている。
2.16S rRNA遺伝子
約1,500塩基対から成る、細菌の系統分類の指標となる遺伝子。全ての細菌が持つ。
3.プラスミド
細菌が持つ環状のDNA分子。可動性を持つため、細菌間をさまざまに移動する。プラスミド上には抗生物質耐性遺伝子などが存在し、受け取った微生物は新たな遺伝情報を獲得する。
4.バクテリオファージ
細菌に感染し、自身の遺伝情報を細菌細胞内に注入する。感染された細菌はファージ由来の遺伝情報を得ることがあるため新たな形質の獲得につながる。
5.4型分泌装置
微生物が宿主細胞にエフェクターと呼ばれるタンパク質を直接打ち込み、宿主細胞の免疫系をかく乱し侵入するシステム。主に病原性微生物が持つとされていたが、近年、非病原性細菌も持ち、宿主との共生に利用していることが明らかになった。
6.ショートリードシークエンサー
数百塩基対の塩基配列を大量にシークエンスすることが可能。一度にシークエンスできる塩基配列の長さが短いため、繰り返し出てくる配列などが続く場合はアセンブリが難しい。
7.アセンブリ、コンティグ
アセンブリとはバイオインフォマティクス手法の一つ。シークエンスし、得られたDNA配列断片をつないで、コンティグと呼ばれる長い塩基配列を構築する手法。
8.持続可能な開発目標(SDGs)
2015年9月の国連サミットで採択された「持続可能な開発のための2030アジェンダ」にて記載された、2016年から2030年までの国際目標。持続可能な世界を実現するための17のゴール、169のターゲットから構成され、発展途上国のみならず、先進国自身が取り組むユニバーサル(普遍的)なものであり、日本としても積極的に取り組んでいる(外務省ホームページから一部改変して転載)。SDGsはSustainable Development Goalsの略。
共同研究グループ
理化学研究所
環境資源科学研究センター 植物免疫研究グループ
グループディレクター 白須 賢(シラス・ケン)
(環境資源科学研究センター 副センター長)
研究員 増田 幸子(マスダ・サチコ)
研究員 パメラ・ガン(Pamela Gan)
テクニカルスタッフⅡ 柴田 ありさ(シバタ・アリサ)
生命医科学研究センター 共生微生物叢研究チーム
チームリーダー 須田 亙(スダ・ワタル)
東京大学
大学院新領域創成科学研究科
教授 岩崎 渉(イワサキ・ワタル)
特任助教 木口 悠也(キグチ・ユウヤ)
(理研 生命医科学研究センター マイクロバイオーム研究チーム 客員研究員)
特任助教 按田 瑞恵(アンダ・ミズエ)
大学院農学生命科学研究科
助教(研究当時)佐々木 和浩(ササキ・カズヒロ)
(現 国際農林水産業研究センター 主任研究員)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業学術変革領域研究(A)「不均一環境変動に対する植物のレジリエンスを支える多層的情報統御の分子機構(研究領域代表者:松下智直)」の計画研究「不均一土壌環境に応答した寄生植物の感染統御機構(研究代表者:吉田聡子)」、同基盤研究(A)「アポプラストにおける植物-病原体相互作用(研究代表者:白須賢)」、同新学術領域研究(研究領域提案型)「超地球生命体を解き明かすポストコッホ機能生態学(研究代表者:高谷直樹)」の公募研究「ロングリードメタゲノム解析による植物共生微生物叢のポストコッホ的理解(研究代表者:増田幸子)」による助成を受けて行われました。
原論文情報
Sachiko Masuda, Pamela Gan, Yuya Kiguchi, Mizue Anda, Kazuhiro Sasaki, Arisa Shibata, Wataru Iwasaki, Wataru Suda and Ken Shirasu, “Uncovering microbiomes of the rice phyllosphere using long-read metagenomic sequencing”, Communications Biology, 10.1038/s42003-024-05998-w
発表者
理化学研究所
環境資源科学研究センター 植物免疫研究グループ
グループディレクター 白須 賢(シラス・ケン)
研究員 増田 幸子(マスダ・サチコ)
生命医科学研究センター 共生微生物叢研究チーム
チームリーダー 須田 亙(スダ・ワタル)
東京大学
大学院新領域創成科学研究科
教授 岩崎 渉(イワサキ・ワタル)
特任助教 木口 悠也(キグチ・ユウヤ)
特任助教 按田 瑞恵(アンダ・ミズエ)
大学院農学生命科学研究科
助教(研究当時)佐々木 和浩(ササキ・カズヒロ)
報道担当
理化学研究所 広報室 報道担当
東京大学 大学院新領域創成科学研究科 広報室