2024-04-17 デューク大学(Duke)
<関連情報>
DNA折り紙の折り畳みのメカニズムがメゾスコピックシミュレーションによって解明される Mechanism of DNA origami folding elucidated by mesoscopic simulations
Marcello DeLuca,Daniel Duke,Tao Ye,Michael Poirier,Yonggang Ke,Carlos Castro & Gaurav Arya
Nature Communications Published:08 April 2024
DOI:https://doi.org/10.1038/s41467-024-46998-y
Abstract
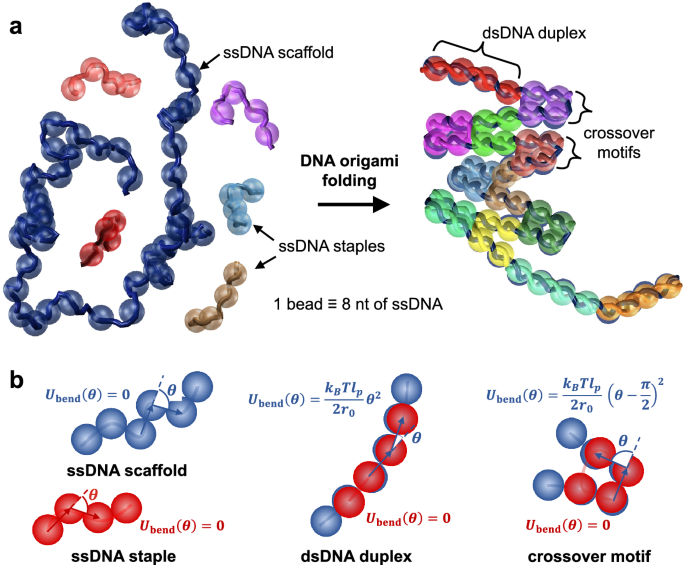
Many experimental and computational efforts have sought to understand DNA origami folding, but the time and length scales of this process pose significant challenges. Here, we present a mesoscopic model that uses a switchable force field to capture the behavior of single- and double-stranded DNA motifs and transitions between them, allowing us to simulate the folding of DNA origami up to several kilobases in size. Brownian dynamics simulations of small structures reveal a hierarchical folding process involving zipping into a partially folded precursor followed by crystallization into the final structure. We elucidate the effects of various design choices on folding order and kinetics. Larger structures are found to exhibit heterogeneous staple incorporation kinetics and frequent trapping in metastable states, as opposed to more accessible structures which exhibit first-order kinetics and virtually defect-free folding. This model opens an avenue to better understand and design DNA nanostructures for improved yield and folding performance.