2024-05-06 カリフォルニア大学サンディエゴ校(UCSD)
<関連情報>
- https://today.ucsd.edu/story/simulated-chemistry-new-ai-platform-designs-tomorrows-cancer-drugs
- https://www.nature.com/articles/s41467-024-47120-y
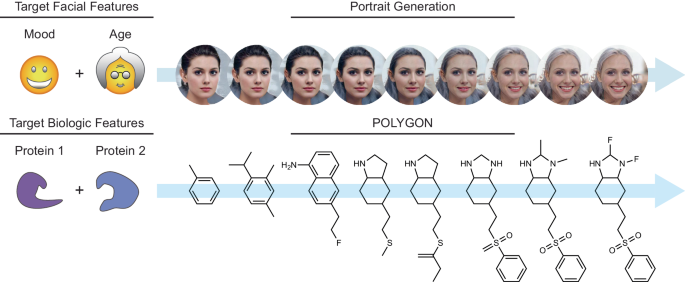
深層生成化学を用いたマルチターゲット化合物のデノボ生成 De novo generation of multi-target compounds using deep generative chemistry
Brenton P. Munson,Michael Chen,Audrey Bogosian,Jason F. Kreisberg,Katherine Licon,Ruben Abagyan,Brent M. Kuenzi & Trey Ideker
Nature Communications Published06 May 2024
DOIhttps://doi.org/10.1038/s41467-024-47120-y
Abstract
Polypharmacology drugs—compounds that inhibit multiple proteins—have many applications but are difficult to design. To address this challenge we have developed POLYGON, an approach to polypharmacology based on generative reinforcement learning. POLYGON embeds chemical space and iteratively samples it to generate new molecular structures; these are rewarded by the predicted ability to inhibit each of two protein targets and by drug-likeness and ease-of-synthesis. In binding data for >100,000 compounds, POLYGON correctly recognizes polypharmacology interactions with 82.5% accuracy. We subsequently generate de-novo compounds targeting ten pairs of proteins with documented co-dependency. Docking analysis indicates that top structures bind their two targets with low free energies and similar 3D orientations to canonical single-protein inhibitors. We synthesize 32 compounds targeting MEK1 and mTOR, with most yielding >50% reduction in each protein activity and in cell viability when dosed at 1–10 μM. These results support the potential of generative modeling for polypharmacology.


