2024-12-18 カリフォルニア工科大学 (Caltech)
カリフォルニア工科大学(Caltech)の研究者たちは、新たな技術を開発し、細胞核内の数百種類のタンパク質を同時にマッピングすることに成功しました。この技術は、細胞の機能や疾患の理解を深めるための強力なツールとなります。
◆従来の技術では、細胞核内のタンパク質の位置や相互作用を詳細に解析することは困難でした。しかし、今回の技術により、研究者たちは細胞核内の複雑なタンパク質ネットワークを高解像度で視覚化できるようになりました。
◆この技術は、がんや神経変性疾患など、さまざまな疾患の研究において、細胞内の異常なタンパク質の分布や機能を特定するのに役立つと期待されています。さらに、細胞生物学や分子生物学の基礎研究においても、細胞核内の構造と機能の関係を解明するための重要な手段となるでしょう。将来的には、この技術を用いて、個々の細胞の状態や疾患の進行状況をより正確に評価し、個別化医療の発展に寄与することが期待されています。
<関連情報>
- https://www.caltech.edu/about/news/new-technique-maps-hundreds-of-proteins-simultaneously-within-cell-nuclei
- https://www.nature.com/articles/s41588-024-02000-5
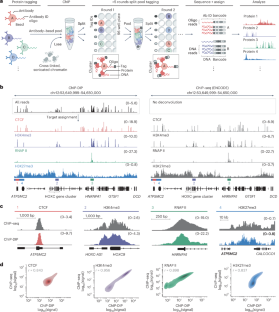
ChIP-DIPで数百のタンパク質とDNAの結合を同時にマッピングし、多様な遺伝子制御エレメントを同定 ChIP-DIP maps binding of hundreds of proteins to DNA simultaneously and identifies diverse gene regulatory elements
Andrew A. Perez,Isabel N. Goronzy,Mario R. Blanco,Benjamin T. Yeh,Jimmy K. Guo,Carolina S. Lopes,Olivia Ettlin,Alex Burr & Mitchell Guttman
Nature Genetics Published:25 November 2024
DOI:https://doi.org/10.1038/s41588-024-02000-5
Abstract
Gene expression is controlled by dynamic localization of thousands of regulatory proteins to precise genomic regions. Understanding this cell type-specific process has been a longstanding goal yet remains challenging because DNA–protein mapping methods generally study one protein at a time. Here, to address this, we developed chromatin immunoprecipitation done in parallel (ChIP-DIP) to generate genome-wide maps of hundreds of diverse regulatory proteins in a single experiment. ChIP-DIP produces highly accurate maps within large pools (>160 proteins) for all classes of DNA-associated proteins, including modified histones, chromatin regulators and transcription factors and across multiple conditions simultaneously. First, we used ChIP-DIP to measure temporal chromatin dynamics in primary dendritic cells following LPS stimulation. Next, we explored quantitative combinations of histone modifications that define distinct classes of regulatory elements and characterized their functional activity in human and mouse cell lines. Overall, ChIP-DIP generates context-specific protein localization maps at consortium scale within any molecular biology laboratory and experimental system.