2025-01-09 国立がん研究センター
発表のポイント
- 疾患原因・創薬標的として注目される「スプライスサイト生成変異」を、シークエンスデータから効率的に同定する新規情報解析手法を開発しました。
- 公共シークエンスデータレポジトリに格納されている30万件以上のデータの再解析を実施し、約3万件のスプライスサイト生成変異を同定しました。
- CRISPRゲノム編集細胞モデルの開発を通じて、検出した「スプライスサイト生成変異」が実際に核酸医薬の標的となることを示しました。
- 今後、さらなる増加が見込まれるトランスクリプトームデータに本手法を適用することで、疾患の原因となり、また核酸医薬等の標的となるゲノム変異をさらに多く同定できることが期待されます。
概要
国立研究開発法人国立がん研究センター(東京都中央区、理事長:中釜 斉) 研究所(所長:間野博行)ゲノム解析基盤開発分野の白石友一分野長らの研究グループは、難病・がん領域で非常に重要な変異のタイプである「スプライスサイト生成変異」注1を、トランスクリプトーム注2シークエンスデータから同定する新規情報解析手法(juncmut)を開発しました。さらに、世界中の研究で利用された膨大なデータを保管するSequence Read Archiveに登録されている322,072件のトランスクリプトームにこの手法を適用しました。その結果、30,130件のスプライスサイト生成変異を同定し、カタログ化しました。得られたカタログを活用し、「Aluエキソン化」を含むスプライスサイト生成変異に関連する多様な現象・性質を明らかにしました。
スプライスサイト生成変異は、配列特異的に設計することでスプライシングを制御できる核酸医薬の標的として近年注目を集めています。今回同定した変異の中から、NOTCH1遺伝子におけるイントロン領域上のスプライスサイト生成変異を選び、CRISPR注3ゲノム編集モデルを作成しました(同研究所分子病理分野の小林祥久研究員との共同研究)。これにより、該当の変異によりNOTCH1の活性化が生じること、さらに、その活性化が核酸医薬により抑制されることを実証しました。すなわち、開発した情報解析手法により、創薬のターゲットとなるゲノム変異を同定できることを示しました。
本研究で構成した変異カタログには、他にも数多くの疾患原因・創薬標的となるゲノム変異を含んでいることが期待され、今後のゲノム医療実装における重要なリソースとなります。また、今回開発した情報解析手法を今後ますます増加するトランスクリプトームデータに適用することで、さらに多くの疾患原因・創薬標的となるゲノム変異を発見できる可能性があります(図1)。
本研究成果は、「Nature Communications」に掲載されました(2025年1月9日オンライン公開)。また、今回収集したスプライスサイト生成変異は、研究グループが新規に開発したポータルサイト、SSCV DB (https://sscvdb.io)で公開されています。
図1:今回開発したプラットフォームの概念図。このプラットフォームをさらに発展させることで、新たなデータの登録に応じて自動的に解析を行い、創薬ターゲットとなるゲノム変異を自律的に蓄積するシステムの実現が期待されます。
背景
ハイスループットシークエンス技術注4の革新、またその有効性が広く認められたことで、オミクスデータ注5の蓄積が国際的に加速しています。これらのデータを集積したレポジトリを活用し、世界中の研究者が新たな視点でシークエンスデータの再解析を行うことで、データの潜在的な価値を最大限に引き出そうとする取り組みが世界中で進められています。その中でも特に重要なデータレポジトリの一つが「Sequence Read Archive (SRA)」注6です。SRAは、主に次世代シークエンサーにより生成されたゲノム、トランスクリプトームなどの膨大な配列データを蓄積したもので、公開されている多くのデータは誰でも自由に利用可能です。18か月ごとに収録データが倍増しており、今後もさらなる拡大が見込まれています。
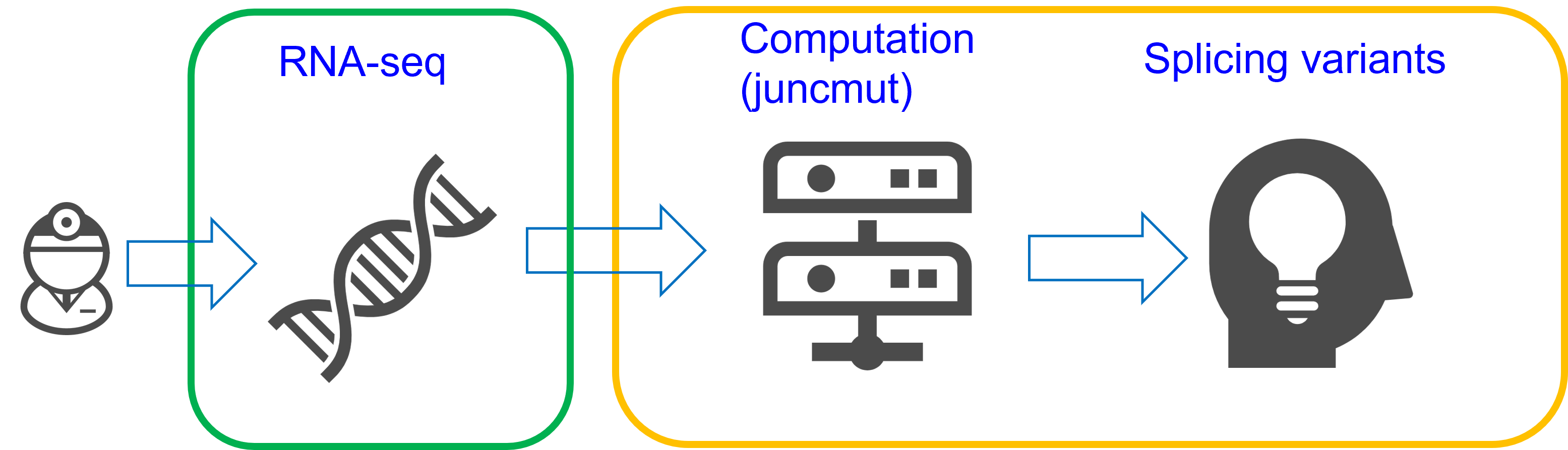
研究グループはこれまでに、SRAに登録されたトランスクリプトームシーケンスデータを活用して、スプライシング異常注7を介した疾患関連のゲノム変異を効率的に同定するためのプラットフォームを開発してきました。過去の研究では、イントロン残存注8というスプライシング異常を引き起こすゲノム変異に注目しました(Shiraishi et al., Nature Communications, 2022; doi.org/10.1038/s41467-022-32887-9)。一方で、本研究では、スプライシング関連変異の中でも特に重要とされる「スプライスサイト生成変異」に焦点を当てています(図2)。 スプライスサイト生成変異はゲノム変異により新たなスプライスサイトが形成されることで、異常なスプライシングパターンを引き起こします。スプライスサイト生成変異は次のような特徴を持ちます。
- スプライスサイト生成変異を同定することは非常に難しく、疾患ゲノム解析の中で多くの場合見逃されてきました。しかし、これらの変異をデータベース化することで、これまで解明されていなかった疾患の原因となる遺伝子変異を発見できる可能性が広がります。
- スプライスサイト生成変異は、核酸医薬を用いた治療の有望なターゲットとして注目されています。このデータベースを構築することで、希少疾患を抱える患者に対する個別化治療の進展が期待されます(Kim et al., Nature, 2023; doi.org/10.1038/s41586-023-06277-0 など)。
図2:スプライスサイト生成変異の例.イントロン領域中の変異によりスプライスサイトが新規に形成され、新しいエキソン(偽エキソン)が転写配列に挿入されます(左図)。
この変異に対する核酸医薬を投与することで、スプライス因子が阻害され、正常なスプライシングが回復します(右図)。
研究成果
スプライシング異常を引き起こすゲノム変異の同定には、ゲノムデータとトランスクリプトームデータの両方が必要とされてきました。しかし、このようなペアデータが存在しているサンプルは非常に限られています。そこで私たちは、Sequence Read Archive (SRA)が提供する膨大なトランスクリプトームシークエンスデータを最大限に活用するため、ゲノムデータなしでトランスクリプトームシークエンスデータのみを用いてスプライスサイト生成変異を同定するアルゴリズム「juncmut」を開発しました(図3)。
研究グループは、国立遺伝学研究所のスーパーコンピューターを用い、322,072件のトランスクリプトームシークエンスデータをjuncmutで再解析しました。その結果、合計30,130件のスプライスサイト生成変異を同定し、それらをカタログ化することに成功しました。収集した変異リストは新たに開発されたポータルサイト、SSCV DB (https://sscvdb.io)に公開されています。
図3:スプライスサイト生成変異周辺のトランスクリプトームシークエンスのアラインメント図。データ中からスプライスサイト生成変異を観測できるという現象に着目することで、juncmutのアルゴリズム開発へと繋がりました。

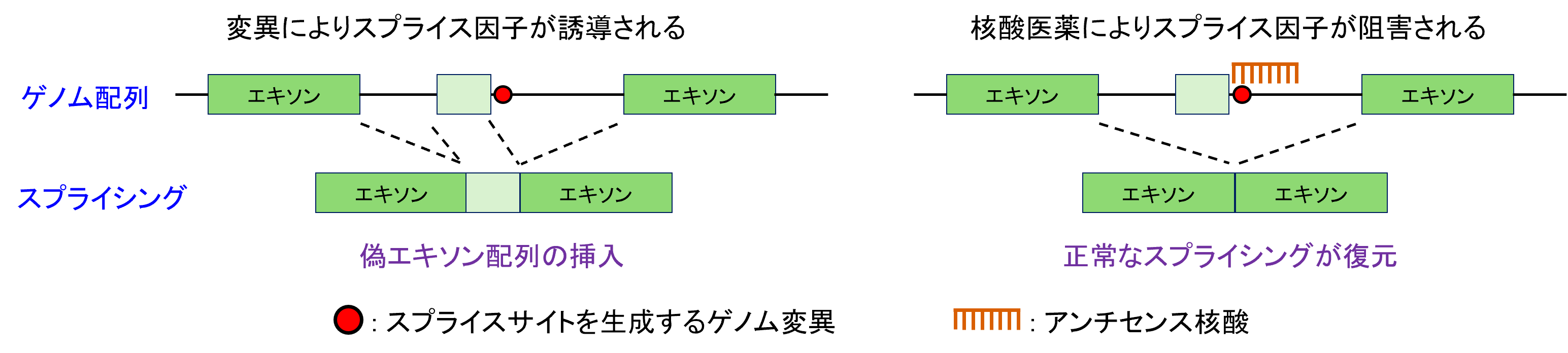
図4: Alu配列上で、スプライスサイト生成により「新規エキソン」形成領域の頻度を示した図。エキソンの始点(アクセプターサイト)と終点(ドナーサイト)には複数のホットスポットがあります。また、「新規エキソン」はこの組み合わせで形成されます。
この大規模な変異カタログを活用し、研究グループはスプライスサイト生成変異に関する様々な性質や現象を調査しました。その一例として、「Aluエキソン化」に関する特性を詳細に解析しました。Alu配列は約300bpの配列で、元来エキソンを形成しやすい性質を有しています。イントロン領域に挿入されたAlu配列は、さらにスプライスサイト生成変異を獲得することにより、新しいエキソンを形成します。この現象は「Aluエキソン化」と呼ばれ、霊長類のゲノム進化の文脈で研究されてきました(図4)。近年では、希少疾患やがんの進展においても、同様の現象が生じていることが明らかになってきました。研究グループは、Alu配列上にある3,102件のスプライスサイト生成変異を調査し、「偽エキソン」が特に形成されやすい領域を特定しました。 特に最も「偽エキソン」が形成されやすいのは、Alu配列の157番目から始まり280番目で終わる領域でした。
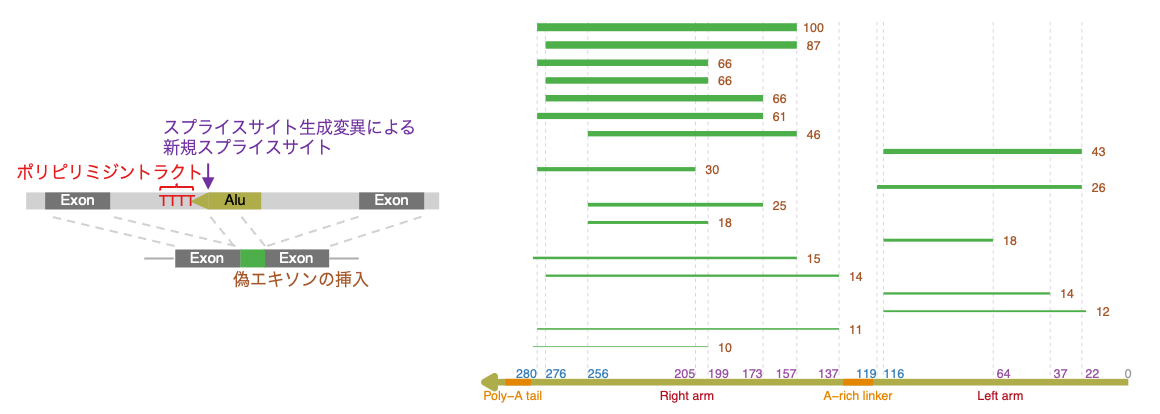
図5:NOTCH1のエキソン28の上流132bpで検出されたスプライスサイト生成変異(左上)。これにより、トランスクリプトの129bpの延長、juxtamembrane領域の43アミノ酸の挿入により、NOTCH1のリガンド非依存的活性化が引き起こされます(右上)。また、この変異に特異的な配列の核酸医薬を設計し、ゲノム編集モデルに投与することで、スプライシング異常が正常に戻り、活性化が抑制されることを確認しました。
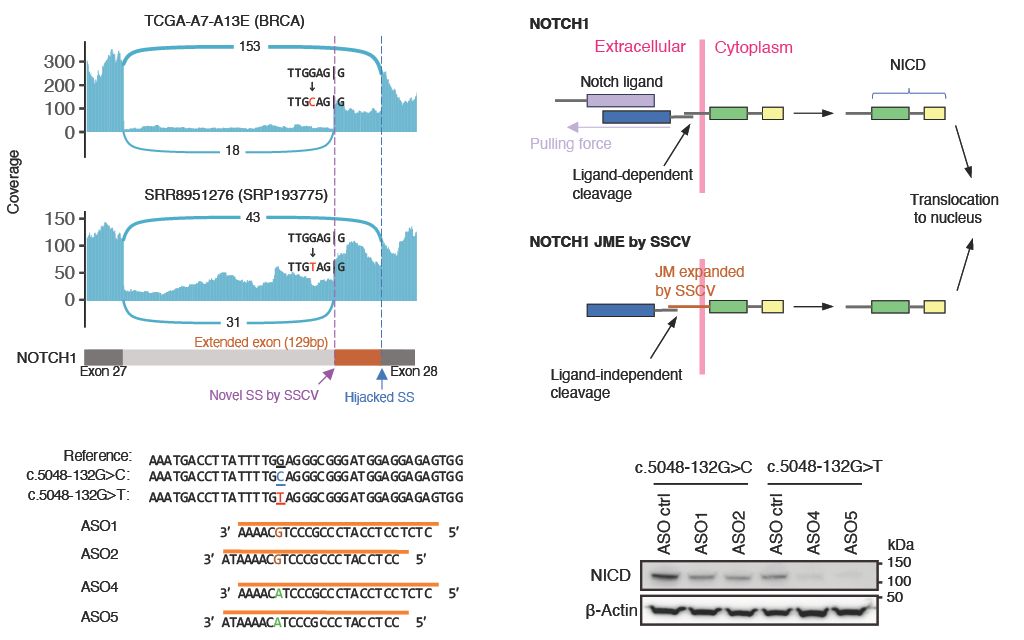
また、得られたスプライスサイト生成変異の中で、がんの進展に関与すると考えられるNOTCH1遺伝子の変異に注目しました。この変異は、NOTCH1遺伝子のexon 28から132bp上流に位置するゲノム変異であり、これによってトランスクリプトが129bp延長され、juxtamembrane領域に43アミノ酸の挿入を引き起こします(図5)。この変異により、NOTCH1がリガンド非依存的注9に活性化されることが予想されました。研究グループは、この変異を再現するCRIPSRゲノム編集細胞モデルを開発し、NOTCH1の顕著な活性化を確認しました。さらに、この変異により形成されるスプライスサイトを標的とする核酸医薬を設計して投与することで、この異常な活性化を効果的に抑制できることを実証しました。
展望
本研究では疾患に関連している遺伝子上に計5,121個のスプライスサイト生成変異が同定されており、これらのほとんどはこれまでに検出されていない、または疾患に関連が知られていなかった変異です。この中には、本研究で詳細に検証を加えたNOTCH1遺伝子の変異の他にも、さらに多くの疾患に関連した変異、創薬標的となる変異が含まれていることが予想されます。
また、Sequence Read Archiveなどのデータベースにおいては、現在進行形で新たなデータの蓄積が進んでいます。今後加速度的に蓄積が進むトランスクリプトームデータに対して、本研究で開発した情報解析プラットフォームを適用することで、さらに多くの新規疾患関連・創薬の標的となる変異を自律的に同定する仕組みを構築することが期待できます。
本研究により、スプライスサイト生成変異は多数検出できるようになりましたが、その一つ一つが実際に疾患に関連しているか、創薬のターゲットになるかについては、専門家が個別に情報を検証し解釈をするが必要があります。今後、生成AIを活用し、アノテーションシステム注10の精緻化を図ることで、疾患や創薬に重要な変異を効率的に特定するアプローチの開発を計画しています。
発表論文
雑誌名 Nature Communications
タイトル Systematically developing a registry of splice-site creating variants utilizing massive publicly available transcriptome sequence data
著者 Naoko Iida†, Ai Okada†, Yoshihisa Kobayashi†, Kenichi Chiba, Yasushi Yatabe, Yuichi Shiraishi* (†Equally contributed, *Corresponding author)
掲載日 2025年1月9日
DOI 10.1038/s41467-024-55185-y
URL https://www.nature.com/articles/s41467-024-55185-y
研究費
- 国立研究開発法人日本医療研究開発機構(AMED)革新的がん医療実用化研究事業「ゲノム医療と創薬に資するスプライスサイト生成変異の探索基盤の開発」(研究代表者名:白石 友一)
- 日本学術振興会 科学研究費助成事業 基盤研究(B)「大規模トランスクリプトームからの自律的知能獲得システム基盤の開発」(研究代表者名:白石 友一)
- 科学技術振興機構 創発的研究支援事業「薬剤耐性から迫る発がん機構」(研究代表者名:小林 祥久)
- 国立研究開発法人日本医療研究開発機構(AMED) 創薬支援推進事業・創薬総合支援事業「アンチセンスオリゴヌクレオチドを用いた新規がん治療標的の検証」(研究代表者名:小林 祥久)
- 日本学術振興会 科学研究費助成事業 基盤研究(B)「薬剤耐性と核酸医薬治療(研究代表者名:小林 祥久)
用語解説
注1 スプライスサイト生成変異
遺伝子内に新たなスプライス部位(イントロンとエクソンの境界)が生成されることで、正常なスプライシングが妨げられ、異常なmRNAが生成されるDNA配列の変異を指す。
注2 トランスクリプトーム
細胞中に存在している全てのmRNAの総体。
注3 CRISPR
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)は、特定のDNA配列を迅速かつ精密に編集する技術で、細菌の免疫システムを基に開発された。ガイドRNAとCasタンパク質を利用して標的DNAを切断し、細胞の修復機構を通じて配列を改変する。
注4 ハイスループットシークエンス技術
大量のDNAまたはRNA配列データを迅速かつ効率的に取得する手法で、次世代シークエンシング(NGS, Next-Generation Sequencing)とも呼ばれる。Illumina社のプラットフォームが世界的に最も広く使用されているが、近年ではロングリードシークエンシングが可能なプラットフォーム(Pacific Biosciences社やOxford Nanopore Technologies社が提供)の利用も拡大している。
注5 オミクスデータ
ゲノム、トランスクリプトーム、プロテオーム、エピゲノム、メタボロームなどの網羅的な生体分子の情報を計測したデータのことを示している。
注6 Sequence Read Archive (SRA)
次世代シークエンサーで生成されたショートリードを格納しているデータレポジトリ。米国のNCBI (National Center for Biotechnology Information)、英国のEBI(European Bioinformatics Institute), 日本のDDBJ センター(DNA Data Bank of Japan)が共同で管理している。大部分のデータは、アクセス制限なしに、自由にダウンロードが可能である。
注7 スプライシング異常
mRNAの前駆体は、「スプライシング」の過程において遺伝子配列から不要な部分であるイントロン領域が取り除かれ、成熟したmRNAとなる。このプロセスに異常が生じることをスプライシング異常という。この結果として、正常とは異なるタンパク質が生成されることや、タンパク質の発現量の低下が引き起こされる。
注8 イントロン残存
スプライシング異常の中でも、特にイントロン領域が適切に取り除かれずに残存してしまう現象のことである。
注9 リガンド非依存的
通常、受容体や分子はリガンド(特定の結合分子や刺激因子)が結合することで活性化し、シグナル伝達を開始する。しかし、何らかの原因でリガンドが存在しなくても受容体や分子が活性化または機能する状態を「リガンド非依存的」と呼ぶ。
注10 アノテーションシステム
検出されたゲノム変異の生物学的意義や病的影響を解釈するためのシステムを指す。一般的に、変異がエクソン、イントロン、プロモーターなどゲノム内のどの領域に位置するかを特定する。コーディング領域ではアミノ酸置換や終止コドン生成などの影響を評価し、非コーディング領域では調節機能やスプライス部位への影響を検討する。また、既知の病的変異や一般的な変異に関する各種データベースとの照合を行い、変異の意義を解釈する。疾患の原因解明や診断、治療標的の特定に重要な役割を果たす。
問い合わせ先
研究について
国立研究開発法人国立がん研究センター
研究所 ゲノム解析基盤開発分野 分野長 白石 友一
広報窓口
国立研究開発法人国立がん研究センター
企画戦略局 広報企画室