RA症例間で症状は似ているが、治療に対する反応は大きく異なることが判明。 Though symptoms are similar between RA cases, response to therapy varies widely; findings could help lead to more effective, individualized therapies
2022-10-24 カリフォルニア大学サンディエゴ校(UCSD)
RAに関与する同じ分子が、異なる患者から採取した細胞では正反対の機能を持つことが明らかになった。
RA の原因およびリスクファクターは多岐にわたり、十分に解明されていない。年齢、性別、ライフスタイル(喫煙など)から肥満、遺伝的特徴まで多岐にわたる。RAを完治させる方法はありませんが、病気の進行を遅らせ、関節の変形を防ぎ、痛みや障害を軽減するための幅広い標的治療法がある。
研究チームは、関節の滑膜(関節を潤滑に動かし、関節の摩耗を最小限に抑える柔らかい結合組織内に存在する特殊な細胞である線維芽細胞様滑膜細胞(FLS)に注目した。FLSは、RAの関節破壊に大きな役割を果たしている。
研究チームは、培養した初代FLSを調べ、個々のRA患者の細胞株で関与する特定の転写因子(遺伝子の転写やコピーを制御するタンパク質)を特定した。この解析により、これらの細胞株は、炎症に関与する活性化経路が異なる少なくとも2つのサブタイプに層別化されることが明らかになった。
<関連情報>
- https://today.ucsd.edu/story/novel-study-identifies-key-molecular-players-in-rheumatoid-arthritis
- https://www.nature.com/articles/s41467-022-33785-w
関節リウマチ線維芽細胞様滑膜細胞のシステム生物学的解析による細胞株特異的な転写因子機能への関与 Systems-biology analysis of rheumatoid arthritis fibroblast-like synoviocytes implicates cell line-specific transcription factor function
Richard I. Ainsworth,Deepa Hammaker,Gyrid Nygaard,Cecilia Ansalone,Camilla Machado,Kai Zhang,Lina Zheng,Lucy Carrillo,Andre Wildberg,Amanda Kuhs,Mattias N. D. Svensson,David L. Boyle,Gary S. Firestein &Wei Wang
Nature Communications Published:20 October 2022
DOI:https://doi.org/10.1038/s41467-022-33785-w
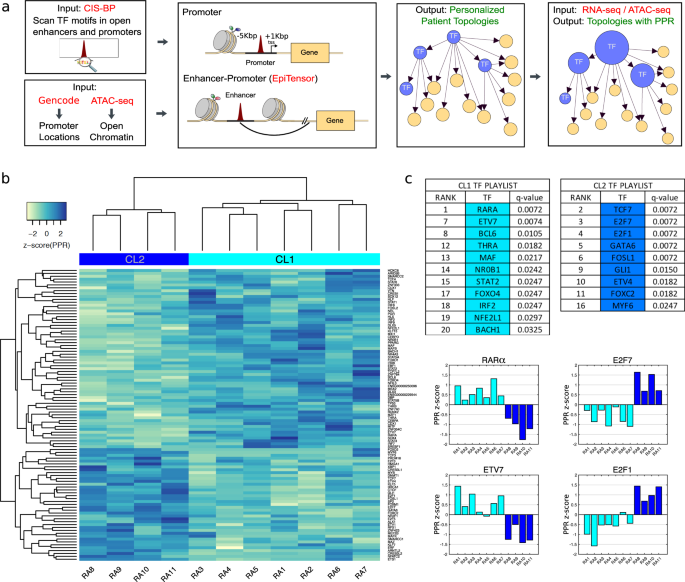
Abstract
Rheumatoid arthritis (RA) is an immune-mediated disease affecting diarthrodial joints that remains an unmet medical need despite improved therapy. This limitation likely reflects the diversity of pathogenic pathways in RA, with individual patients demonstrating variable responses to targeted therapies. Better understanding of RA pathogenesis would be aided by a more complete characterization of the disease. To tackle this challenge, we develop and apply a systems biology approach to identify important transcription factors (TFs) in individual RA fibroblast-like synoviocyte (FLS) cell lines by integrating transcriptomic and epigenomic information. Based on the relative importance of the identified TFs, we stratify the RA FLS cell lines into two subtypes with distinct phenotypes and predicted active pathways. We biologically validate these predictions for the top subtype-specific TF RARα and demonstrate differential regulation of TGFβ signaling in the two subtypes. This study characterizes clusters of RA cell lines with distinctive TF biology by integrating transcriptomic and epigenomic data, which could pave the way towards a greater understanding of disease heterogeneity.


