2024-05-10 海洋研究開発機構,東京大学,理化学研究所
1. 発表のポイント
- 深海堆積物のメタゲノム解析によって、極めて新規な糖質分解酵素(β-N-アセチルガラクトサミニダーゼ)を発見しました。さらに得られた知見を元に公共タンパク質データベースの大規模な再解析を行った結果、4つもの新規酵素グループを発見しました。
- 新規酵素グループの詳細な解析を行い、これらの酵素が多様な機能を獲得してきた進化の過程を紐解きました。医療分野等への産業応用を目指した酵素の分子設計やβ-N-アセチルガラクトサミン含有糖鎖の生物学的機能の解明などへの波及が期待される成果です。
- 深海環境の微生物は、そのサンプリングの難しさのために研究例が非常に限定的です。本研究は、深海微生物が未知の有用遺伝子を探索する上で有望な遺伝資源であることを実証した好例です。
2. 概要
国立研究開発法人海洋研究開発機構(理事長 大和 裕幸、以下「JAMSTEC」という。)海洋機能利用部門 生命理工学センターの澄田智美副主任研究員、平岡聡史研究員らは、東京大学の伏信進矢教授、理化学研究所 開拓研究本部の石渡明弘専任研究員ら、横浜市立大学、西オーストラリア大学と共同で、深海に生息する微生物群集のメタゲノム情報とそこから得られた手がかりをもとに、β-N-アセチルガラクトサミン(β-GalNAc)含有糖鎖に作用する4つの新規酵素グループを発見し、その酵素グループの詳細な機能解析と分子進化のメカニズムを解明しました。
本研究では新規酵素探索の対象として、陸上とはかけ離れた深海環境に生息する微生物叢に着目しました。深海の極限環境に適応した微生物は、地上に生息する微生物とは全く異なる独自の特徴を持っており、深海微生物叢のメタゲノム情報は、未知の性質を持つ新規酵素の探索対象として有望な遺伝子資源です。しかし、深海からの微生物サンプリングには技術的な困難が伴うため、過去の研究例は世界的にも限られていました。研究グループは、JAMSTECが採取した深海堆積物を対象に、メタゲノム解析とバイオインフォマティクス解析によってβ-GalNAc含有糖鎖の分解酵素β-N-アセチルガラクトサミニダーゼを探索しました。
その結果、既知の酵素とはアミノ酸配列や基質特異性が大きく異なる新規酵素を発見しました。次に新規酵素に特徴的な配列情報をもとに、公共のタンパク質データベースへ探索範囲を広げたところ、細菌・古細菌・真核生物に跨って普遍的に存在する4つの新規酵素グループを発見しました。さらに、これら一連の新規酵素グループの機能とタンパク質の立体構造を解析し、既知の酵素を含む5つの酵素グループが進化の過程でどのように多様な機能を獲得してきたかを解明しました。本研究成果は、β-GalNAc含有糖鎖の生物学的機能の解明はもとより、将来的には新機能を持つ酵素の人工的な設計や、新規酵素を利用した産業応用への展開が期待されます。
本成果は、「Nature Communications」に5月10日付け(日本時間)で掲載されました(オープンアクセス)。なお、本研究の一部は科学研究費助成事業(JP20K15444、JP22K05398)、創薬等先端技術支援基盤プラットフォーム(BINDS)の支援(課題番号JP22ama121001(支援番号3118)、および公益財団法人水谷糖質科学振興財団の研究助成によって実施されました。
論文情報
タイトル
Genetic and functional diversity of β-N-acetylgalactosamine-targeting glycosidases expanded by deep-sea metagenome analysis
著者
澄田智美1、 平岡聡史1、 臼井けい子1、 石渡明弘2、 仙石徹3、 Keith A. Stubbs4、 田中克典2,5、 出口茂1、 伏信進矢6、 布浦拓郎1
所属
1. 国立研究開発法人海洋研究開発機構
2. 理化学研究所
3. 横浜市立大学
4. 西オーストラリア大学
5. 東京工業大学
6. 東京大学
DOI
10.1038/s41467-024-47653-2
論文公開日
2024年5月10日(日本時間)
3. 背景
地球上のあらゆる生物は、細胞内外の多様な複合糖質(多糖、糖脂質、糖タンパク質など)を糖質関連酵素(糖転移酵素や糖質分解酵素など)を用いて合成・分解することで、様々な生命現象を駆動しています。しかし、複合糖質の多様性に対して、糖質関連酵素の機能解析は圧倒的に不足しています。新しい機能を持つ糖質関連酵素を探索し、その諸性質を解明することは、その酵素を介した生命現象の解明に重要です。また、糖質関連酵素は機能性オリゴ糖の生産、糖鎖構造解析、疾患診断への利用など、食品・医療をはじめとする様々な産業分野でも利用されています。そのため、新しい糖質関連酵素の探索は基礎科学と応用科学の両面で重要です。
β-N-アセチルガラクトサミン(β-GalNAc)は複合糖質を構成する糖の一種で、β-GalNAcを含有する複合糖質も自然界に広く分布しており、細菌・古細菌・真核生物において、細胞間認識や接着・感染などの相互作⽤、情報伝達、免疫応答などの様々な生物学的プロセスに重要な役割を果たしています。β-GalNAc含有糖鎖を分解することで機能調整に関わる酵素としてβ-N-アセチルガラクトサミニダーゼ(β-NGA)が知られていますが、今日まで、陸上環境(土壌とヒト腸内)で細菌由来の3種の酵素しか研究されていませんでした。しかし、β-GalNAc含有糖鎖の多様性や存在量、さらにはそれらの糖鎖が関与する生命現象の重要性を考えると、自然界には未知のβ-NGAが多数存在していると考えるのが妥当です。
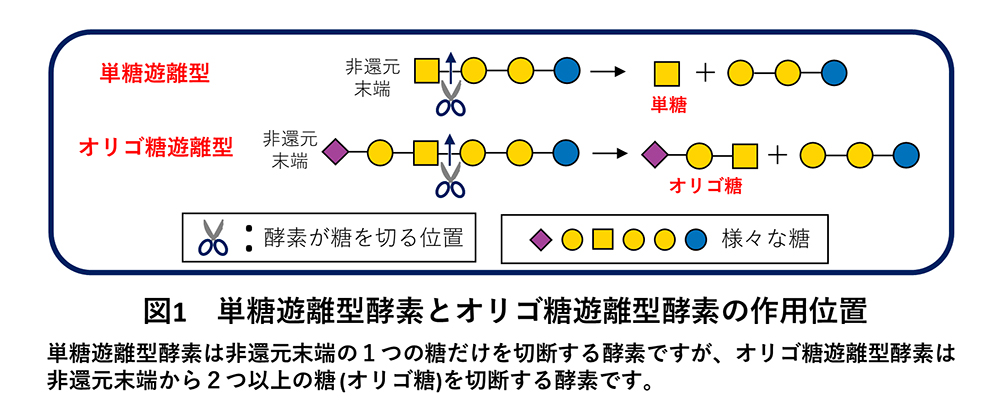
既報の酵素はすべて、糖質関連酵素をアミノ酸配列に基づき分類しているデータベース(Carbohydrate-Active enZymes Database; CAZy database)において、糖質分解酵素ファミリー 123(GH123; Glycoside Hydrolase Family 123)に分類されています。GH123に分類されるβ-NGAは、機能的にもβ-GalNAc含有糖鎖の非還元末端の単糖だけを切断する単糖遊離型酵素しか登録されておらず、糖鎖の内部を切断するオリゴ糖遊離型酵素は見つかっていません(図1)。β-GalNAc含有糖鎖は陸上環境だけではなく海洋環境においても、例えば微生物の細胞外多糖や、魚介類の不可食部に多く含まれるコンドロイチン硫酸に含まれています。しかしながら、海洋環境におけるβ-NGAを介した生命現象は今日まで研究対象とされてきませんでした。よって海洋環境から新しい機能を持つβ-NGAを探索し機能を同定することは、自然界におけるβ-GalNAcを介した生命現象を包括的に理解するためにも重要となります。そこで本研究では、深海微生物叢のメタゲノム情報を用いて、β-GalNAcに作用する新規糖質分解酵素を探索しました。
4. 成果
本研究では、深海調査研究船「かいれい」(2022年2月に退役)と大深度小型無人探査機「ABISMO」(2017年5月に喪失)を用いて伊豆小笠原海溝水深5,747 mで採取された深海堆積物を対象にメタゲノム解析※1 を行い、得られた配列情報からβ-NGA遺伝子を探索するバイオインフォマティクス解析を実施しました。
その結果、既報の単糖遊離型β-NGAとはアミノ酸配列が大きく異なる新規酵素の候補配列が得られました。この配列由来の酵素を作製して活性測定を行った結果、既報のGH123の酵素が示す単糖遊離型の活性(図2a)だけではなく、糖鎖の内部のβ-GalNAcにも作用できるオリゴ糖遊離型の活性も示されました(図2b)。これまでオリゴ糖遊離型のβ-NGAは発見されていないため、これは深海メタゲノム情報から新規機能酵素(オリゴ糖/単糖遊離型のβ-NGA)を発見した成果になりました。
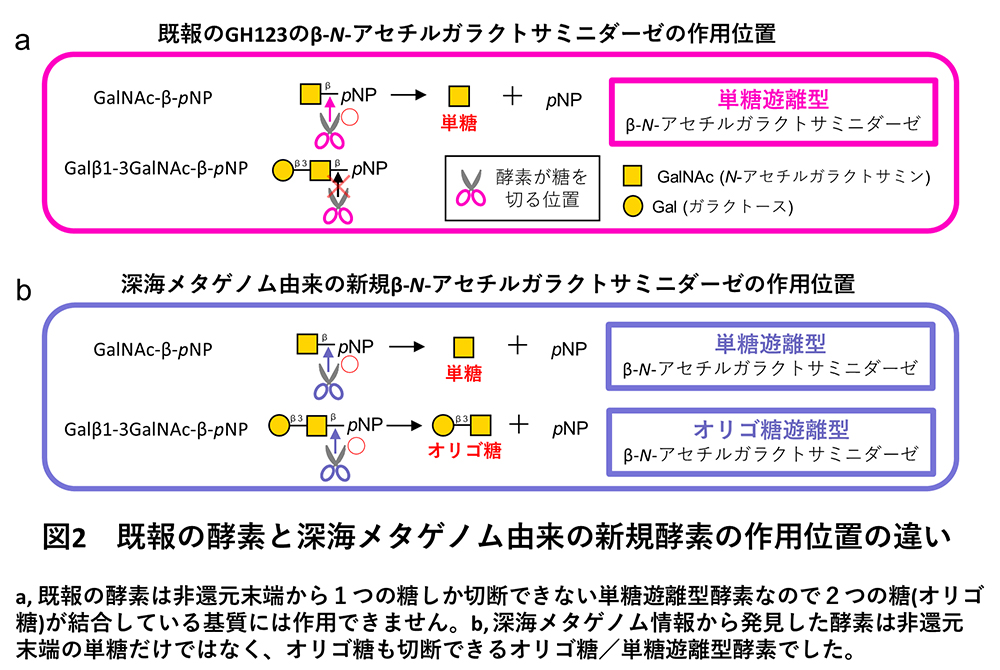
新規酵素と既報酵素のアミノ酸配列は、全体的に大きく異なっていたものの、両者で弱く共通している部分領域として、約70アミノ酸からなる機能未知領域(DUF4091; Domain of unknown function 4091)を見出しました。このDUF4091を手がかりに公共のタンパク質ファミリーデータベースであるPfamから新規酵素候補配列を探索したところ、細菌・古細菌・真核生物のすべての生物から多数の配列が取得されました。このことは既報のGH123酵素グループは主に細菌由来の配列のみから構成されていることと対照的でした。
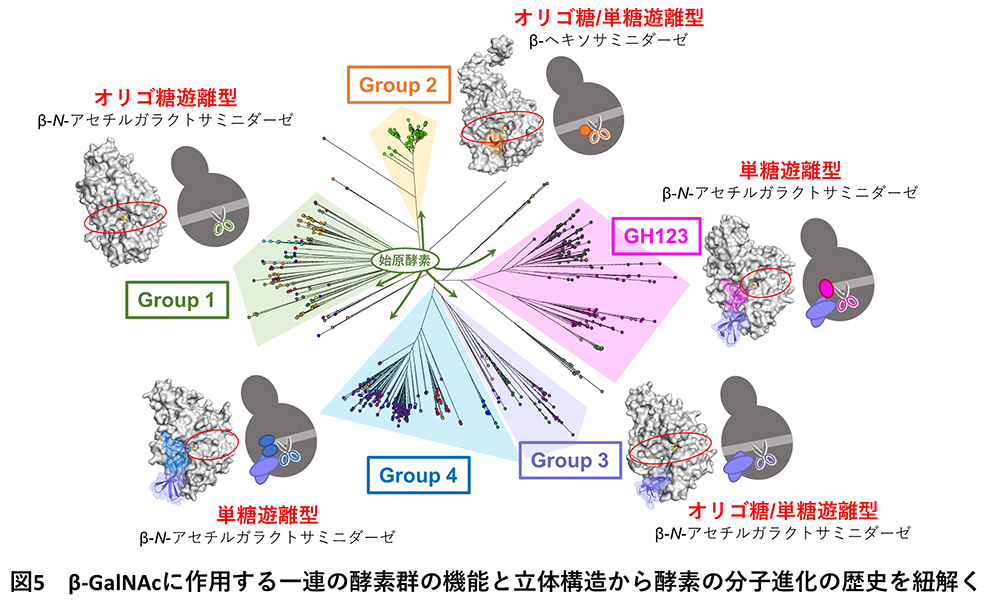
得られた配列740個を用いた大規模な系統解析から、DUF4091を持つ配列は、既報の酵素グループも含め、大きく5系統(Group 1〜4とGH123)に分かれることが分かりました(図3a)。各グループからいくつかの配列を選択し酵素活性測定を行ったところ、各グループの機能に明確な違いがみられました。そこで、X線結晶構造解析※2 という手法を用いて酵素の立体形状を調べることで、酵素の機能と立体構造を統合的に分析しました(図3b〜f)。立体構造解析に必要なデータ収集は、大型放射光施設SPring-8のBL32XU※3 にて、全自動データ収集システムZOO※4 と自動パイプラインNABE※5 を利用して行いました。各グループの酵素の機能と立体構造を比較したところ、Group 1の酵素は単糖を遊離せず、オリゴ糖のみ遊離する厳密なオリゴ糖遊離型β-NGAで、5系統の中で一番シンプルな形をしていました(図3b)。Group 1の酵素と比較して、Group 2〜4とGH123の酵素には、酵素が糖を切る位置(図3b〜fのはさみで示した部分)の周辺に様々な付加構造や付加ループが存在していました(図3c〜fの各色で示した部分)。Group 2の酵素はオリゴ糖も単糖も遊離することができ、またβ-GalNAcだけではなくβ-GalNAcの立体異性体であるβ-N-アセチルグルコサミンも遊離することのできる、オリゴ糖/単糖遊離型β-ヘキソサミニダーゼであることが判明しました(図3c)。Group 3の酵素はオリゴ糖と単糖の両方を遊離するオリゴ糖/単糖遊離型β-NGAでした(図3d)。深海メタゲノム情報から見出された酵素も、このGroup 3に含まれていました。Group 4の酵素は、単糖のみを遊離しオリゴ糖を遊離することのできない、厳密な単糖遊離型β-NGAでした(図3e)。既報のGH123酵素も、Group 4と同様に単糖遊離型β-NGAです(図3f)。
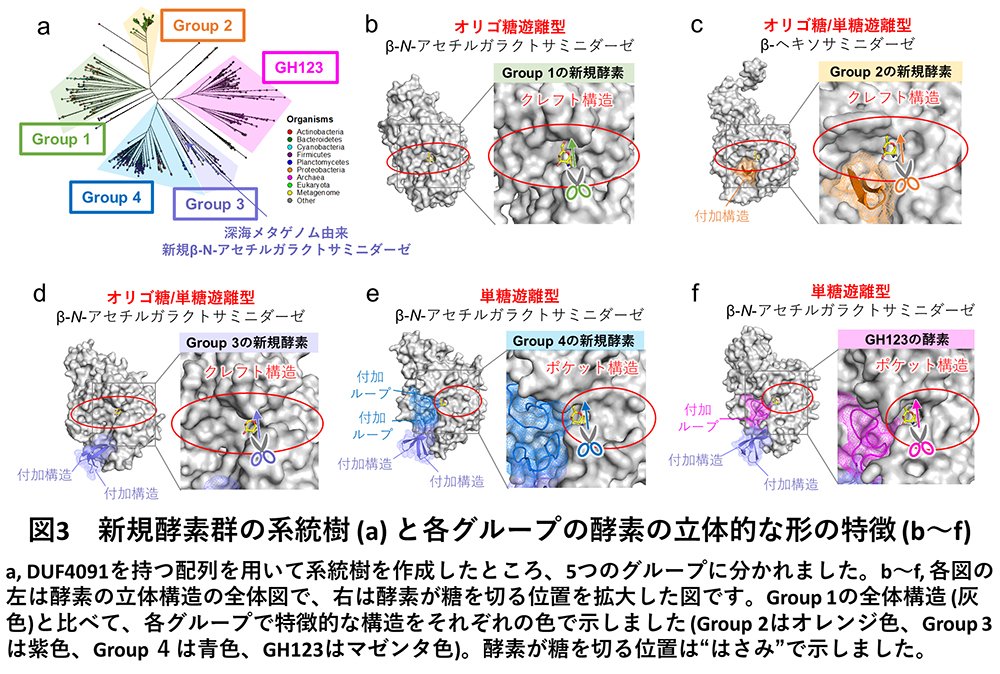
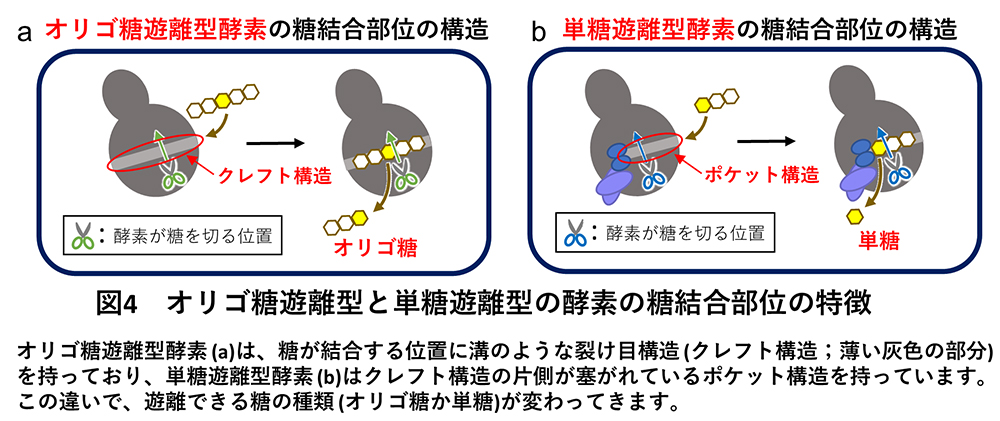
このような酵素グループ間の機能の違いは、酵素のタンパク質の立体形状から説明することができました(図3、図4)。すなわち、Group 1、2、3のオリゴ糖遊離型酵素は、酵素の表面に溝のような裂け目構造(クレフト構造)を持っているため(図4a)、長いオリゴ糖が結合して糖鎖内部の標的の糖(図4の黄色で示している糖)を切断してオリゴ糖を遊離できました(図4a)。一方でGroup 4、5ではクレフト構造の片側(図4bの薄い灰色の部分(クレフト)の左側)が付加ループ(図4b、青色で示している構造)で塞がれており、糖鎖の末端しか入り込めないポケット構造になっているため、末端の単糖のみを切断することのできる酵素でした(図4b)。これらのオリゴ糖遊離型酵素と単糖遊離型酵素の糖結合部位の違いは、たった10数アミノ酸の配列の挿入により引き起こされていました。このように酵素の機能(基質特異性)は大きく異なっていましたが、これらの5系統の酵素グループは全て同じ触媒反応機構で糖を分解していることがNMR(核磁気共鳴)※6 解析から分かりました。
一連の酵素グループの機能と立体構造を比較することで、β-GalNAcに作用する酵素の機能が多様化してきた分子進化の歴史を予想しました(図5)。β-GalNAcに作用する酵素の始原酵素はGroup 1に属し、遺伝子配列の変異が蓄積することで、現在では5系統に分岐したことが考えられます。さらに、遺伝子の変異に加えて、遺伝子の挿入が起こった結果、この酵素の機能が、オリゴ糖遊離型酵素、オリゴ糖/単糖遊離型酵素、単糖遊離型酵素と多様化した可能性が考えられました(図5)。
用語解説
※1 メタゲノム解析
多様な原核生物種を含む細菌叢から直接DNAを抽出し、ゲノムシーケンシングと情報解析を行うことによって、分離培養を経ることなくゲノム解析を行う手法。
※2 X線結晶構造解析
タンパク質などの生体分子の立体構造を決定するための手法の一つ。対象とする分子の結晶を作製し、その結晶にX線を照射して得られる回折データを解析することで、タンパク質の立体構造を原子レベルで詳細に解析する方法です。
※3 大型放射光施設「SPring-8」およびビームライン「BL32XU」
SPring-8は兵庫県の播磨科学公園都市にある世界最高性能の放射光を生み出すことができる大型放射光施設で、名前はSuper Photon ring-8 GeV(80億電子ボルト)に由来しています。国内外の産学官の研究者等に開かれた共同利用施設です。BL32XUは結晶サイズ10 μm以下の微小結晶からの回折強度データ収集、構造解析を目標として設計された、微小結晶構造解析が実現できるビームラインです。
※4 ZOOシステム
タンパク質結晶からX線結晶構造解析に必要な高品質のデータを自動で収集するシステム。このシステムにより放射光施設を利用したタンパク質の高分解能データ収集が無人自動で誰でも簡単にできるようになりました。
※5 NABEシステム
ZOOを利用して収集された大量の回折データに対して、簡易的な構造解析を自動で実施し結果を閲覧・比較できるシステム。ユーザーが指定した位置周辺(化合物結合部位など)の電子密度マップとモデルとを、データごとに表示して構造比較することが可能です。例えば、化合物導入の可否などの結果を迅速に判別できるため、目的に応じた最良データを効率よく選択できるようになりました。
※6 NMR(Nuclear magnetic resonance; 核磁気共鳴)解析
静磁場に置かれた原子核の共鳴を観測し、分子の構造や運動状態などの性質を調べる分光学的解析法。溶媒に分子を溶解させて計測する溶液NMR解析や固体状態の分子を計測する固体NMR解析などがあり、幅広い状態の試料を計測し解析することができます。
5. 今後の展望
本研究で得られた一連の新規酵素グループの機能・構造解析の結果は、β-GalNAc含有糖鎖の生物学的機能の解明や産業応用展開を目指した酵素デザインなどの、今後の研究に役立つものです。また本研究は、陸上とは異なる極限環境である深海のメタゲノム情報が、新規酵素の探索に有用であることを示した成果です。
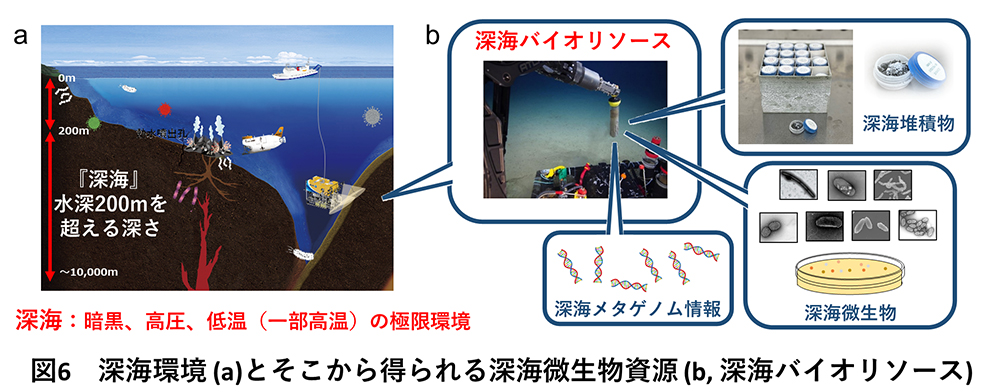
地球表面の約70%を占めている海洋の平均深度は3,700 mですが、そのうち水深200 m以深を深海と呼びます(図6a)。深海は暗黒・高圧・低温の極限環境でありながら、実に多様な深海生物が生存し生態系を営んでいます。中でも微生物は、進化の過程で深海環境に適応した独自の生存戦略を作り出しています。そのため深海微生物は、陸上とは大きく異なる未知のタンパク質が数多く眠ることが期待されており、新規機能や新規配列を持つ酵素を探索する上で魅⼒的な微生物資源(バイオリソース)です。JAMSTECでは現在、深海微生物の産業利用の促進を目指し、JAMSTECが保有する深海バイオリソース(図6b, 深海堆積物と深海微生物)を、外部機関に提供する深海バイオリソース提供事業を行っています(https://www.jamstec.go.jp/cebn/bioresource/j/)。また、深海環境では、陸上環境とは明確に異なる微生物群集構造が観察され、微生物の遺伝子構造も大きく異なることが知られています。深海微生物叢のメタゲノム情報には、深海環境に固有の微⽣物や、培養することの難しい未培養・難培養系統の細菌情報も多く含まれているため、深海メタゲノム情報は有用な遺伝⼦資源としての可能性が秘められています。
今回の研究から得られた成果をもとに、今後産業利用に向けて株式会社 糖鎖工学研究所(GlyTech, Inc)と共同して研究を実施し、新規酵素の有用性を実証していく予定です。また、今回の研究では1地点の堆積物コアを用いたメタゲノム解析から機能酵素の探索を行いましたが、今後は様々な海域・深度のメタゲノム情報を大規模に活用することで、産業利用に向けた応用が期待される、より多様な機能酵素の探索に取り組んでいきます。
本研究のお問い合わせ先
国立研究開発法人海洋研究開発機構
海洋機能利用部門生命理工学センター
副主任研究員 澄田 智美
報道担当
国立研究開発法人海洋研究開発機構
海洋科学技術戦略部 報道室
東京大学大学院農学生命科学研究科・農学部
事務部 総務課総務チーム 総務・広報情報担当(広報情報担当)
国立研究開発法人理化学研究所
広報室 報道担当