2025-01-31 東京大学医科学研究所
発表のポイント
- 空間トランスクリプトーム解析(Spatial Transcriptomics, ST)において、遺伝子発現・空間情報・組織学的画像といった多次元データを統合するための新規深層学習モデル「STAIG」を開発した。
- STAIGは、既存手法と異なり、組織切片間での座標整合や追加的な手動操作なしに、画像情報を活用してバッチ効果を低減し、空間ドメインを高精度に同定することが可能である。
- 脳やがん組織など多種多様な実データセットにおいて、STAIGは既存手法を上回る空間区画化能力を示し、がん微小環境の新たな特徴を明らかにするなど、生物学的知見の深化への寄与が期待できる。
ST技術の概略
概要
東京大学医科学研究所機能解析イン・シリコ分野の中井謙太教授による研究グループは、空間トランスクリプトーム解析において、遺伝子発現・空間座標・組織学的画像を統合活用できる新規深層学習モデル「STAIG」を開発し、サンプル間のバッチ効果を低減した高精度な空間ドメイン解析を可能にしました。
本研究では、ガウシアンフィルタやバンドパスフィルタでノイズを除去した病理組織学において最も標準的なHE(ヘマトキシリン・エオジン)染色画像から得た特徴を、自己教師あり学習モデル(BYOL)により埋め込みベクトルへ変換し、空間情報に基づくグラフ構造と組み合わせる技術を用いることで、グラフノード(スポット)間の近傍関係を保ったまま統合解析することに成功しました。先行研究と比較して座標整合を必要としない点に新規性があり、多種多様な組織サンプルに適用可能であるため、がん微小環境などの空間生物学研究のさらなる発展が期待されます。
本研究成果は2025年1月26日、「Nature Communications」にオンライン掲載されました。
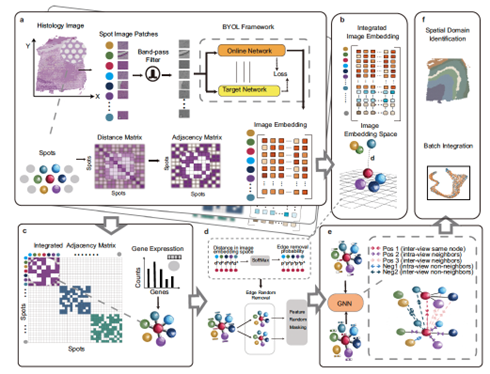
図1:STAIGフレームワークの概要
STAIGフレームワークは、空間トランスクリプトミクスデータを効果的に解析するための一連のプロセスを統合的に示しており、画像処理とグラフ対照学習を組み合わせることで高精度な空間ドメインの同定と統合を実現しています。
発表内容
近年、空間トランスクリプトーム解析(ST、注1)技術の進展により、空間的な遺伝子発現パターンを組織内にマッピングすることが可能となりました。しかし、空間ドメイン同定には、しばしば遺伝子発現情報や座標のみを用いる手法では限界があり、組織学的画像情報を統合することで、より精度の高い空間区分を行うことが求められています。既存手法には、画像の染色濃度変化に左右されやすい、あるいは事前に大量の画像データで学習が必要であるなどの問題点がありました。また、複数の組織切片を統合解析する際には座標整合が必要となるケースが多く、バッチ効果の存在が統合解析を困難にしていました。そこで、東京大学大学院新領域創成科学研究科の大学院生YANG Yitao(ヨウ ヤクトウ)を中心とする東京大学医科学研究所機能解析イン・シリコ分野の中井研究チームは、新たなアプローチ「STAIG」を提案しました。STAIGは以下の特長を有します。
・画像情報からの頑健な特徴抽出
STAIGはHE染色画像から色むらを減らし、組織構造に基づく特徴を抽出するため、ガウシアンおよびバンドパスフィルタ(注2)を活用します。さらに自己教師あり学習手法BYOL(注3)によって、追加の巨大な画像学習セット無しで、既存の組織画像から高品質な特徴ベクトルを獲得可能にしました(図2)。
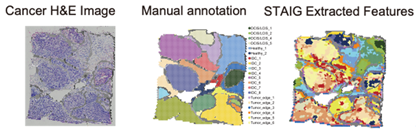
図2:画像特徴抽出の結果
左は標準的ST情報取得システムVisium 10x由来のヒト乳腺癌切片のHE染色画像、中はアノテーション(正解と見做せる)、右は抽出された画像特徴によるクラスタリング結果を示す。
・グラフ対照学習による空間的関係維持とバイアス低減
STデータのスポットをノードとするグラフを構築し、空間的近傍関係や画像類似度を反映させながら、ランダムな辺除去や遺伝子特徴マスクを加えた二つの異なるグラフビュー間で対照学習を実行します。これにより、ノイズや初期バイアスの影響を抑えながら近傍関係を強調し、高精度なドメイン分割が可能になりました(図3)。
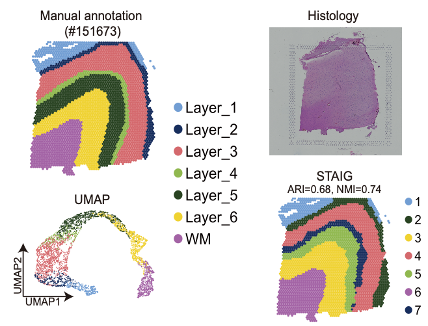
図3:Visiumヒト脳DLPFCデータセットにおける空間的ドメイン解析
左から順に、アノテーション、HE染色画像、UMAP解析結果、STAIG解析結果を示す。
・アラインメント不要な統合解析とバッチ効果除去
STAIGは、異なる組織切片や異なる実験条件で得られたデータを、事前の座標整合なしで統合できます。対照学習によって、同一生物学的ドメインに属するサンプル同士の埋め込み空間での近接性を確保し、バッチ効果を大幅に低減します。これにより、連続するスライス間や異種データ間での統合的な特徴抽出が可能となりました(図4)。
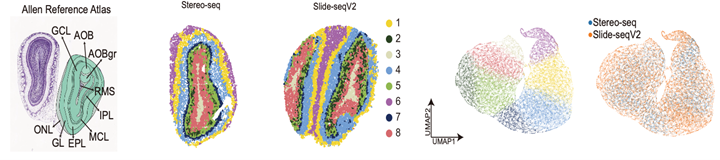
図4:クロスプラットフォーム統合結果
異なる実験プラットフォームであるStereo-seqおよびSlide-seqV2由来のヒト脳海馬領域のデータセットに対する統合結果を示す。
発表者・研究者等情報
東京大学
大学院新領域創成科学研究科メディカル情報生命専攻
YANG Yitao(ヨウ ヤクトウ) 博士課程
CUI Yang(サイ ヨウ) 博士課程
ZENG Xin(ゼン シン) 博士課程(研究当時)
ZHANG Yubo(チョウ ウハ) 博士課程(研究当時)
医科学研究所 附属ヒトゲノム解析センター 機能解析イン・シリコ分野
中井 謙太 教授
兼:東京大学大学院新領域創成科学研究科メディカル情報生命専攻
朴 聖俊 准教授
Martin de Jesus LOZA-LOPEZ 助教
論文情報
雑誌名:Nature Communications
題 名:STAIG: Spatial transcriptomics analysis via image-aided graph contrastive learning for domain exploration and alignment-free integration
著者名:Yitao Yang, Yang Cui, Xin Zeng, Yubo Zhang, Martin Loza, Sung-Joon Park & Kenta Nakai
DOI: 10.1038/s41467-025-56276-0
URL: https://www.nature.com/articles/s41467-025-56276-0
研究助成
本研究は、科研費「22K06189」、「JP22K21301」、「20H05940」及JST SPRING [JPMJSP2108]の支援により実施されました。
用語解説
(注1)空間トランスクリプトーム解析(ST)
組織内の空間情報と遺伝子発現データを同時に取得し、細胞の空間的配置や相互作用を解析する手法。
(注2)ガウシアンおよびバンドパスフィルタ
画像のノイズ低減や特定の周波数帯域の信号抽出に使用されるフィルタ。ガウシアンフィルタは画像の平滑化、バンドパスフィルタは特定の構造情報の抽出に適用される。
(注3)BYOL
Bootstrap Your Own Latent の略称。自己教師あり学習の一種で、教師ラベルなしに高品質な特徴表現(埋め込みベクトル)を学習するモデル。
問合せ先
〈研究に関する問合せ〉
国立大学法人東京大学大学院医科学研究所 附属ヒトゲノム解析センター 機能解析イン・シリコ分野
教授 中井 謙太(なかい けんた)
〈報道に関する問合せ〉
国立大学法人東京大学医科学研究所 プロジェクトコーディネーター室(広報)