2021-11-25 理化学研究所,広島大学
理化学研究所(理研)生命医科学研究センターがんゲノム研究チームの中川英刀チームリーダー、広島大学大学院医系科学研究科の茶山一彰教授らの国際共同研究グループは、B型肝炎から発生した肝臓がんのゲノム解析により、B型肝炎ウイルス(HBV)のゲノムが肝臓がんのゲノムに組み込まれ、「染色体転座[1]」を引き起こすことを発見しました。
本研究成果は、B型肝炎から発生する肝臓がんの予防法や治療法の開発に貢献すると期待できます。
今回、国際共同研究グループは、全ゲノムがん種横断的解析プロジェクト(PCAWG)[2]で収集した296例の肝臓がんの全ゲノムシークエンスデータを再解析しました。51例に計148カ所のHBVゲノムの組み込み[3]現象を検出しましたが、そのうち106カ所の全体像は不明でした。そこで、最新の長鎖シークエンス技術[4]を用いて、9例について全ゲノム解析を行った結果、7例でHBVゲノムの組み込みを介して染色体転座が起きていることを発見しました。これらの染色体転座は、TP53などの重要ながん抑制遺伝子[5]が存在する染色体のテロメア[6]領域の欠損につながることも分かりました。染色体転座およびそれに伴うテロメア領域の欠損は、少なくとも約8%の肝臓がんにおいて、発生の初期段階で起きていると推定されました。
本研究は、オンライン科学雑誌『Nature Communications』(11月25日付:日本時間11月25日)に掲載されます。
背景
B型肝炎ウイルス(HBV)の感染者は全世界で約3億5000万人以上に上り、日本では約130~150万人(約100人に1人)と推定されています注1)。HBVは肝臓に感染し、炎症を起こします。B型肝炎が持続すると、慢性肝炎から肝硬変、さらには肝臓がんへと進展する可能性があり、全世界で毎年約88万人が肝臓がんや肝硬変といった合併症で死亡しています注1)。
HBVはDNAウイルスです。HBVゲノムは全長が3,200塩基対(bp)程度の環状2本鎖DNAであり、6種類の遺伝子をコードしています。HBVが肝細胞に感染すると、そのDNAが細胞核に送り込まれ、自己複製します。その際、HBVのDNAが肝細胞のヒトゲノム(約30億bp)に組み込まれることが以前から分かっていました。
中川チームリーダーらはこれまで、短鎖次世代シークエンサー[7]を用いて、ウイルス性肝臓がんを主とした全ゲノムシークエンス解析[7]を行ってきました注2)。また、国際連携全ゲノムがん種横断的解析プロジェクト(PCAWG)において、38種類のがん腫のゲノム構造異常[8]やウイルスゲノムの組み込みを含む変異の全ゲノムレベルにおけるカタログを作成し公開しました注3)。これらの解析では、肝臓がんにおいてHBVや他のウイルスのヒトゲノムへの組み込み現象を検出しています。ウイルスゲノムが組み込まれると、肝細胞内の組み込み部位周辺のヒトゲノムが不安定になり、がん関連遺伝子の発現が変化し、肝臓がんの発生につながると考えられています。
今回、国際共同研究グループは、肝臓がんにおけるヒトゲノムへのHBVの組み込みについて、新しい長鎖シークエンス技術を用いて、その詳細と全体像について調べました。
注1)WHOホームページ(英語)
注2)2016年4月12日プレスリリース「肝臓がん300例の全ゲノムを解読」
注3)2020年2月6日プレスリリース「国際連携によるがん全ゲノムの大規模解析」
研究手法と成果
国際共同研究グループは、国際がんゲノムコンソーシアム/米国大規模がんゲノムプロジェククト(ICGC/TCGA)のPCAWGで収集した296例の肝臓がんの短鎖シークエンス(長さ100~150bp)での全ゲノムシークエンスデータを再解析した結果、51例の肝臓がんに計148カ所のHBVゲノムの組み込み現象を検出しました。しかし、そのうち組み込み部位(ヒトゲノムとHBVゲノムの境界)の両端が同定できたのは42カ所しかなく(両接続点同定)、残りの106カ所の組み込みの全体像は不明だったことから、HBVゲノムの組み込みを通して、複雑なゲノム構造異常が起きている可能性が考えられました(単接続点同定、図1)。
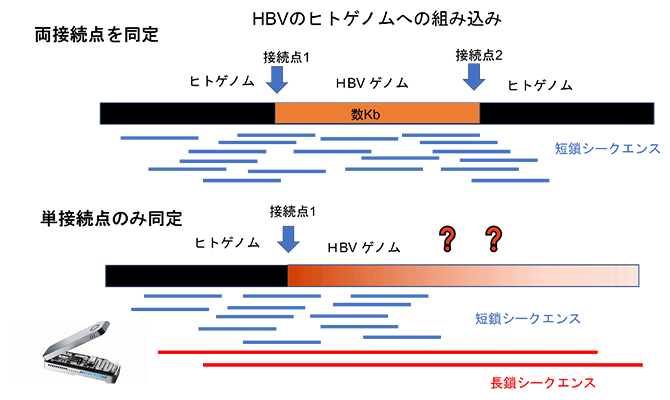
図1 長鎖シークエンスによる単接続点同定のHBV組み込みの解析
通常のウイルスゲノムの組み込みは、短鎖シークエンス解析にて両端の接続点が同定される(上)。しかし、106カ所のHBV組み込みについては接続点が一つしか同定されず、その全体像は不明であったため、長鎖シークエンス解析を行った。
そこで、近年進歩が著しい長鎖シークエンス技術であるオックスフォード・ナノポア[4]の技術を用いて、51例のうち9例の肝臓がんについて全ゲノムシークエンス解析を行い、HBVゲノムの組み込みに関わるゲノム構造異常の全体像を調べました。平均のシークエンスリードの長さは7.8キロベース(kb、1kbは1,000ベース)、最大で217kb長のリードが得られ、HBVゲノム配列(約3.2kb)を含む長鎖シークエンスリードを詳細に解析しました。
その結果、HBVゲノムの組み込みによって、肝臓がん内での別々の染色体同士がつながっていることが分かりました。1例目では、11番染色体と19番染色体とが640bpのHBVゲノム配列を通してつながっていました(図2)。これは、HBVの組み込みによって、11番染色体と19番染色体の間で「染色体転座(染色体の異常な再構成)」が起きたことを示しています。
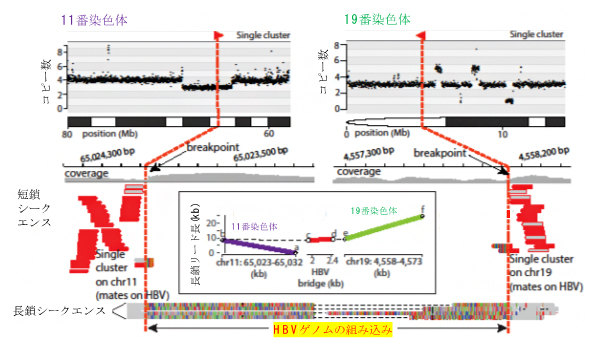
図2 長鎖シークエンスにより11番染色体と19番染色体間の転座を同定
11番染色体のDNA配列と19番染色体のDNA配列とが、640塩基のHBVのDNA配列を介してつながっている。
このような全体像は、従来の短鎖次世代シークエンサーによる解析では分かりませんでした。9例の肝臓がんのうち7例について、計9カ所のHBVのDNAを介する染色体転座の全体像を調べることができました。その例を図3左に示しますが、多種多様な染色体転座が検出されています。
また、これら染色体転座をコピー数異常[9]のプロファイルデータと比較したところ、単接続点同定のHBV組み込み部位と、コピー数異常が起きている染色体部位が一致しており、染色体転座を通してテロメア領域を欠損しているものが、296例の肝臓がんのうち23例(約8%)で見つかりました。テロメア領域の欠損を起こしている染色体部位には、TP53遺伝子(17番染色体短腕、図3右)、ARID1A遺伝子(1番染色体短腕末端)、RB1遺伝子(13番染色体長腕)、RPS6KA3遺伝子(X染色体短腕末端)、IRF2遺伝子(4番染色体長腕末端)など、さまざまながん抑制遺伝子やドライバー遺伝子[5]が存在していました。この結果から、HBVの組み込みを通して染色体転座が起こり、同時にがん抑制遺伝子/ドライバー遺伝子が存在する染色体のテロメア領域の欠損も起こり、これらの遺伝子が欠失されることで、肝臓がんの発生につながっていると考えられます。
また、これらコピー数異常のデータを使って、進化論的な解析を行ったところ、これら単接続点同定型のHBVゲノム組み込みとそれに伴う染色体転座は、がん発生の初期段階で起きていると推定されました。
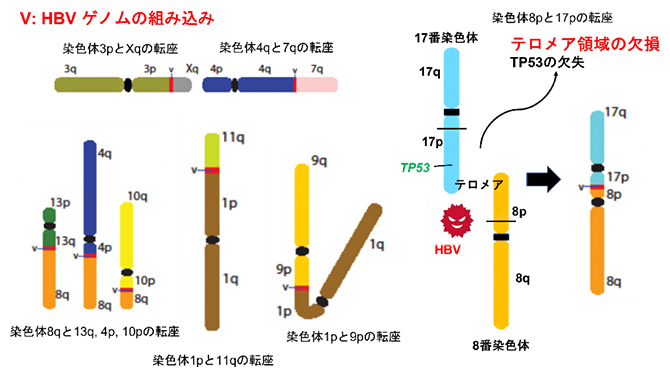
図3 HBV組み込みを介した肝臓がんの染色体転座とテロメア領域の欠損
左)HBVゲノムの配列(赤)を介して、さまざまな染色体間の転座が起きている。pは短腕、qは長腕を意味する。
右)染色体8pと17pの転座により、がん抑制遺伝子TP53を含む17pのテロメア部位が欠損する。
今後の期待
HBV感染は、慢性の感染状態(慢性肝炎)の状態から、肝臓の線維化・肝硬変、そして肝がんの発生につながる反面、それらの治療法は限られています。今回得られたHBVのヒトゲノムへの組み込み機序の研究結果は、今後、B型肝炎から発生する肝臓がんの新しい治療薬・早期診断法、予防法の開発およびウイルス組み込みに照準を絞った新しいウイルス治療薬の開発へ貢献するものと期待できます。
補足説明
1.染色体転座
ヒトの染色体は46本あるが、別々の染色体が融合して、染色体が異常に再構成されることを染色体転座という。がんの場合、多くの染色体転座や異常が観察され、染色体転座によって融合遺伝子が発現し、それががん化の最大の原因になることがある。9番染色体と22番染色体間の転座は、慢性骨髄性白血病の発生原因である。
2.全ゲノムがん種横断的解析プロジェクト(PCAWG)
37カ国から1,300人以上が参加する国際共同研究グループ「The ICGC/TCGA Pan-Cancer Analysis of Whole Genomes Consortium」が2,800例以上の短鎖シークエンスでの全ゲノムシークエンス解析を行った結果、4600万個を超える変異・異常を同定し、その特徴を明らかにした。理研のチームは、この大規模がんゲノム解析に10%の貢献をしてきた。PCAWGはPan-Cancer Analysis of Whole Genomesの略。
3.HBVゲノムの組み込み
ウイルスゲノムの組み込みとは、ウイルスのDNA配列がヒトのゲノム配列に入り込むことを指す。HBVの場合、まずウイルスに感染した正常肝臓組織においてHBVゲノムの組み込みが起こり、それが契機となってがん化が起こり、がん化に伴いがん細胞にも引き継がれると考えられている。
4.長鎖シークエンス技術、オックスフォード・ナノポア
近年、DNA配列の長さが数kb以上にもなる次世代シークエンサーが開発されている。、今回の研究で用いた、オックスフォード・ナノポア(Oxford Nanopore)という技術では、膜状の多数のナノレベルの小さな穴(ポア)を配置し、その穴にDNA1分子が通過する際に発生する電流の変化を解析して、DNAの配列を決定する。最大1,000kb以上までのDNA配列を決めることができ、大きなゲノム構造を決めるには非常に有用である。しかし、エラーが10%以上あり、小さな塩基の変化を見るには適していない。
5.がん抑制遺伝子、ドライバー遺伝子
がん抑制遺伝子は、そのコピー数の欠失や機能喪失変異によって機能を喪失し、がんが発生する。TP53やRB1は、代表的ながん抑制遺伝子である。また、さまざまな発がん過程で、変異によってその機能や発現が変化し、直接、発がんやがんの進展と関連するものをドライバー遺伝子と呼ぶ。TP53、CTNNB1、ARID1A、ARID2、RPS6KA3、IRF2は肝臓がんの代表的はドライバー遺伝子である。
6.テロメア
染色体の末端部にある構造であり、染色体末端を保護する役割を担う。
7.短鎖次世代シークエンサー、全ゲノムシークエンス解析
ヒトゲノムの全配列(約30億塩基)を1,000米ドル以下のコストで解読すべく、欧米の政府や企業が技術開発を行った結果、より高速高精度の性能を持つシークエンサーが開発された。これを「短鎖次世代シークエンサー」という。従来の方法に比べ、超大量のDNAシークエンス反応を並列して行うことができる。100~150塩基の長さのDNA配列(ATGC配列)を大量に読み込んで、個人やがんの全ゲノム情報を解読し、配列の違いや変化を同定する「全ゲノムシークエンス解析」が現在のゲノム解析の基本手法になりつつある。しかし、短鎖次世代シークエンサーではゲノム構造異常などの大きな配列の変化を捉えにくい。
8.ゲノム構造異常
1塩基の配列が変化する点突然変異と異なり、数百~数百万塩基の配列が欠失、組み込み、重複、逆位(方向が逆になる)、染色体間で転座する(移動する)など、大きなゲノム配列の変化をいう。全ゲノムシークエンス解析にて、網羅的に検出できるようになった。
9.コピー数異常
ヒトゲノムの常染色体は、2コピー(父方と母方由来)存在する。がんゲノムは、さまざまな領域または染色体の全体にわたって、コピー数が1コピーあるいは0コピーに減ることがある。その領域には、がん抑制遺伝子を含むことが多い。反対に、3コピー、4コピー~数十コピーに増える場合もある。比較的小さい領域(100bp~数MBbp)が数十コピーにまで増える現象を局所増幅といい、その領域にはHer2、MYC、EGFRなどのがん遺伝子が含まれることが多い。
国際共同研究グループ
理化学研究所 生命医科学研究センター がんゲノム研究チーム
チームリーダー 中川 英刀(なかがわ ひでわき)
広島大学 大学院医系科学研究科
教授 茶山 一彰(ちゃやま かずあき)
サンティアゴ・デ・コンポステーラ大学(スペイン)
教授 ホセ・トゥビオ(Jose M. C. Tubio)
フランシスクリック研究所(英国)
教授 ピータ・フォンロー(Peter Van Loo)
ウエルカムサンガー研究所(英国)
上級グループリーダー ピータ・キャンベル(Peter J. Campbell)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金基盤研究(A)「肝胆膵がんの多元的オミックス解析による分子機構の解明(研究代表者:中川英刀)」、および理化学研究所 共生生物学プロジェクトによる支援を受けて行われました。
原論文情報
Eva G. Alvarez, Jonas Demeulemeester, Paula Otero, Clemency Jolly, Daniel Garcia-Souto, Ana Pequeno, Jorge Zamora, Marta Tojo, Javier Temes, Adrian Baez-Ortega,Bernardo Rodriguez-Martin, Ana Oitaben, Alicia L. Bruzos, Monica Martinez-Fernandez, Kerstin Haase, Martin Santamarina, Sonia Zumalave, Rosanna Abal, Jorge Rodriguez-Castro, Aitor Rodriguez-Casanova, Angel Diaz-Lagares, Yilong Li, Keiran Raine, Adam P. Butler, Kazuaki Maejima, Atsushi Ono, Hiroshi Aikata, Kazuaki Chayama, Masaki Ueno, Shinya Hayami, Hiroki Yamaue, Miguel G. Blanco, Xavier Forns, Carmen Rivas, Juan Ruiz-Banobre, Sofia Perez-del-Pulgar, Raul Torres-Ruiz, Sandra Rodriguez Perales, Urtzi Garaigorta*, Peter J. Campbell*, Hidewaki Nakagawa*, Peter Van Loo* & Jose M. C.Tubio (* equally contribution), “Aberrant integration of Hepatitis B virus DNA promotes major restructuring of human hepatocellular carcinoma genome architecture”, Nature Communications, 10.1038/s41467-021-26805-8
発表者
理化学研究所
生命医科学研究センター がんゲノム研究チーム
チームリーダー 中川 英刀(なかがわ ひでわき)
広島大学 大学院医系科学研究科
教授 茶山 一彰(ちゃやま かずあき)
報道担当
理化学研究所 広報室 報道担当
広島大学 財務・総務室広報部広報グループ