2022-10-27 京都大学iPS細胞研究所
ポイント
- 新型コロナウイルス感染においてEXOC2は重要な宿主因子である。
- ACE2発現iPS細胞および気道オルガノイドにおいてEXOC2の発現抑制を行うことによって、新型コロナウイルス感染効率が低下する。
- EXOC2発現抑制による新型コロナウイルス感染効率の低下は、IFNW1の発現制御を介する。
1. 要旨
Renxing Yi大学院生(CiRA増殖分化機構研究部門)、高山 和雄 講師(CiRA同部門)らの研究チームは、新型コロナウイルス(SARS-CoV-2)注1感染においてExocyst component 2 (EXOC2) 注2が重要な宿主細胞のタンパク質(宿主因子)であることを確認しました。また、EXOC2発現抑制はInterferon omega-1 (IFNW1) 注3の発現誘導を介して、SARS-CoV-2感染効率を低下させることを示した。
本研究では、まず、既報の遺伝子解析のデータ(CRISPRスクリーニングやRNA-seq解析)に基づき、SARS-CoV-2の感染に、EXOC2が特に重要であることを予想しました。実際に、ACE2注4発現iPS細胞および気道オルガノイド注5を使用して、CRISPR干渉(CRISPRi)注6システムによりEXOC2の発現抑制をすると、SARS-CoV-2感染効率が低下することを確認しました。また、EXOC2の発現抑制によるSARS-CoV-2感染効率の低下はIFNW1の発現増加を介したものであることを明らかにしました。
これらの結果から、EXOC2はSARS-CoV-2の感染に重要な宿主因子であることが分かるとともに、EXOC2およびIFNW1の発現制御が新型コロナウイルス感染症(COVID-19)注7の新規治療法の開拓に繋がることが期待されます。
この研究成果は2022年10月22日(米国時間)に「iScience」でオンライン公開されました。
2. 研究の背景
COVID-19治療薬の多くは、SARS-CoV-2の構造タンパク質や非構造タンパク質を標的としています。ウイルスの構造タンパク質および非構造タンパク質は次々の変異を獲得する恐れがあり、新たな変異の出現によりこれらの治療薬が効果を示さなくなる可能性があります。そこで、SARS-CoV-2の感染に必須の宿主因子に着目した創薬研究を行うことで、ウイルスの変異の獲得に影響されない治療薬を開発できると期待されています。しかしながら、SARS-CoV-2の感染に必須の宿主因子の解析は、ACE2やTMPRSS2注8などの受容体やプロテアーゼに関するものが中心であり、それ以外の宿主因子の解析はいまだ十分には進んでおりません。
本研究では、SARS-CoV-2感染に関与する宿主因子のCRISPRスクリーニングの結果とSARS-CoV-2感染時のRNA-seq解析データを調べ、SARS-CoV-2感染に重要な宿主因子としてEXOC2に着目し、その機能解析研究を実施しました。
3. 研究結果
1) SARS-CoV-2 感染に必要な宿主因子のスクリーニング
まず、SARS-CoV-2感染に必要な宿主因子をCRISPRスクリーニングにより探索している複数の論文から、51個の宿主因子をリストアップしました(図1A)。iPS細胞のRNA-seq解析データに基づき、内在性の遺伝子発現量が高い15遺伝子を抽出しました。この15遺伝子のうち、ACE2発現iPS細胞と気道オルガノイドにおいて、SARS-CoV-2感染によって発現量が変動する遺伝子を探索し、EXOC2、RAD54L2、DRG1、ARID1Aの計4遺伝子に絞り込みました。
続いて、気道オルガノイドにおいてこれら4遺伝子の発現量をsiRNA注9により抑制し、SARS-CoV-2感染効率への影響を評価しました。いずれの遺伝子を発現抑制した場合も培養上清中のウイルスゲノム量は減少しましたが、特にEXOC2発現抑制によるウイルスゲノム量の減少が顕著であることが分かりました(図1B)。したがって、EXOC2は、SARS-CoV-2感染における重要な宿主因子であることが示唆されました。
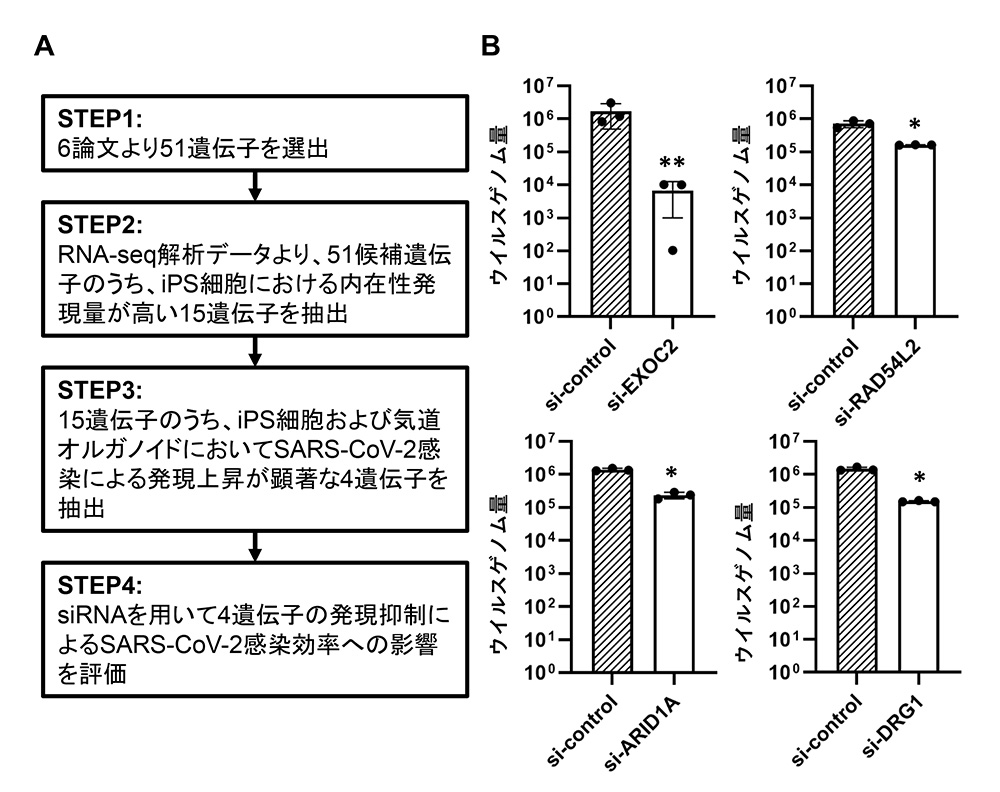
図1:SARS-CoV-2感染に重要な宿主因子の探索
(A) 宿主因子の絞り込みの流れ。(B) siRNAを用いた4遺伝子のノックダウンによるウイルスゲノム量への影響を評価。*:p <0.05、**p <0.01の有意差を示す。
2) EXOC2ノックダウンiPS細胞の樹立
EXOC2の機能解析のため、CRISPRiシステムを用いて、EXOC2の発現量を可逆的に減らすことができるiPS細胞を樹立しました(図2A)。EXOC2を標的にしたガイドRNAを発現させたiPS細胞において、DOXを作用させるとEXOC2の発現量が減少します(1%以下)(図2B)。
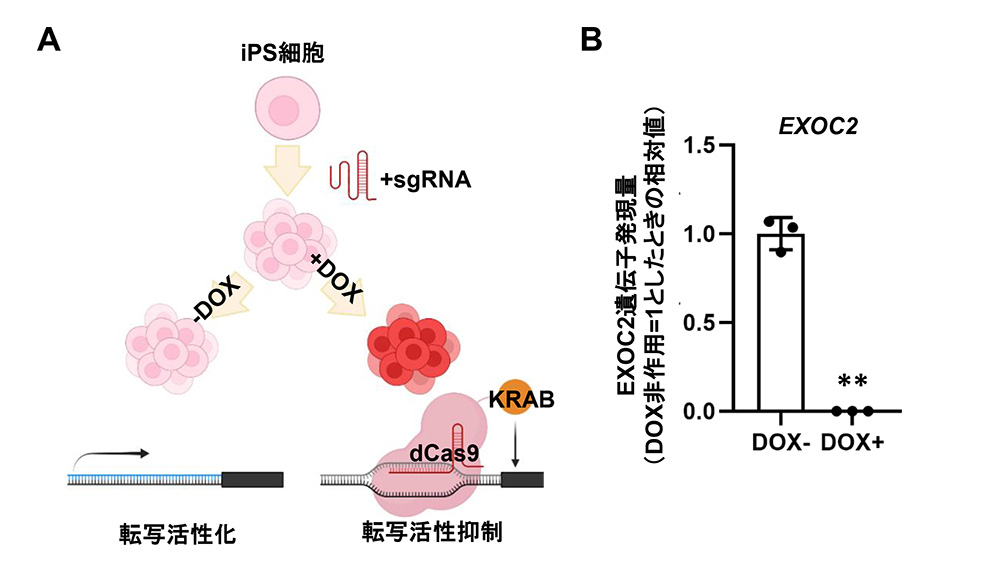
図2:EXOC2ノックダウンiPS細胞の樹立
(A) 本研究で用いたCRISPRiシステムの仕組み。(B) DOX作用によりEXOC2遺伝子発現量が減少。**p <0.01の有意差を示す。
3) EXOC2発現抑制によりSARS-CoV-2感染効率が低下する
上記で樹立したEXOC2ノックダウンiPS細胞を用いて、EXOC2発現抑制のSARS-CoV-2感染効率への影響を評価しました。
その結果、EXOC2発現抑制によりウイルスゲノム量は1.7%に減少しました(図3A)。その際に、ウイルスタンパク質の存在を示すSARS-CoV-2ヌクレオカプシド(N)注10陽性である細胞も減少しました(図3B)。また、SARS-CoV-2 B.1.1.214、BA.1、BA.2、B.1.617.2注11の4種類の変異株すべてに対して、EXOC2発現抑制による感染効率の低下を確認しました(図3C)。
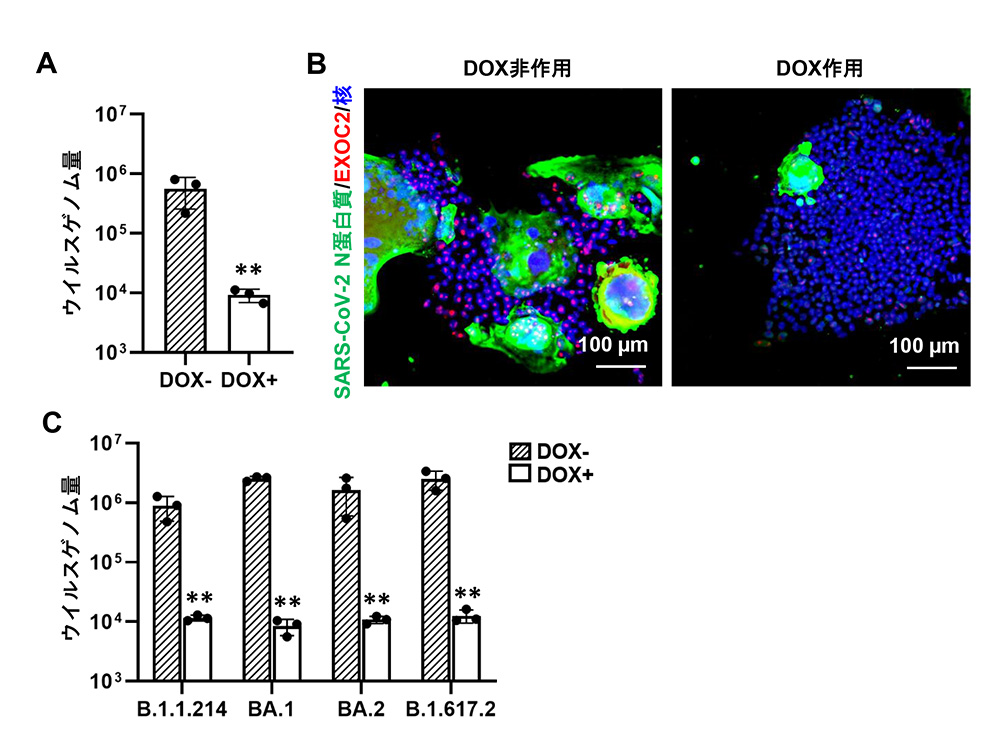
図3:EXOC2発現抑制によりSARS-CoV-2感染効率が低下する
(A) 本研究で用いたCRISPRiシステムの仕組み。(B) DOX作用によりEXOC2遺伝子発現量が減少。**p <0.01の有意差を示す。
4) EXOC2発現抑制によるウイルス感染効率低下はIFNW1発現制御を介する
EXOC2は自然免疫応答に重要であることが報告されています。EXOC2発現抑制によって、IFNW1遺伝子含むいくつかのIFN遺伝子の発現量が上昇しました(図4A)。TMPRSS2を発現させたVero細胞(アフリカミドリザル腎由来細胞株)を用いた検討で、IFNW1/IFN-ωが抗ウイルス効果を示すことを確認できたため、EXOC2発現抑制がIFNW1の発現誘導を介して、SARS-CoV-2感染効率を低下させるという仮説を立てました。
この仮説を検証するため、EXOC2およびIFW1の両方を発現抑制できるiPS細胞を作製しました。EXOC2発現抑制によるウイルスゲノム量の減少およびSARS-CoV-2 Nタンパク質の減少は、IFNW1の発現抑制によりキャンセルされました(図4B、4C)。以上の結果から、EXOC2発現抑制は、IFNW1の発現誘導を介して、SARS-CoV-2感染効率を低下させることが示唆されました。
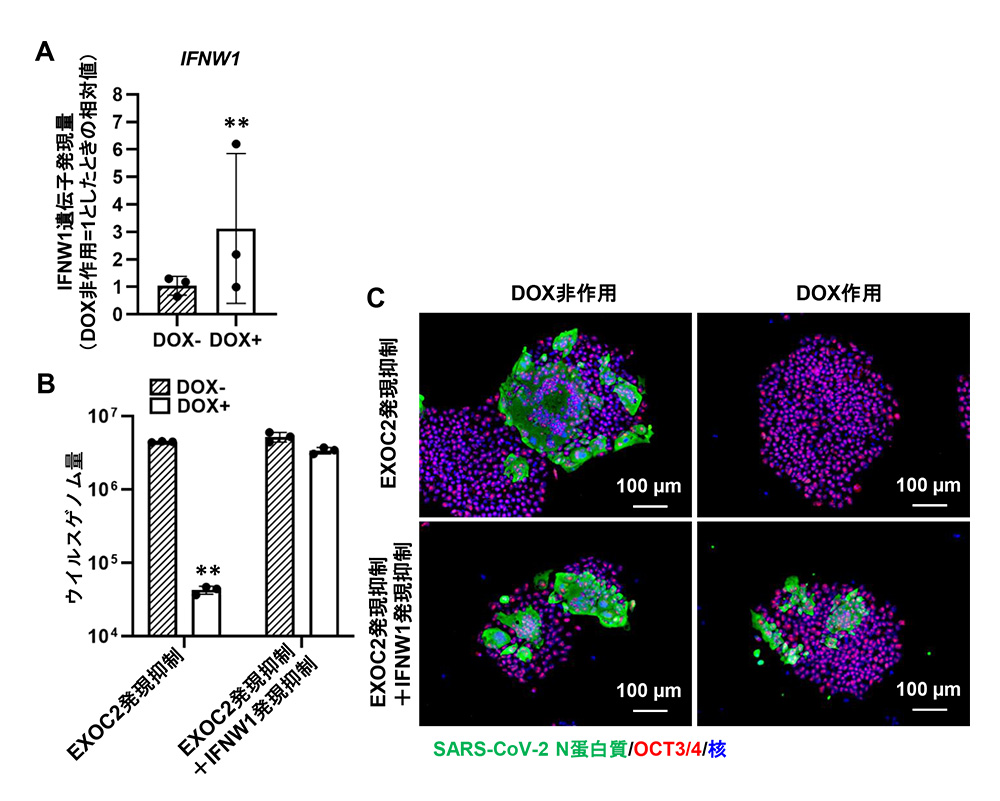
図4:EXOC2発現抑制によるSARS-CoV-2感染効率の低下には、IFNW1の発現誘導を介する
(A) EXOC2発現抑制によりIFNW1発現量が増加。(B) (C) EXOC2発現抑制によるウイルスゲノム量およびSARS-CoV-2 Nタンパク質の減少が、IFNW1発現抑制によりキャンセルされる。**:p <0.01の有意差を示す。
4. まとめ
本研究では、EXOC2がSARS-CoV-2感染に必要な宿主因子の一つであることを示しました。CRISPRiシステムおよびsiRNAを用いたEXOC2発現抑制は、ACE2発現iPS細胞および気道オルガノイドにおけるSARS-CoV-2の感染効率を低下させました。EXOC2発現抑制はIFNW1の発現誘導を介してSARS-CoV-2の感染効率を低下させることを見出しました。本研究により得られた知見が、COVID-19治療薬開発に役立つことを期待します。
5. 論文名と著者
- 論文名
Exocyst complex component 2 is a potential host factor for SARS-CoV-2 infection - ジャーナル名
iScience - 著者
Renxing Yi1#, Rina Hashimoto1, Ayaka Sakamoto1, Yasufumi Matsumura2, Miki Nagao2, Kazutoshi Takahashi1, Kazuo Takayama1,3*
#筆頭著者 *責任著者 - 著者の所属機関
- 京都大学iPS細胞研究所(CiRA)
- 京都大学大学院医学研究科臨床病態検査学
- 日本医療研究開発機構革新的先端研究開発支援事業(AMED-CREST)
6. 本研究への支援
本研究は、下記機関より支援を受けて実施されました。
- 京都大学基金 iPS細胞研究基金
- 京都大学iPS細胞研究所山中伸弥研究室への新型コロナウイルス特別研究助成
- 京都大学 ウイルス感染症・生命科学先端融合的共同研究拠点
- 国立研究開発法人科学技術振興機構(JPMJSP2110)
- 国立研究開発法人日本医療研究開発機構(JP20fk0108533, JP21fk0108492, JP21gm1610005)
7. 用語説明
注1)SARS-CoV-2
新型コロナウイルス(severe acute respiratory syndrome coronavirus 2)。新型コロナウイルス感染症(COVID-19)の原因ウイルス。様々な変異型が存在することが知られている。
注2)EXOC2
Exocyst component 2 (EXOC2)。エクソシスト複合体の構成タンパク質の一つである。CRISPRスクリーニングによりSARS-CoV-2感染との関与が示唆されている。
注3)IFNW1
Interferon omega-1 (IFNW1)。I型 IFNに分類されます。細胞側のIFN受容体に結合して、JAK/STATシグナル経路などを活性化する。組換えタンパク質は抗ウイルス効果を示す。
注4)ACE2
アンジオテンシン変換酵素Ⅱ(Angiotensin-converting enzyme 2)。SARS-CoV-2の受容体。
注5)気道オルガノイド
線毛細胞、クラブ細胞、ゴブレット細胞、神経内分泌細胞からなる気道様三次元構造体。SARS-CoV-2感染実験に使用することができ、感染細胞の特定、宿主細胞における応答、治療薬の評価などを実施できる。
注6)CRISPR干渉(CRISPRi)
CRISPRiとは、特異性の高い遺伝子発現抑制技術である。ヌクレアーゼ活性の無いdCas9と転写抑制ドメインKRABを融合したタンパク質とsgRNAを用いることで、任意の遺伝子の発現を抑制することができる。ゲノム編集技術に使用されているCas9を応用した技術である。
注7)新型コロナウイルス感染症(COVID-19)
新型コロナウイルス感染症の正式名称。CO=「corona」、VI=「virus」、D=「disease」。
注8)TMPRSS2
Ⅱ型膜貫通型セリンプロテアーゼ(transmembrane protease, serine 2)の一種。SARS-CoV-2の表面にあるタンパク質を切断することで宿主細胞への侵入を促進する。
注9)siRNA(small interfering RNA)
21-23塩基対からなる低分子二本鎖RNAで、細胞内に導入する事で遺伝子発現を抑制する。
注10)SARS-CoV-2ヌクレオカプシド(N)
SARS-CoV-2に最も多く存在するタンパク質であり、ウイルスの中身に存在する。
注11)SARS-CoV-2 B.1.1.214、BA.1、BA.2、B.1.617.2
SARS-CoV-2変異株の一種。B.1.1.214は2020年秋以降の第三波で確認された変異株。B.1.617.2は2021年夏に流行した変異株。B.1.617.2はデルタ株とも呼ばれる。BA.1、BA.2は2021年末以降に流行した変異株。BA.1、BA.2はオミクロン株とも呼ばれる。