先天性異常、流産、がんに関する知見を得る可能性がある May provide insight into birth defects, miscarriage, cancer
2023-02-09 ワシントン大学セントルイス校
◆このような遺伝子実験は、通常、実験室でマウスやゼブラフィッシュなどの動物モデルを用いて行われ、数十年にわたり発生生物学の研究の中心となってきた。遺伝子を欠損させたり過剰発現させたりする動物実験では、遺伝子の機能について多くを知ることができるが、こうした実験には費用と時間がかかるという欠点もある。
◆これに対して、新たに開発された『CellOracle』(2月8日付『Nature』誌に掲載)は、数百の遺伝子実験を数分でモデル化することができ、科学者が、発生において重要な役割を果たすが旧来の遅い手法では見落とされていたかもしれない主要遺伝子を特定するのに役立つという。CellOracleはオープンソースであり、コードとソフトウェアに関する情報はこのリンクから入手できます。
◆Nature誌の最近の技術特集に掲載されたCellOracleは、細胞内の遺伝子制御に関する洞察をモデル化するために設計された、比較的新しいソフトウェアシステムの一つです。CellOracleは、単にネットワークを特定するだけでなく、特定の方法でネットワークが破壊されたときに何が起こるかを研究者に検証させることができる点が特徴である。
<関連情報>
- https://source.wustl.edu/2023/02/computer-model-ids-roles-of-individual-genes-in-early-embryonic-development/
- https://www.nature.com/articles/s41586-022-05688-9
- https://www.cell.com/stem-cell-reports/fulltext/S2213-6711(22)00543-4
ネットワーク推論とインシリコ遺伝子摂動による細胞アイデンティティの解き明かし Dissecting cell identity via network inference and in silico gene perturbation
Kenji Kamimoto,Blerta Stringa,Christy M. Hoffmann,Kunal Jindal,Lilianna Solnica-Krezel & Samantha A. Morris
Nature Published:08 February 2023
DOI:https://doi.org/10.1038/s41586-022-05688-9
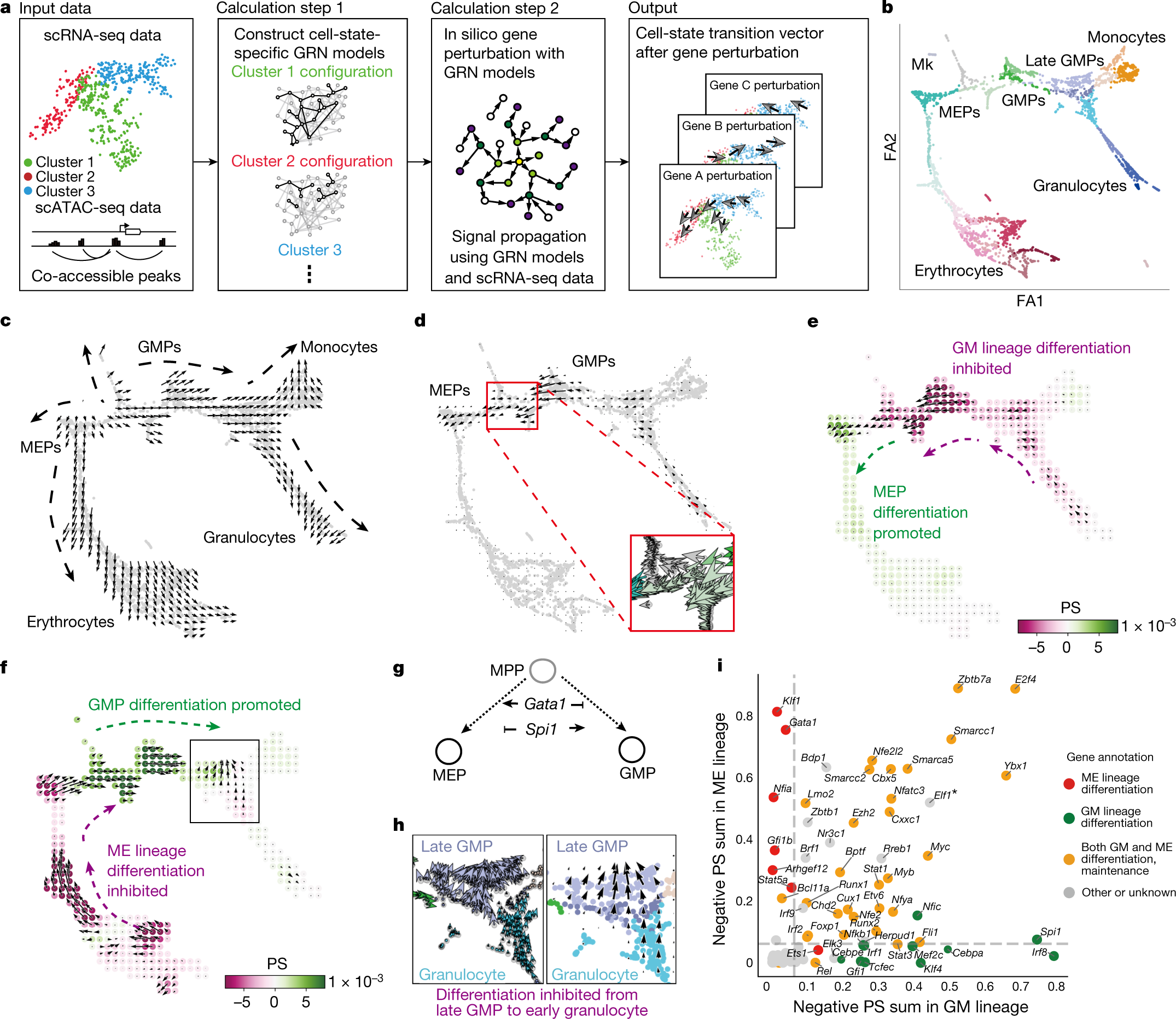
Abstract
Cell identity is governed by the complex regulation of gene expression, represented as gene-regulatory networks1. Here we use gene-regulatory networks inferred from single-cell multi-omics data to perform in silico transcription factor perturbations, simulating the consequent changes in cell identity using only unperturbed wild-type data. We apply this machine-learning-based approach, CellOracle, to well-established paradigms—mouse and human haematopoiesis, and zebrafish embryogenesis—and we correctly model reported changes in phenotype that occur as a result of transcription factor perturbation. Through systematic in silico transcription factor perturbation in the developing zebrafish, we simulate and experimentally validate a previously unreported phenotype that results from the loss of noto, an established notochord regulator. Furthermore, we identify an axial mesoderm regulator, lhx1a. Together, these results show that CellOracle can be used to analyse the regulation of cell identity by transcription factors, and can provide mechanistic insights into development and differentiation.
直接的な系統の初期化における遺伝子制御ネットワークの再構築 Gene regulatory network reconfiguration in direct lineage reprogramming
Kenji Kamimoto,Mohd Tayyab Adil,Kunal Jindal,Christy M. Hoffmann,Wenjun Kong ,Xue Yang,Samantha A. Morris
Stem Cell Reports Published:December 29, 2022
DOI:https://doi.org/10.1016/j.stemcr.2022.11.010

Highlights
•CellOracle dissects gene regulatory network reconfiguration in direct lineage reprogramming
•Lineage tracing fibroblast to endoderm progenitor reprogramming reveals early network changes
•In silico interrogation of network reconfiguration identifies new reprogramming regulators
•These analyses reveal a role for Fos with Hippo signaling effector, Yap1, in reprogramming
Summary
In direct lineage conversion, transcription factor (TF) overexpression reconfigures gene regulatory networks (GRNs) to reprogram cell identity. We previously developed CellOracle, a computational method to infer GRNs from single-cell transcriptome and epigenome data. Using inferred GRNs, CellOracle simulates gene expression changes in response to TF perturbation, enabling in silico interrogation of network reconfiguration. Here, we combine CellOracle analysis with lineage tracing of fibroblast to induced endoderm progenitor (iEP) conversion, a prototypical direct reprogramming paradigm. By linking early network state to reprogramming outcome, we reveal distinct network configurations underlying successful and failed fate conversion. Via in silico simulation of TF perturbation, we identify new factors to coax cells into successfully converting their identity, uncovering a central role for the AP-1 subunit Fos with the Hippo signaling effector, Yap1. Together, these results demonstrate the efficacy of CellOracle to infer and interpret cell-type-specific GRN configurations, providing new mechanistic insights into lineage reprogramming.


