2023-05-10 ワシントン大学セントルイス校
◆このプロジェクトには、多様な背景を持つ47人のゲノム配列が含まれており、研究者は2024年半ばまでに350人のゲノムを配列することを目標としています。このようにサンプルサイズを大きくすることで、世界のヒト集団の多様性をより包括的に理解することができるようになります。パンゲノムのリファレンスは、ヒトの遺伝学、健康、病気の研究に役立つだろう。DNA配列決定技術の進歩により、現在の参照ゲノム配列のギャップが埋まり、より完全な配列となりました。
◆このプロジェクトには、ワシントン大学、カリフォルニア大学サンタクルーズ校、ハーバード大学医学部、イェール大学、ハインリッヒ・ハイネ大学、テネシー大学など、さまざまな機関の研究者が参加しています。
<関連情報>
- https://source.wustl.edu/2023/05/detailed-human-pangenome-reference-captures-human-diversity/
- https://medicine.wustl.edu/news/detailed-human-pangenome-reference-captures-human-diversity/
- https://www.nature.com/articles/s41586-023-05896-x
ヒトパンゲノムリファレンスの草稿 A draft human pangenome reference
Wen-Wei Liao,Mobin Asri,Jana Ebler,Daniel Doerr,Marina Haukness,Glenn Hickey,Shuangjia Lu,Julian K. Lucas,Jean Monlong,Haley J. Abel,Silvia Buonaiuto,Xian H. Chang,Haoyu Cheng,Justin Chu,Vincenza Colonna,Jordan M. Eizenga,Xiaowen Feng,Christian Fischer,Robert S. Fulton,Shilpa Garg,Cristian Groza,Andrea Guarracino,William T. Harvey,Simon Heumos,Kerstin Howe,Miten Jain,Tsung-Yu Lu,Charles Markello,Fergal J. Martin,Matthew W. Mitchell,Katherine M. Munson,Moses Njagi Mwaniki,Adam M. Novak,Hugh E. Olsen,Trevor Pesout,David Porubsky,Pjotr Prins,Jonas A. Sibbesen,Jouni Sirén,Chad Tomlinson,Flavia Villani,Mitchell R. Vollger,Lucinda L. Antonacci-Fulton,Gunjan Baid,Carl A. Baker,Anastasiya Belyaeva,Konstantinos Billis,Andrew Carroll,Pi-Chuan Chang,Sarah Cody,Daniel E. Cook,Robert M. Cook-Deegan,Omar E. Cornejo,Mark Diekhans,Peter Ebert,Susan Fairley,Olivier Fedrigo,Adam L. Felsenfeld,Giulio Formenti,Adam Frankish,Yan Gao,Nanibaa’ A. Garrison,Carlos Garcia Giron,Richard E. Green,Leanne Haggerty,Kendra Hoekzema,Thibaut Hourlier,Hanlee P. Ji,Eimear E. Kenny,Barbara A. Koenig,Alexey Kolesnikov,Jan O. Korbel,Jennifer Kordosky,Sergey Koren,HoJoon Lee,Alexandra P. Lewis,Hugo Magalhães,Santiago Marco-Sola,Pierre Marijon,Ann McCartney,Jennifer McDaniel,Jacquelyn Mountcastle,Maria Nattestad,Sergey Nurk,Nathan D. Olson,Alice B. Popejoy,Daniela Puiu,Mikko Rautiainen,Allison A. Regier,Arang Rhie,Samuel Sacco,Ashley D. Sanders,Valerie A. Schneider,Baergen I. Schultz,Kishwar Shafin,Michael W. Smith,Heidi J. Sofia,Ahmad N. Abou Tayoun,Françoise Thibaud-Nissen,Francesca Floriana Tricomi,Justin Wagner,Brian Walenz,Jonathan M. D. Wood,Aleksey V. Zimin,Guillaume Bourque,Mark J. P. Chaisson,Paul Flicek,Adam M. Phillippy,Justin M. Zook,Evan E. Eichler,David Haussler,Ting Wang,Erich D. Jarvis,Karen H. Miga,Erik Garrison,Tobias Marschall,Ira M. Hall,Heng Li & Benedict Paten
Nature Published:10 May 2023
DOI:https://doi.org/10.1038/s41586-023-05896-x
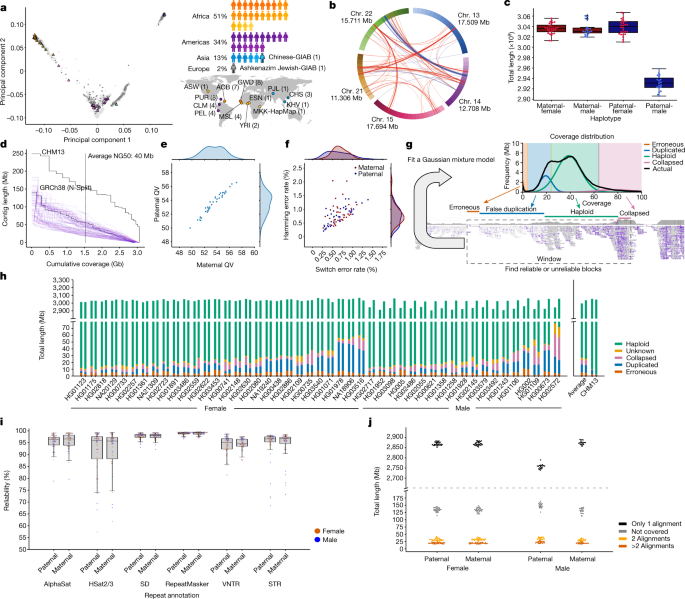
Abstract
Here the Human Pangenome Reference Consortium presents a first draft of the human pangenome reference. The pangenome contains 47 phased, diploid assemblies from a cohort of genetically diverse individuals1. These assemblies cover more than 99% of the expected sequence in each genome and are more than 99% accurate at the structural and base pair levels. Based on alignments of the assemblies, we generate a draft pangenome that captures known variants and haplotypes and reveals new alleles at structurally complex loci. We also add 119 million base pairs of euchromatic polymorphic sequences and 1,115 gene duplications relative to the existing reference GRCh38. Roughly 90 million of the additional base pairs are derived from structural variation. Using our draft pangenome to analyse short-read data reduced small variant discovery errors by 34% and increased the number of structural variants detected per haplotype by 104% compared with GRCh38-based workflows, which enabled the typing of the vast majority of structural variant alleles per sample.


