2021-04-08 理化学研究所
理化学研究所(理研)開拓研究本部新宅マイクロ流体工学理研白眉研究チームの新宅博文理研白眉研究チームリーダー、小口祐伴研究員、尾崎由佳テクニカルスタッフ(研究当時)、マハメッド・ナディ・アブデルモエズ特別研究員(研究当時)の研究チームは、マイクロ流体技術により一つの細胞から核RNAと細胞質RNAを分画し、それぞれをナノポアシーケンサー[1]で解析し、RNAアイソフォーム[2](遺伝子ファミリー)の細胞内局在を定量する「ナノ1細胞分画解読法(NanoSINC-seq法)」を開発しました。
本研究成果は、1細胞におけるRNAアイソフォームの局在様式を明らかにし、それを制御する基盤情報を与えるものであり、医学・生物学への幅広い貢献が期待できます。
研究チームは2018年に、通常の次世代シーケンサー[3]を用いて、短く断片化した相補的DNA[4]を解析する「1細胞RNA分画解読法(SINC-seq法)」を開発しました。今回はこの手法を発展させ、相補的DNAを断片化することなく、全長の相補的DNAを解析することで、高精度にRNAアイソフォームを同定できるNanoSINC-seq法を開発しました。そしてこの方法により、細胞内の核膜を境界とする空間において、RNAアイソフォームの量が緻密に制御されていることを示しました。特に、核内のDNAからRNAが転写される際に生じる量的なゆらぎが、細胞質への輸送を経ることで増幅される場合と減衰される場合の2種類存在し、それぞれが細胞の機能と紐づいていることが明らかになりました。
本研究成果は、オンライン科学雑誌『Science Advances』(4月7日付:日本時間4月8日)に掲載されます。
背景
近年、「1細胞RNA-seq法[5]」を用いて、個々の細胞が持つRNAの種類とその量を調べ、それぞれの細胞が持つ個性(細胞の種類や生理状態)を理解することが可能になりました。しかし、従来の1細胞RNA-seq法は、1細胞レベルでRNAの量を計測する方法であり、重要な細胞小器官である核や細胞質におけるRNAの局在を詳しく調べることはできませんでした。
そこで、研究チームは1細胞の核RNAと細胞質RNAをマイクロ流体技術により分画し、それぞれを次世代シーケンサーで解析する「1細胞分画解読法(Single cell Integrated Nuclear and Cytoplasmic RNA-seq法;SINC-seq法)」を2018年に開発しました注1)。SINC-seq法では、分画した核RNAおよび細胞質RNAそれぞれから相補的DNAを合成し、従来の次世代シーケンサーの技術的制約から、それらを短いDNAに断片化した上で解析していました。
しかし、この方法では断片的な配列情報をつなぎ合わせて全長のRNA分子を推定する必要があり、類似した配列を持つRNAアイソフォーム(遺伝子ファミリー)を識別し、定量することは困難でした。今回は、SINC-seq法を発展させ、相補的DNAの断片化を経ることなく全長配列を得られる手法の開発に取り組みました。
注1)2018年6月6日プレスリリース「1細胞RNA分画解読法の開発に成功」
研究手法と成果
研究チームは、マイクロ流体技術を用いて、1細胞の細胞質RNAと核RNAを高精度に分画し、それぞれのRNA分子から合成した相補的DNAをそのまま解読する「ナノ1細胞分画解読法(Nanopore-based Single cell Integrated Nuclear and Cytoplasmic RNA-seq法;NanoSINC-seq法)」を開発しました。
NanoSINC-seq法は、ナノポアシーケンサーと呼ばれる長鎖のDNAを直接解読できる技術を活用した方式で、従来の次世代シーケンサーを用いた解析で不可欠だったDNAの断片化を経ることなく、長鎖のまま配列を解読できます。この方法により、構造がわずかに異なるRNAアイソフォームを特定しながら、1細胞の細胞質と核それぞれに含まれるRNAの定量が可能になりました(図1)。
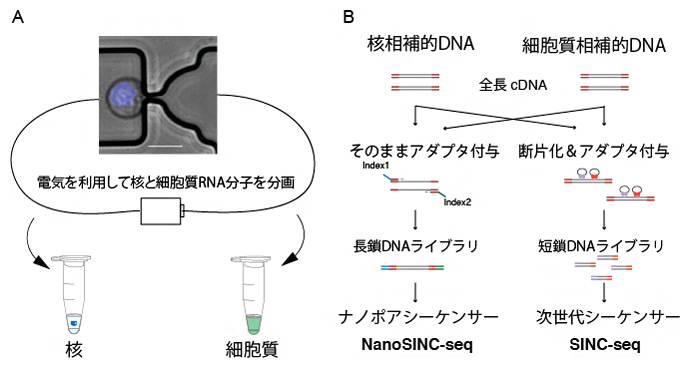
図1 従来のSINC-seq法と今回開発したNanoSINC-seq法の違い
(A)どちらの手法でも、まずマイクロ流体技術と電場を使って、細胞を核と細胞質に分画する。
(B)左のNanoSINC-seq法では、全長相補的DNAにアダプタを付与して、長鎖のままナノポアシーケンサーで配列を解読する。右のSINC-seq法では、相補的DNA分子を断片化する処理が不可欠である。
まず、NanoSINC-seq法を用いて得られたヒト細胞の1細胞レベルにおける細胞質と核のRNAアイソフォームデータから、同じRNAであっても細胞質と核とで、分子の長さや機能群が異なることを明らかにしました(図2)。また、同一の遺伝子ファミリーに属する異なるRNAアイソフォームが細胞質あるいは核のどちらかに多く存在するなど、局在状態の全貌を把握することに成功しました。
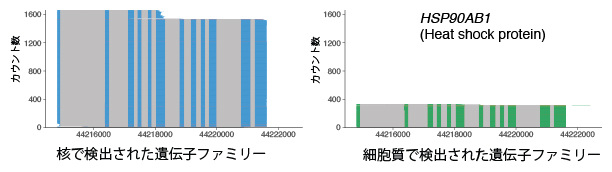
図2 NanoSINC-seq法で検出したRNAアイソフォーム
核で検出された遺伝子ファミリー(左)と細胞質で検出された遺伝子ファミリー(右)。同じ熱ショックタンパク質をコードする遺伝子(HSP90AB1)であっても、異なるRNAアイソフォームが核と細胞質のそれぞれに局在することが分かった。
さらに、NanoSINC-seq法で得られたデータを活用して、核が果たす役割を考察しました。真核生物は核内部でRNAを生産し(転写)、RNA分子を細胞質に輸送した後にタンパク質を合成します(翻訳)。核を覆う核膜は、いわば転写と翻訳の場を隔てる仕切りであり、RNAの量に対してこの仕切りが果たす役割を調べました(図3)。
RNAは1分子ずつDNAから転写されることから、その過程は確率的だと考えられ、RNAの量は細胞ごとにばらつくと推測されます。一方で、細胞が恒常性を維持するには、タンパク質、ひいては細胞質におけるRNAの量を安定的に保つ必要があると考えられます。そこで、核内のDNAからRNAが転写される際に生じる量的なゆらぎが、核膜を介して増幅されるかまたは減衰されるかについて遺伝子網羅的に解析したところ、増幅と減衰の両過程がともに存在することを突き止めました(図3)。
さらに、増幅と減衰に関わる遺伝子はそれぞれ異なる細胞機能と紐づいており、真核生物が核膜を介した制御を巧妙に活用している可能性が示されました。例えば、翻訳に関わるリボソーム[6]を構成するタンパク質に関連する遺伝子は核膜を介してばらつきが増幅する一方で、RNAスプライシング[7]に関連する遺伝子は減衰することが分かりました。これらの結果は、今回1細胞レベルかつRNAアイソフォーム解像度のデータを取得することで初めて明らかになりました。
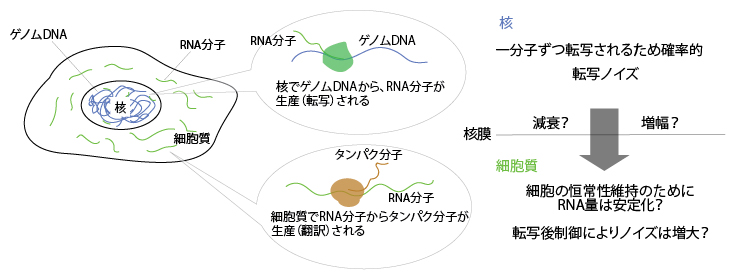
図3 真核生物におけるRNA分子の発現制御
真核生物の細胞では核においてRNA分子が生産(転写)され、それが細胞質に輸送された後にタンパク分子が合成(翻訳)される。核膜は転写の場と翻訳の場を物理的に隔てる仕切りであり、その仕切りをまたぐRNA分子の発現量制御を本研究で開発したNanoSINC-seq法により解析した。その結果、核内のDNAからRNAが転写される際に生じる量的なゆらぎ(転写ノイズ)が核膜を介して増幅する場合と減衰する場合の両方があることが分かった。
今後の期待
従来の1細胞RNA-seq法やSINC-seq法に加えて、今回開発したNanoSINC-seq法を利用することにより、核-細胞質の階層をまたぐRNA分子の転写後制御の理解が進み、将来的にはそれらの知見をもとに1細胞かつRNAアイソフォームレベルで遺伝子発現を効果的に制御することで、遺伝子治療や創薬などへ応用展開されることが期待できます。
補足説明
1.ナノポアシーケンサー
Oxford Nanopore Technologies社が開発したシーケンサー。ナノスケールの穴を持つタンパク質をDNA分子が通過する際の電気信号から塩基配列を読み取る方法。現在次世代シーケンサーとして広く用いられているIllumina社の読み取り方法と比較すると、DNA分子の断片化を経ることなく長鎖のまま解析ができることが特長。
2.RNAアイソフォーム
同じゲノムDNAの領域から転写されるRNA分子であり、基本的な配列は共通しているが、一部の領域(例えばエクソン)が含まれないなど、全体としては異なるRNA分子群。異なるRNAアイソフォームからは異なるタンパク質が翻訳され、タンパク質の多様性に寄与していると考えられている。
3.次世代シーケンサー
蛍光ヌクレオチドを使ったSequence by synthesis(シーケンス・バイ・シンセシス)に代表される手法を活用してDNAの塩基配列を読み出す装置であり、Sanger法と比較すると大幅に高いスループットを達成する。
4.相補的DNA
RNA分子から逆転写反応により合成された、RNA分子と相補配列を持つDNA分子のこと。
5.1細胞RNA-seq法
1細胞中に含まれるRNAを、ハイスループットDNAシーケンサーを用いて解読(塩基配列決定)し、網羅的かつ定量的にその量や種類を決定する方法。
6.リボソーム
翻訳というRNAの配列情報からタンパク質を合成する過程を担う細胞内構造。
7.RNAスプライシング
転写されたRNA前駆体の一部が除去されて、残りの部分が結合するRNAの転写後修飾過程を指す。
研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金挑戦的萌芽研究「単一細胞のRNAおよびDNA同時抽出・解析技術の開発(研究代表者:新宅博文)」、基盤研究(B)「マイクロ流体分画技術を用いた1細胞内構造体の泳動解析(研究代表者:新宅博文)」、内閣府革新的研究開発推進プログラム(ImPACT)「セレンディピティの計画的創出による新価値創造(プログラムマネージャー:合田圭介)」、日本学術振興会研究拠点形成事業「パンパシフィック・セレンディピティラボ(代表:合田圭介)」、ホワイトロック財団「セレンデピティラボを利用した科学者育成(代表:合田圭介)」の支援を受けて行われました。
原論文情報
Yusuke Oguchi, Yuka Ozaki, Mahmoud N. Abdelmoez, Hirofumi Shintaku, “NanoSINC-seq dissects the isoform diversity in subcellular compartments of single cells”, Science Advances, 10.1126/sciadv.abe0317
発表者
理化学研究所
開拓研究本部 新宅マイクロ流体工学理研白眉研究チーム
理研白眉研究チームリーダー 新宅 博文(しんたく ひろふみ)
研究員 小口 祐伴(おぐち ゆうすけ)
テクニカルスタッフ(研究当時) 尾崎 由佳(おざき ゆか)
特別研究員(研究当時) マハメッド・ナディ・アブデルモエズ (Mahmoud N. Abdelmoez)
報道担当
理化学研究所 広報室 報道担当