2019-11-04 理化学研究所
理化学研究所(理研)環境資源科学研究センター細胞機能研究チームの杉本慶子チームリーダー、バート・ライメン研究員(研究当時)、アリス・ランボレズ研修生らの国際共同研究グループ※は、植物細胞のリプログラミング[1]をクロマチン[2]レベルで制御する新たな仕組みを明らかにしました。
本研究成果は、組織培養技術を効率化し、有用植物の増産や育種、有用物質生産を進める技術開発に貢献すると期待できます。
今回、国際共同研究グループは、傷害ストレスによって細胞リプログラミングが誘導される際のクロマチン構造の変化と遺伝子発現の関係について調べました。シロイヌナズナ[3]の根において、傷害を与えてから数時間以内に発現する遺伝子領域を含むヌクレオソーム[2]のヒストン[4]H3タンパク質が傷害直後にアセチル化されるか、傷害前から既にアセチル化されていることが分かりました。さらに、このようなヒストンアセチル化[5]が、細胞リプログラミングを促進する「WIND1[6]」や「RAP2.6L[7]」などの転写因子[8]の発現誘導に必須であることを明らかにしました。また、アセチル基転移酵素[9]である「HAG1[9]」と「HAG3」が、傷害誘導性のヒストンアセチル化を触媒することを突き止めました。
本研究は、英国のオンライン科学雑誌『Communications Biology』(11月4日付け)に掲載されます。
背景
植物は傷害などのダメージを受けると、傷口付近の細胞をリプログラムすることで新たな組織や器官を再生します。例えば、挿し木に用いられる植物の茎では、切り口付近の細胞が新しく根の細胞としての分化運命を持つようリプログラムされて根を再生します。また、葉挿しによって増殖できる植物でも、葉の切り口から新たな葉や根を再生する際に、同様の細胞リプログラミングが起きます。
杉本チームリーダーらは、これまでに植物が傷口から新たな器官を再生する仕組みについて、分子レベルでの研究を進めてきました。2011年には細胞リプログラミングの鍵である転写因子WIND1の発見注1)、2017年にはWIND1を介して植物が茎葉を再生する仕組みの解明注2)などを発表してきました。
また、細胞の分化運命を制御するクロマチンレベルの仕組みについても研究を進め、2015年には、ヒストンメチル化[10]の一つであるヒストンH3タンパク質のテールと呼ばれる部分の27番目のリジンをメチル化する酵素複合体のPRC2[10]が、通常の発生過程で分化全能性の発揮を抑える役割を果たすことを解明しました注3)。
傷害によって誘導される細胞リプログラミングには、ダイナミックな遺伝子発現変化が伴います。このため、これらの遺伝子発現変化もクロマチンレベルでの制御を受けることが予想されますが、その具体的な仕組みは分かっていませんでした。
注1)2011年3月11日プレスリリース「植物細胞の脱分化を促進するスイッチ因子を発見」
注2)2017年1月17日プレスリリース「植物が傷口で茎葉を再生する仕組み」
注3)2015年6月30日プレスリリース「植物の分化全能性抑制の分子メカニズムの一端を解明」
研究手法と成果
国際共同研究グループはまず、シロイヌナズナの根を傷つけ、傷害による遺伝子発現の変化を、RNA次世代シーケンス解析[11]によって調べました。その結果、傷害を与えてから12時間以内に、3,665個の遺伝子の発現が誘導されることを見いだしました。
次に、これらの遺伝子領域が傷害を受ける前後にどのようなクロマチン構造を持っているのかを、クロマチン免疫沈降シーケンス法(ChIP-seq)[12]によって調べました。その結果、傷害を与えてから数時間以内に発現する遺伝子領域のヌクレオソームでは、ヒストンH3タンパク質テールの9番目と14番目のリジンが、傷害前からすでにアセチル化されているか(H3K9Ac、H3K14Ac)、もしくは傷害直後にアセチル化されることが分かりました(図1左)。一般にヒストンH3タンパク質のアセチル化は、遺伝子発現制御を促進することが知られています。これは、ヒストンアセチル基転移酵素がヒストンH3タンパク質にアセチル基を付加すると、ヒストンタンパク質とDNAの電気的な相互作用が減少してヌクレオソーム構造が緩み、RNAポリメラーゼ[13]がプロモーター領域[14]に結合しやすくなるためだと考えられています。
ChIP-seq解析で得られたデータセットを用いて、傷害誘導性の細胞リプログラミング因子をコードする遺伝子領域でのヒストンアセチル化の変化を調べました。傷害処理後いち早く発現誘導されるWIND1の遺伝子領域では、傷害前から既にアセチル化が起きていました。また、これに遅れて発現上昇するRAP2.6Lの遺伝子領域では、傷害誘導後にアセチル化が起きることが分かりました。
通常の発生過程では、多くのリプログラミング誘導因子の発現は、PRC2を介したヒストンのメチル化によって抑制されています。これらの遺伝子も傷害後数時間以内に発現しますが、ヒストンのメチル化には大きな変化は観察されませんでした(図1右)。
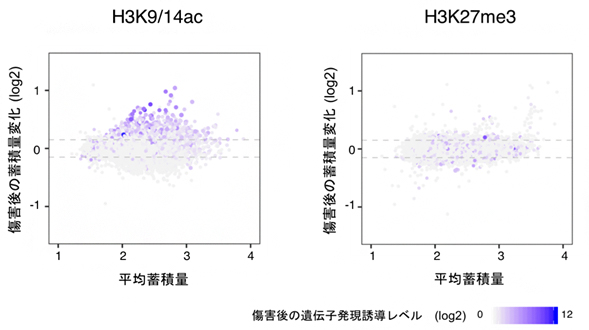
図1 植物に傷害を与えてから数時間後にみられるヒストン修飾の変化
- 左:傷害を与える前と比較すると、傷害後数時間でヒストンH3の9番目と14番目のリジンのアセチル化(H3K9/14ac)が著しく上昇し、それに伴って遺伝子発現も誘導される。
- 右:ヒストンH3の27番目のリジンのメチル化(H3K27me3)にはほとんど変化がみられず、遺伝子発現とも相関しない。
さらに、こうしたヒストンのアセチル化が、傷害によって誘導される細胞リプログラミングに必要であるかどうかを調べました。シロイヌナズナには12個のヒストンアセチル基転移酵素をコードする遺伝子があります。このうちHAG1とHAG3の機能を抑制したシロイヌナズナの変異体では、傷口からのカルス化[15]が抑制されました(図2)。このことから、傷害によって誘導される細胞リプログラミングには、HAG1とHAG3を介したヒストンのアセチル化が必要であることが分かりました。
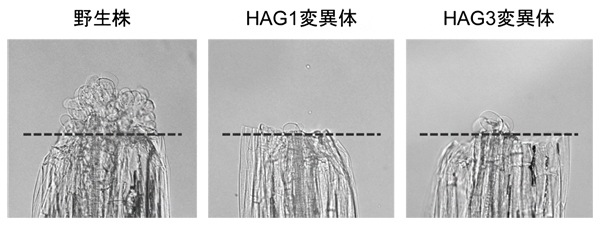
図2 ヒストンアセチル基転移酵素HAG1の機能を抑制した結果
ヒストンアセチル基転移酵素HAG1およびHAG3の変異体では、傷口からのカルス形成が抑えられた。破線は傷口を示す。
これらの解析から、ヒストンアセチル基転移酵素HAG1、HAG3は、WIND1やRAP2.6Lなどのリプログラミング制御因子の遺伝子領域でヒストンH3タンパク質をアセチル化し、傷害後にこれらの遺伝子発現を誘導しやすくすることで、細胞リプログラミングを促進することが明らかになりました(図3)。
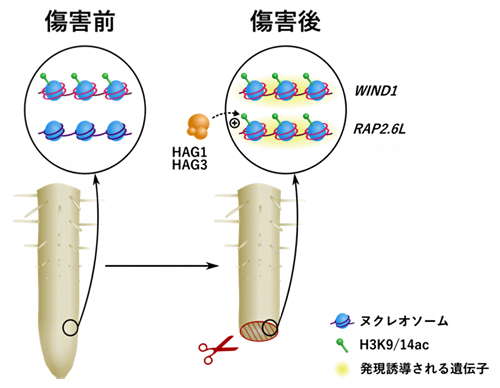
図3 ヒストンアセチル基転移酵素HAG1とHAG3によるリプログラミングの誘導の仕組み
ヒストンアセチル基転移酵素HAG1とHAG3は、WIND1やRAP2.6Lなどのリプログラミング制御因子の遺伝子領域でヒストンをアセチル化し、これらの遺伝子の発現を誘導する。
今後の期待
植物細胞のリプログラミングを引き起こすには、ヒストンのアセチル化によってクロマチン構造を変化させることが重要であることが今回示されました。これまで再生が困難であった植物種でも、ヒストンのアセチル化を人為的に操作することで効率よく細胞リプログラミングを誘導し、再生しやすくすることが可能になるかもしれません。
今回の研究は、組織培養技術の効率化による有用植物の増産や育種などを通じて、国際連合が2015年に定めた17項目の「持続可能な開発目標(SDGs)のうち、「2.飢餓をゼロに」及び「15.陸の豊かさも守ろう」に大きく貢献する成果です。
補足説明
- 1.細胞リプログラミング
分化した体細胞が発生運命を変えること。厳密な定義として、分化した細胞が発生過程で獲得したエピジェネティック修飾を初期化し、分化多能性を持った幹細胞へと変化することを示すが、最近では、細胞の脱分化、再分化、分化細胞から別の分化細胞への転換(ダイレクトリプログラミング)などを総称して用いられることも多い。 - 2.クロマチン、ヌクレオソーム
膨大な遺伝情報を小さな細胞核に収めるためには、DNAをコンパクトに収納する必要がある。DNAがヒストンタンパク質に巻き付いたものをヌクレオソームと呼び、このヌクレオソームがさらに凝集したものがクロマチンである。クロマチンの凝集が強い場合は、DNAに転写因子などが結合できず遺伝子の発現調節ができないが、凝集が弱い状態では、DNAがさまざまな因子の影響を受けやすくなる。このように、クロマチン構造は単にDNAをパッケージするためだけでなく、遺伝子の発現制御にも関与することが分かっている。 - 3.シロイヌナズナ
アブラナ科の植物の一つで、「ぺんぺん草」で知られるナズナの近縁種である。持っている遺伝子の総量が比較的少なく、発芽から開花し種子が採れるまでの時間が比較的短いことから、被子植物の代表として世界中で研究対象になっている。2000年に植物としては初めて全遺伝子の解読が終了したことから、特に分子生物学分野で活躍している。普遍的な生命現象の解明に用いられる代表的な生物をモデル生物と呼ぶが、これらの理由によりシロイヌナズナはモデル植物となっている。 - 4.ヒストン
DNAを巻きつけて核の中に収納するタンパク質がヒストンである。コアヒストンとしては、H2A、H2B、H3、H4の4種類がある。H2AとH2B、H3とH4が結合したものがさらに結合し8量体を形成する。それぞれのヒストンタンパク質が、メチル化、アセチル化、ユビキチン化などの化学修飾を受け、それがクロマチン構造などに影響を与え、遺伝子発現にも大きな変化を起こすことが明らかになりつつある。 - 5.ヒストンアセチル化
ヒストンにアセチル基が付加されるヒストン修飾のこと。ヒストンアセチル化が起こると、ヒストンとDNA間の電気的な結合が弱くなり、クロマチン構造が緩むと考えられている。この結果、転写因子など種々の因子がDNAへアクセスしやすくなり、遺伝子の発現などがコントロールされる。アセチル基をヒストンに付加するのは、ヒストンアセチル化酵素(histone acetyltransferase:HAT)である。逆に、ヒストンの脱アセチル化(アセチル化の除去)が起こると、クロマチン構造が凝縮することで、遺伝子発現制御が抑えられる。ヒストンからアセチル基を取り除くのは、ヒストン脱アセチル化酵素(histone deacetylase:HDAC)である。 - 6.WIND1
転写因子WOUND INDUCED DEDIFFERENTIATION 1の略称。WIND転写因子は理研植物科学研究センター細胞機能研究ユニット(研究当時)らによって2011年に発見され、現在も研究が進んでいる。傷ストレスによって傷口で発現が上昇し、植物細胞の脱分化と引き続いて起こる再分化にも関与することが分かっている。 - 7.RAP2.6L
RELATED APETALA2.6-Likeの略称。WIND1と同様にAP2/ERFファミリー転写因子の一つ。植物組織培養条件においてカルスからの茎葉再生を促進するほか、傷ついた組織が再び癒合する際に重要な機能を持っていることがこれまで報告されている。 - 8.転写因子
特定のDNA配列に結合し、遺伝子発現を制御するタンパク質の一群。 - 9.アセチル基転移酵素、HAG1
アセチル基転移酵素(アセチルトランスフェラーゼ)は、アセチル基を基質に転移する酵素。HAG1はHistone Acetyltransferase of the GNAT/MYST superfamily 1の略称で、ヒストンアセチルトランスフェラーゼの一つであり、ヒストンタンパク質にアセチル基を付与する。この酵素の遺伝子が壊れたり、阻害剤によって機能が抑えられたりすると、ターゲットとなるヒストンのアセチル化が起こらなくなる。 - 10.ヒストンメチル化、PRC2
ヒストンにメチル基が付加されるヒストン修飾のこと。メチル基が入る位置によって遺伝子発現に抑制的なクロマチン状態になったり、促進的なクロマチンになったりすると考えられている。例えば、ヒストンH3のテールと呼ばれる部位の27番目のリジンがトリメチル化される(H3K27me3)と、抑制的なクロマチン状態になり、その周囲の遺伝子発現制御は不活性化する。一方、ヒストンH3テールの4番目のリジンがトリメチル化される(H3K4me3)と、その周囲の遺伝子発現制御が活性化する。H3K27にメチル基を入れる酵素複合体としては、PRC2(Polycomb repressive complex 2)がよく知られている。 - 11.RNA次世代シーケンス解析
RNA-seqと略称される。次世代シーケンサーを用いて、ある細胞、組織で発現する遺伝子の種類と発現量を網羅的に捉える方法。RNAを抽出したのち、相補的なDNAに変換したものの配列を読むことで、ゲノムのどこの配列がどれくらい転写されていたかを見積もる。 - 12.クロマチン免疫沈降シーケンス法(ChIP-seq)
クロマチン免疫沈降法は、生体内におけるタンパク質とゲノムDNAの結合部位を検出するための実験手法である。ホルムアルデヒドによってDNAとDNAに結合しているタンパク質を架橋した後に、DNAの断片化を行い、タンパク質の抗体を用いてタンパク質-DNA複合体を回収する。さらに、DNAとタンパク質の架橋を解いてDNAのみを回収し、その配列を調べることでタンパク質がどのようなDNA配列に結合していたかが分かる。次世代シーケンスと組み合わせることで、ゲノム上のどこに、どれくらいの強さで結合していたかが分かる。ChIPは、Chromatin Immunoprecipitationの略。 - 13.RNAポリメラーゼ
RNAを合成する酵素。DNAの塩基配列をもとに、リボヌクレオチドを重合させてmRNAを合成する酵素は特にDNA依存性RNAポリメラーゼと呼ばれる。転写を担う実体であり、mRNAの情報をもとにアミノ酸が重合されタンパク質が合成される。 - 14.プロモーター領域
DNAがコードする情報(遺伝子)をmRNAに転写するには、読み取るDNAの読み始めを規定する必要がある。この転写の開始に関与する遺伝子の上流領域をプロモーター領域という。RNAポリメラーゼや転写因子などのタンパク質が結合することで、転写が調節される。 - 15.カルス化
植物科学分野では、もともと植物が傷口に作る不定形の細胞塊を示す言葉。癒傷(ゆしょう)組織ともいう。現在では、植物の組織培養で形成される不定形の細胞塊のことを広く示す。カルスが形成されることをカルス化という。
研究チーム
理化学研究所 環境資源科学研究センター
細胞機能研究チーム
チームリーダー 杉本 慶子(すぎもと けいこ)
研究員(研究当時) バート・ライメン(Bart Rymen)
(現フランスIBMP研究員)
テクニカルスタッフⅡ 河村 彩子(かわむら あやこ)
研修生 アリス・ランボレズ(Alice Lambolez)
(東京大学大学院 博士課程)
テクニカルスタッフⅡ 竹林 有理佳(たけばやし ありか)
研究員 岩瀨 哲(いわせ あきら)
研修生 坂本 優希(さかもと ゆうき)
(東京大学大学院 修士課程)
特別研究員 デイビッド・ファベロ(David S.Favero)
訪問研究員 池内 桃子(いけうち ももこ)
(日本学術振興会 特別研究員)
植物ゲノム発現研究チーム
チームリーダー 関 原明(せき もとあき)
客員研究員 佐古 香織(さこ かおり)
国立遺伝学研究所 エピゲノム研究室
教授 角谷 徹仁(かくたに てつじ)
助教(研究当時) 稲垣 宗一(いながき そういち)
中部大学 応用生物学部
准教授 鈴木 孝征(すずき たかまさ)
フランス国立農学研究所 植物発生学研究室
教授 フランソワ・ルディエ(Francois Roudier)
研究支援
本研究は、文部科学省 科学研究費補助金新学術領域「植物の成長可塑性を支える環境認識と記憶の自律分散型統御システム(領域代表者:木下俊則)」及び日本学術振興会(JSPS)科学研究費助成事業基盤研究B「植物の器官再生を制御する分子機構(研究代表者:杉本慶子)」、基盤研究C「傷害誘導性脱分化の分子ネットワーク解析(研究代表者:岩瀬哲)」による支援を受けて行われました。
原論文情報
Bart Rymen, Ayako Kawamura, Alice Lambolez, Soichi Inagaki, Arika Takebayashi, Akira Iwase, Yuki Sakamoto, Kaori Sako, David S. Favero, Momoko Ikeuchi, Takamasa Suzuki, Motoaki Seki, Tetsuji Kakutani, François Roudier and Keiko Sugimoto, “Histone acetylation orchestrates wound-induced transcriptional activation and cellular reprogramming in Arabidopsis”, Communications Biology, 10.1038/s42003-019-0646-5
発表者
理化学研究所
環境資源科学研究センター 細胞機能研究チーム
チームリーダー 杉本 慶子(すぎもと けいこ)
研究員(研究当時)バート・ライメン(Bart Rymen)
(現 フランスIBMP研究員)
研修生 アリス・ランボレズ(Alice Lambolez)
報道担当
理化学研究所 広報室 報道担当