2023-05-31 国立遺伝学研究所
腸内細菌群集はヒトの健康に重要なので研究が活発におこなわれています。これらの研究では、DNA抽出や配列データの情報解析などさまざまな解析手法が開発されています。こういった研究の活用で特に問題となるのが「腸内細菌群集の個人差」です。個人差のある研究対象について、個々の研究成果からより一般性の高い結論を得るためには、複数の研究から得られたデータを統合的に解析する「メタ解析」が重要になります。メタ解析をおこなう上で一番の課題は、異なる研究成果間の実験上のバイアスの存在であり、バイアスを定量するためには、群集を構成する細菌のメンバーリストや各メンバーの混合割合があらかじめわかったヒト腸内細菌群集の「模擬群集」が必要不可欠です。
今回、情報・システム研究機構 国立遺伝学研究所の森宙史准教授、理化学研究所の大野博司チームリーダーが率いる研究チームは、ヒト腸内細菌群集の模擬群集を作製し、その模擬群集を用いて様々な解析手法を評価しました。
本研究チームは、ヒト腸内細菌群集中で最も多く存在する細菌の種の中から、代表的な18種18株を選び等量混合して模擬群集を作成しました。この18株のDNAを定量するために、信頼性の高いqPCR法を開発しました。この模擬群集とqPCR法によるDNA定量法を用いて実際の各菌株の混合割合を定量した上で、ヒト腸内細菌群集の研究で広く用いられている10種類のDNA抽出法、7種類の16S rRNA遺伝子のプライマーセット、ショットガンメタゲノムの4種類の情報解析手法のそれぞれについて、菌株組成の推定結果に与える実験上のバイアスを評価しました。
本研究成果は、異なる解析方法で得られたヒト腸内細菌群集の系統組成データを比較解析するための重要な基盤データを提供し、新たな解析手法の開発を促進することが期待できます。
本研究は、日本医療研究開発機構 革新的先端研究開発支援事業(AMED-CREST) (JP19gm1010006)、革新的先端研究開発支援事業(PRIME) (JP19gm6010007)の支援を受けておこなわれました。
本研究成果は、国際科学雑誌「DNA Research」に2023年5月31日(日本時間)に掲載されました。
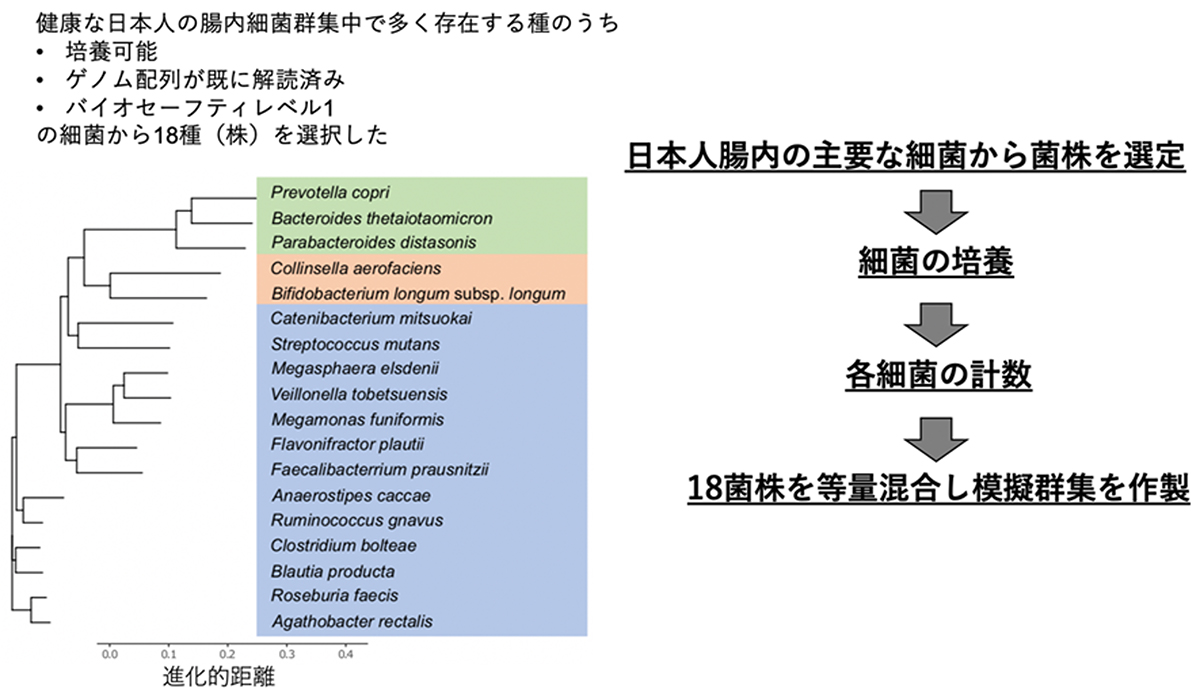
図: ヒト腸内細菌群集の模擬群集の作製方法。
系統樹の色枠は各系統が属する門を表しており、それぞれ緑枠がBacteroidota、橙枠がActinomycetota、青枠がBacillotaになります。
Assessment of metagenomic workflows using a newly constructed human gut microbiome mock community
Hiroshi Mori*, Tamotsu Kato*, Hiroaki Ozawa, Mitsuo Sakamoto, Takumi Murakami, Todd D. Taylor, Atsushi Toyoda, Moriya Ohkuma, Ken Kurokawa, Hiroshi Ohno
*equal contribution
DNA Research 2023 May 31 DOI:10.1093/dnares/dsad010