2023-09-20 ロスアラモス国立研究所(LANL)
◆リボソームとtRNAの相互作用の分子動力学をシミュレートし、抗生物質がtRNAの幾何学に影響を与え、リボソームが不正確なtRNAまたは全く受け入れないようにすることを示しました。これにより、細菌が死滅します。
◆この研究は、抗生物質の改良、薬剤耐性細菌との戦い、新しい抗生物質とワクチンの設計に道を開く可能性があります。
<関連情報>
- https://discover.lanl.gov/news/0919-drug-resistance-bacteria/
- https://www.nature.com/articles/s41467-023-40404-9#Abs1
リボソームの触媒中心に対するアミノアシルtRNAの幾何学的アライメントがmRNAの正確な解読を支える Geometric alignment of aminoacyl-tRNA relative to catalytic centers of the ribosome underpins accurate mRNA decoding
Dylan Girodat,Hans-Joachim Wieden,Scott C. Blanchard & Karissa Y. Sanbonmatsu
Nature Communications Published:11 September 2023
DOI:https://doi.org/10.1038/s41467-023-40404-9
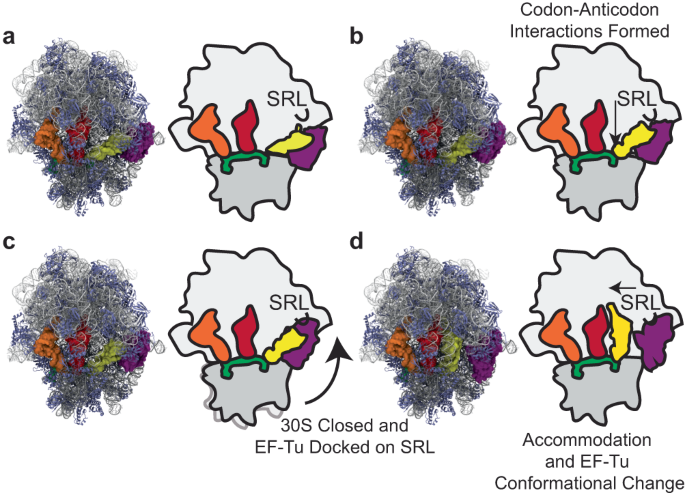
Abstract
Accurate protein synthesis is determined by the two-subunit ribosome’s capacity to selectively incorporate cognate aminoacyl-tRNA for each mRNA codon. The molecular basis of tRNA selection accuracy, and how fidelity can be affected by antibiotics, remains incompletely understood. Using molecular simulations, we find that cognate and near-cognate tRNAs delivered to the ribosome by Elongation Factor Tu (EF-Tu) can follow divergent pathways of motion into the ribosome during both initial selection and proofreading. Consequently, cognate aa-tRNAs follow pathways aligned with the catalytic GTPase and peptidyltransferase centers of the large subunit, while near-cognate aa-tRNAs follow pathways that are misaligned. These findings suggest that differences in mRNA codon-tRNA anticodon interactions within the small subunit decoding center, where codon-anticodon interactions occur, are geometrically amplified over distance, as a result of this site’s physical separation from the large ribosomal subunit catalytic centers. These insights posit that the physical size of both tRNA and ribosome are key determinants of the tRNA selection fidelity mechanism.