2023-09-19 東京大学定量生命科学研究所
中戸 隆一郎(定量生命科学研究所 大規模生命情報解析研究分野・准教授)
王 健康(大学院医学系研究科・博士後期課程(研究当時))
ルイス アウグスト エイジ ナガイ(定量生命科学研究所 大規模生命情報解析研究分野・助教)
長岡 勇也(定量生命科学研究所 大規模生命情報解析研究分野・特任研究員)
大庭 ジーナ未来(大学院新領域創成科学研究科/博士後期課程)
坂田 豊典(定量生命科学研究所 ゲノム情報解析研究分野・助教)
坂東 優篤(定量生命科学研究所 ゲノム情報解析研究分野・講師)
白髭 克彦(定量生命科学研究所 ゲノム情報解析研究分野・教授)
発表概要
【ポイント】
- 大規模なゲノム情報データを統合的に解析する新規立体構造解析手法CustardPyを開発しました。
- CustardPyを大規模データに適用したデータ駆動型解析により、遺伝子の発現制御に強く関わる立体構造部位を発見するなど、ゲノムの複雑な立体構造とその機能の一端を解明しました。
- 本研究の成果は、がんをはじめとした立体構造の破綻で引き起こされる様々な病気の原因解明に役立つと期待されます。
東京大学定量生命科学研究所の中戸隆一郎准教授と白髭克彦教授らによる研究グループは、ゲノム立体構造と遺伝子発現がどのように制御されているかを探る新規の情報解析手法”CustardPy”を開発しました。また、本手法を活用したデータ駆動型大規模ゲノム解析(注1)により、立体構造に関する様々な知見を獲得することに成功しました。
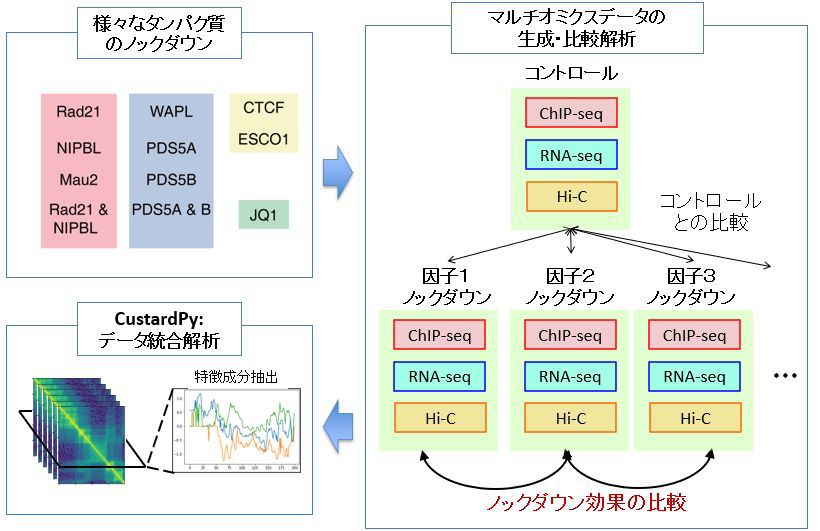
ゲノムの立体構造が正しく保たれることは、遺伝子の働きやエピゲノム(注2)、DNA複製など、ゲノムの多くの機能にとって非常に重要です。このゲノム立体構造の制御に重要な役割を持つタンパク質にコヒーシンがあります。コヒーシンの機能不全は急性骨髄性白血病などのがんやCdLSと呼ばれる先天性疾患の要因となることがわかっていますが、その具体的なメカニズムは明らかになっていません。本研究では、立体構造制御に関連するタンパク質群をノックダウンしたサンプルから様々なゲノム情報を観測し、統合的に解析する「大規模マルチオミクス解析(注3)」を実施しました(図1)
図1:本研究の概要
立体構造制御に関わるさまざまなタンパク質をノックダウンしたサンプルを生成し、マルチオミクスデータを生成します。得られたデータを統合的に解析し、因子ごとのノックダウンの影響の類似性を網羅的に評価するとともに、これらのデータ群をもとにゲノム領域を機能別に分類しました。
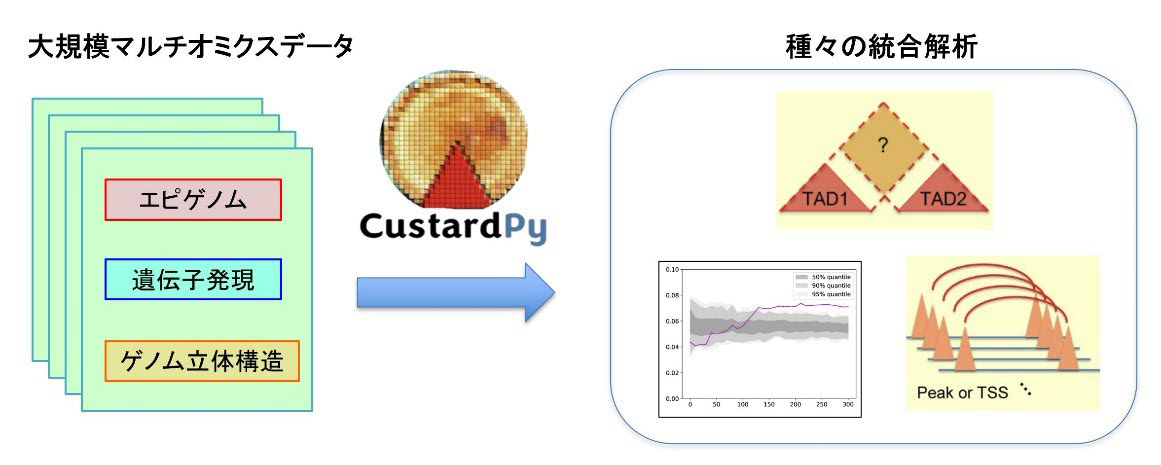
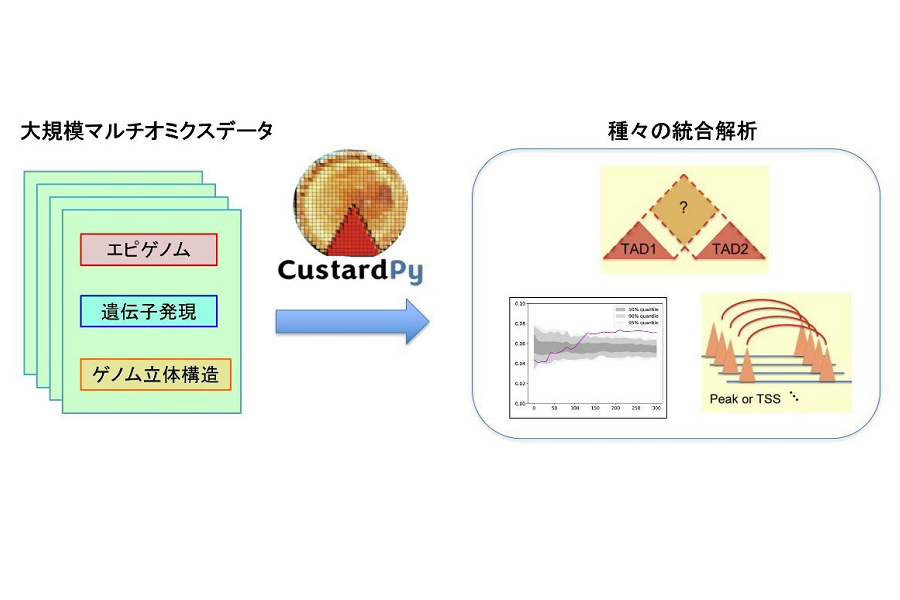
技術的に難度の高い大規模マルチオミクス解析を実施するため、新しい立体構造比較解析手法CustardPyを開発しました(図2)。解析の結果、遺伝子の働きに特に重要なゲノムの立体構造と、それをコントロールする因子、その領域のエピゲノムなどが明らかになりました。本成果は、これまで区別されていなかったコヒーシン関連因子群の詳細を理解し、これらの因子の変異が引き起こす疾患の原因解明に貢献することが期待されます。
図2 新しい立体構造比較解析手法CustardPy
発表内容
【研究の背景】
ゲノムの立体構造の適切な保持はゲノムの複製や遺伝子の転写などの機能にとって重要です。ゲノム立体構造に異常があると遺伝子の発現パターンに異常が生じ、結果としてがんなどの疾患の原因となります。近年開発されたHi-Cという手法を用いることで、ゲノム立体構造を網羅的に観測することができます。Hi-Cを活用したこれまでの研究によって、ゲノムは特定の構造(クロマチンループ(注4)、トポロジカルドメイン(TAD)(注5)、コンパートメント(注6))によって、細胞核内で規則的に折りたたまれていることがわかっています。その一方で、これらの構造が具体的に複製や転写にどのように作用しているのかについてはまだよくわかっていません。
この立体構造の制御に中心的な役割を果たす因子としてコヒーシンというタンパク質複合体が知られています。コヒーシンは、クロマチンループ形成、TAD形成、コンパートメント形成のためにそれぞれ異なる機能を果たします。つまり、コヒーシンはゲノム上で複数の役割を同時に果たしながら立体構造と遺伝子発現を制御しています。さらに、各役割において、それぞれ異なる他の因子と相互作用することが必要です。たとえばTAD形成ではCTCFというタンパク質と相互作用することが重要ですが、クロマチンループ形成ではCTCFを必要としません。このように、コヒーシンが複数の役割をどのように同時に果たしているのか、そこで相互作用する関連因子は互いに協調的に作用しているのかなど、その詳細な働き方はまだ完全には理解されていません。コヒーシンの変異による機能喪失は急性骨髄性白血病などのがんやCdLSと呼ばれる先天性疾患の要因となることがわかっており、コヒーシンの機能解明は治療法を確立するために重要となります。
【研究の内容】
Hi-Cを用いたこれまでの研究では限られた数の因子に焦点を当てていたため、関連因子群の全体像が把握できていないことが課題でした。そこで本研究では、コヒーシンとその関連因子群を広範囲に調査し、統一的な環境下での比較解析を行うことで、その機能の類似性・独立性を明らかにすることを目指しました。具体的には、それぞれの因子をノックダウンし機能欠失させたサンプルを作製し、それぞれのサンプルからエピゲノム・遺伝子発現・立体構造データから成るマルチオミクスデータを生成し、大規模なマルチオミクス比較解析を実施しました(図1)。本研究の重要な点は、(1)コントロール株とノックダウン株の比較によってノックダウンの影響を調べるのみならず、「因子間でノックダウンの影響の類似性を比較する」ことにより、関連因子群の協調的な機能・独立した機能を推定する点、(2)マルチオミクスデータ解析によって、立体構造の変化が遺伝子発現の変化と直接的な相関があるどうかを分析できる点にあります。
これらの解析を行う上では、多数のサンプルから生成されるマルチオミクスデータを横断的に解析し、影響を比較する情報解析手法が必要になります。この解析のため、本研究では新規のゲノム解析手法「CustardPy」を開発しました(図2)。このツールは、ノックダウンの影響を多サンプル間で比較し、類似度を調査できます。さらに、その影響のパターンに基づいてゲノムの特定のエリアをグループ化することができます。
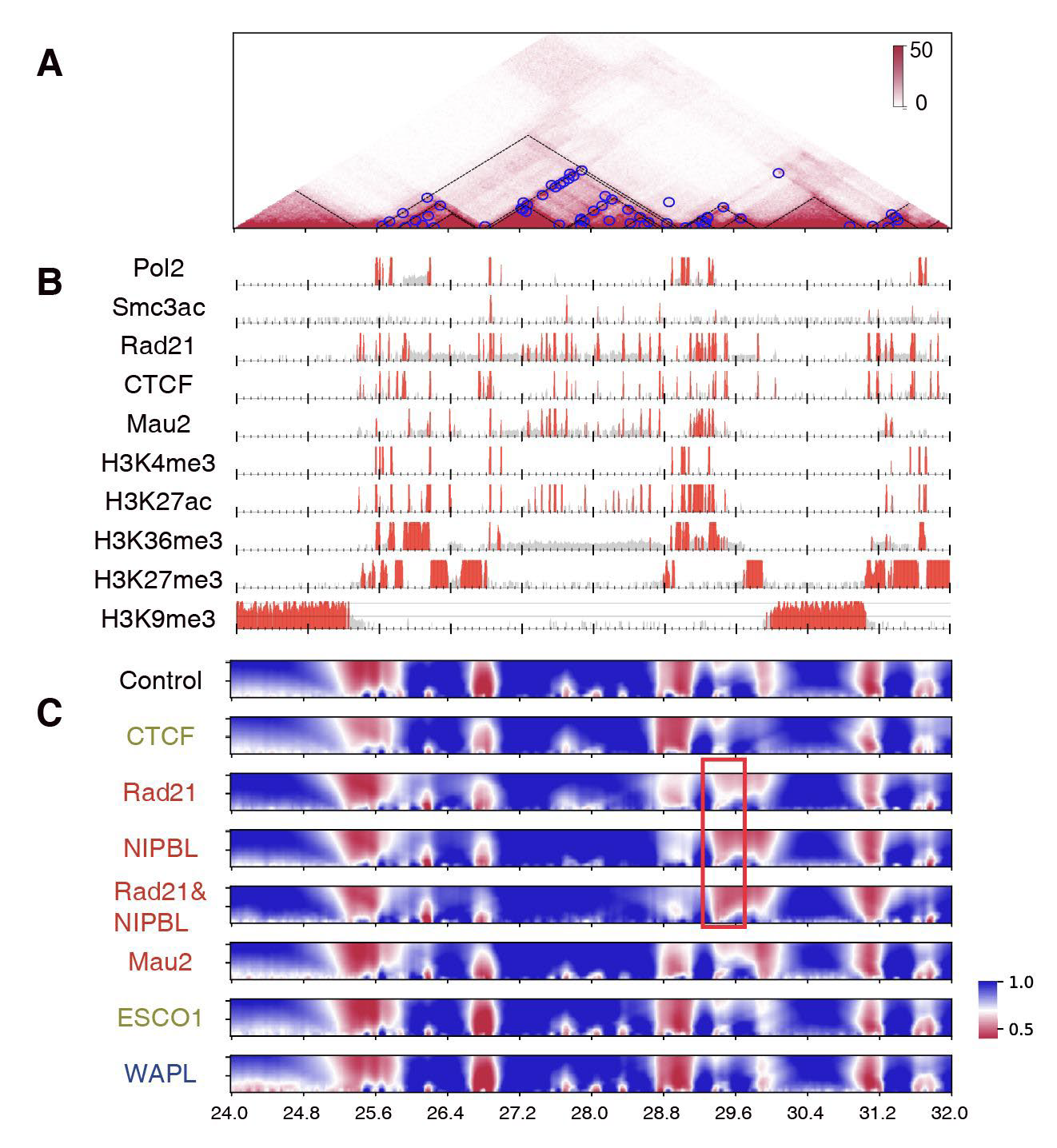
本研究の解析によって、以下のことを新たに発見しました(図3)。
・ノックダウンの結果生じるTADの分裂が、遺伝子発現の異常と強く関連していること。
・ 染色体の活性領域と不活性領域の間で、コヒーシンの存在量に顕著な差があること。
・TAD-TAD間立体相互作用(より遠距離の相互作用)では、近距離の立体相互作用と異なる機構が働いており、それはTAD内のエピゲノムと関連していること。
これにより、これまで不明だったゲノム立体構造の詳細な制御機構の一端が明らかになりました。
図3:統合解析の可視化の例(22番染色体, 24M塩基―32M塩基)
A:Hi-Cデータの可視化の例。赤い色が立体相互作用の強さを表します。黒の点線がTAD、青の丸がループを示します。
B:今回の対象とする因子のゲノム結合領域と、エピゲノム修飾状態の可視化。赤い領域に多く存在しています。
C:CustardPyが計算する立体構造の特徴量のひとつである「インシュレーションスコア」の可視化。青い領域がTAD内部、赤い領域がTADの境界を表しています。図の左部に示す因子がノックダウンされた因子を示します。このインシュレーションスコアを用いたクラスタリングにより、特定の因子に制御されるTAD境界を抽出することが可能です。
【今後の展望】
本研究で得られた知見は、コヒーシンによるエピゲノム・遺伝子発現・立体構造制御機構の詳細を明らかにするものであり、関連する疾患のメカニズム解明への貢献が期待されます。また、本研究で開発したCustardPyは、これまで難しかった多サンプルマルチオミクス統合解析を大きく前進させるものとなります。CustardPyはオンラインで利用でき、今後さらに多くのデータセットに適用して、知見の獲得を加速する予定です。
CustardPyの詳細はこちらで確認できます:https://custardpy.readthedocs.io/en/latest/
研究助成
本研究は、革新的先端研究開発支援事業(AMED-PRIME)(課題番号: JP23gm6310012h0004 研究代表者:中戸隆一郎)、科研費「基盤研究(B)(課題番号:23H02466 研究代表者:中戸隆一郎)」、「基盤研究(S)(課題番号:20H05686 研究代表者:白髭克彦)」、「学術変革領域研究(A)(課題番号:20H05940 研究代表者:白髭克彦)」、戦略的創造研究推進事業(JST CREST)(課題番号:JPMJCR18S5 研究代表者:白髭克彦)の支援により実施されました。
用語解説
(注1) データ駆動型大規模ゲノム解析:
大規模なゲノムデータを横断的に解析し、有意に現れる特徴を抽出・分析することで、これまでの知見に頼らずに新規の発見を行う解析手法です。
(注2) エピゲノム:
DNAのメチル化やヒストン修飾などのゲノム化学修飾の総称です。その細胞において発現する遺伝子群を制御するスイッチの役割を持ちます。
(注3) マルチオミクス解析:
同じサンプルから遺伝子発現量、エピゲノムと呼ばれる種々のゲノム修飾、立体相互作用などの異なる複数のゲノム情報を取得し、統合的に解析することでこれらのゲノム情報の相関を調査する手法です。
(注4) クロマチンループ:
物理的に近接して相互作用するゲノムの2つの部位です。
(注5) トポロジカルドメイン(TAD):
ゲノムの立体相互作用のまとまりとして定義される1Mbp程度のゲノム領域です。
(注6) コンパートメント:
Hi-Cデータに基づく主成分分析により、ゲノム全体をアクティブ領域・非アクティブ領域に分けたものです。それぞれAコンパートメント・Bコンパートメントと呼ばれます。
アイキャッチ画像
雑誌名等
雑誌名:Nature Communications
論文タイトル:Context-dependent perturbations in chromatin folding and the transcriptome by cohesin and related factors
著者:Ryuichiro Nakato*, Toyonori Sakata, Jiankang Wang, Luis Augusto Eijy Nagai, Yuya Nagaoka, Gina Miku Oba, Masashige Bando and Katsuhiko Shirahige*
DOI番号:10.1038/s41467-023-41316-4
URL:https://doi.org/10.1038/s41467-023-41316-4
問い合わせ先
東京大学定量生命科学研究所 高度細胞多様性研究センター 大規模生命情報解析研究分野
中戸 隆一郎(なかと りゅういちろう)