2018-03-22 理化学研究所
要旨
理化学研究所(理研)脳科学総合研究センター タンパク質構造疾患研究チームの大橋祐美子研究員(研究当時)、田中元雅チームリーダーらの国際共同研究グループ※は、溶液核磁気共鳴(NMR)法[1]を用いて、アミロイド[2]形成前のタンパク質の構造や揺らぎを測定し、タンパク質が異なる構造を持つアミロイドを生成する仕組みを明らかにしました。
アミロイドの生成は、プリオン病[3]やアルツハイマー病などの神経変性疾患[4]に関与します。アミロイドには構造の多様性があることが知られており、アミロイドの構造によって、それが細胞にもたらす毒性が異なることも報告されています。しかし、モノマー(単量体)であるタンパク質が多様な構造のアミロイドを生成する分子機構は、これまでに十分理解されていませんでした。
今回、国際共同研究グループは、NMR法などを用いた酵母プリオンタンパク質[5]「Sup35」の構造解析および酵母を用いた細胞表現型の解析を行いました。その結果、天然変性である酵母プリオンタンパク質のモノマ-における「揺らぎ」や「局所構造」が、最終的に生じるアミロイドの構造に大きな影響を与えることを発見しました。
原因タンパク質によるアミロイド生成が関与する神経変性疾患の多くは、治療法が確立されていません。本研究は、アミロイドが関与する神経変性疾患の治療に対して、モノマーの構造における揺らぎに着目した新たな薬剤の開発に寄与すると期待できます。
本研究は、米国の科学雑誌『Proceedings of the National Academy of Science of the United States of America』(3月6日号)に掲載されました。
※国際共同研究グループ
理化学研究所
脳科学総合研究センター タンパク質構造疾患研究チーム
チームリーダー 田中 元雅 (たなか もとまさ)
グローバル研究クラスタ 糖鎖構造生物学研究チーム
チームリーダー 山口 芳樹 (やまぐち よしき)
岐阜大学大学院 連合創薬医療情報研究科
教授 桑田 一夫 (くわた かずお)
デュッセルドルフ大学 物理生物学研究所
教授 ヘンリケ・ヘイセ(Henrike Heise)
背景
生命は、多種多様なタンパク質の働きにより維持されています。個々のタンパク質はそれぞれの機能を発揮するために、「天然構造」と呼ばれる構造を取る必要がありますが、まれに自己複製能を持つ線維状のタンパク質凝集体(アミロイド)へと構造変化します。その一部は、プリオン病やアルツハイマー病などの重篤な神経変性疾患に関与しています。アミロイド形成の引き金となるのは、原因タンパク質の濃度の上昇、細胞内外の環境の変化、タンパク質のアミノ酸が置き換わる変異などさまざまです。
アミロイドが関与する疾患では、プリオン病が古くから研究されており、「プリオン株」という現象が観察されてきました。プリオン株とは、同じプリオンタンパク質の凝集が引き起こす疾患であるにもかかわらず、病状や進行に大きな違いがみられる現象のことです。近年の研究の進展により、プリオン株は、原因タンパク質のアミロイド構造の違いにより、異なる細胞毒性を示したり異なる部位への沈着が誘導された結果として引き起こされることが示されています。しかし、モノマー(単量体)のタンパク質から、多様な構造のアミロイドが生じる分子機構は、これまでに十分理解されていませんでした。
そこで、国際共同研究グループは酵母プリオンタンパク質「Sup35」を用いて、各種の核磁気共鳴(NMR)法などから、アミロイド形成前のモノマーに対して、詳細な構造や「揺らぎ」を明らかにすることで、異なるアミロイド構造が生成する分子機構の解明に取り組みました。
研究手法と成果
酵母プリオンタンパク質Sup35は、アミノ酸685残基からなるタンパク質です。本研究では、アミロイド形成に関与しているN末端の天然変性領域[6]であるNドメイン(1-123アミノ酸)とMドメイン(124-253アミノ酸)(Sup35NM)のみを用いました。
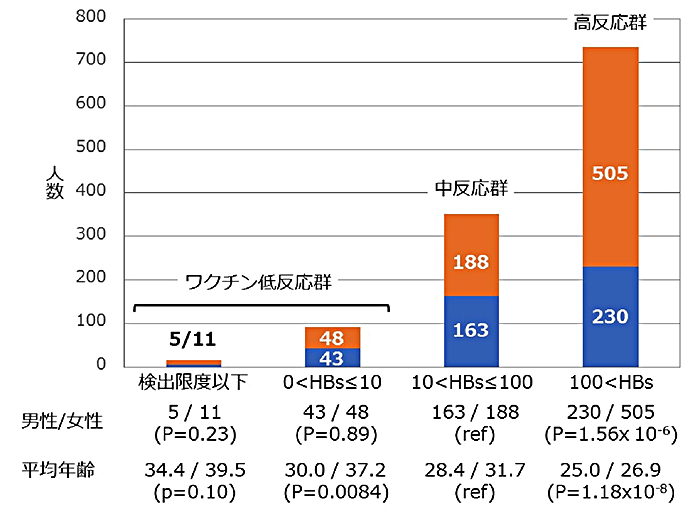
国際共同研究グループはまず、質量分析法[7]を用いたアミロイドコア領域の検出法を確立しました。その手法により、アミロイド構造の変化が誘導されるSup35NMアミノ酸変異を探索しました。以前の研究で、プリオン化した酵母の表現型に特徴的な変化がみられるSup35のアミノ酸変異が数種類報告されています。その中から、17番目のセリン(S)をアルギニン(R)に置換した変異体(S17R)のアミロイド構造が、天然型(WT)のSup35NMと大きく異なることを発見しました(図1)。WTのアミロイドコアはN末端に形成されるのに対し、S17R変異体のアミロイドコアは中心付近にありました。そのアミロイド構造を「PrD-C」と名付けました(図1)。
また、WTでもPrD-C構造を取り得るかを調べるため、WTモノマーにS17R 変異体アミロイドの種を加えた実験を行いました。すると、WTの速やかなアミロイド化が進行し、形成されたアミロイドの構造はPrD-C構造であることが確認できました。つまり、PrD-C構造はWTでも取り得る構造であることが示されました。
次に、WTとS17R変異体のアミロイド形成前の状態を、NMR法を用いて詳しく調べました。Sup35NMは特定の構造を取らない天然変性タンパク質であることが以前から知られています。しかし、局所的な構造の有無を調べることができる常磁性緩和促進法[8]を用いてその構造を調べたところ、Nドメインの30-120アミノ酸領域に「局所構造」が存在することが分かりました(図2)。さらに、速い時間スケールの構造揺らぎを観察できる1H-15N異種核NOE測定[9]でも、同様のことが示されました。しかしこれらの測定では、WTとS17R変異体間で、著しい違いは観察されませんでした。
続いて、NMR法の一つであるCLEANEX-PM[10]を用いて、モノマーのWTとS17R変異体において、アミド基の水素と溶媒中の水素の交換速度を見積もりました。この交換速度が速いほど、そのアミノ酸はより表面に露出していること、つまり、そのアミノ酸近辺の構造が緩んでいることを示します。その結果、S17R変異体が広範囲において、WTよりも表面に露出していることが分かりました。この結果は、Sup35NMの局所構造が、S17R変異体では緩くなっていることを示しています(図3左)。
また、飽和移動差NMR法[11]を用いて、アミロイド形成前の分子間相互作用の位置を推定しました。この測定はモノマーとオリゴマー[12]の混合状態の条件で行いました。オリゴマーの相互作用部位が45-90アミノ酸領域にあることは以前から知られており、その領域には明らかな相互作用が観察できました。そのオリゴマー相互作用に加え、WTではN末端側に、S17R 変異体ではC末端側に相互作用部位が観察されました。これらの相互作用部位が双方のアミロイドコア領域に含まれることから、相互作用部位は、アミロイド形成につながるモノマー同士の初期接触領域である可能性が示されました(図3右)。さらに、飽和移動差NMR法で示されたアミロイド形成の初期接触領域に変異を入れ、破壊することを試みました。WTとS17R変異体の初期接触領域はともに、アスパラギン残基を多く含むことが明らかになり、この領域のアスパラギン(N)をアラニン(A)に置換する変異を数種類作製しました。その結果、S17R/N100A/N109Aは、S17R変異を持つにもかかわらず、WT様のN末端コアを形成することを見いだしました。したがって、S17R変異体のモノマーにおけるアスパラギン100, 109のタンパク表面への露出が、PrD-Cコアを形成する上で重要な役割を果たしていることが分かりました。
これらの結果から、次のような結論を得ました。Sup35NMは二つの全く異なるアミロイド構造を取る能力が本来備わっています。しかしWTでは、天然変性であるにもかかわらず局所構造が存在します。そのため、PrD-C構造を作るための初期接触領域が露出せず、内側に隠れることによって、N末端コア領域を持つアミロイド構造を作りやすくなります(図4左)。一方で、S17R変異体ではモノマーの局所構造に緩みが生じ、その結果PrD-Cの領域が露出します。その結果、N末端の初期接触領域にあるアミノ酸であるセリン17の置換により、N末端コア領域を持つアミロイドが形成されにくくなることも影響し、N末コアよりもPrD-Cコアを持つアミロイド構造が優先して生成されます(図4右)。
今後の期待
原因タンパク質によるアミロイド生成が関与する神経変性疾患の多くは、治療法が確立されていません。治療を目指した研究では、アミロイドの生成を止めることが注目されがちですが、アミロイドの構造を、毒性の少ない構造に誘導するという方法も十分な効果が期待できます。
本成果は、今後、原因タンパク質の揺らぎを変化させる新たな薬剤の開発にも寄与すると期待できます。
原論文情報
Yumiko Ohhashi, Yoshiki Yamaguchi, Hiroshi Kurahashi, Yuji O. Kamatari, Shinju Sugiyama, Boran Uluca, Timo Piechatzek, Yusuke Komi, Toshinobu Shida, Henrik Müller, Shinya Hanashima, Henrike Heise, Kazuo Kuwata, and Motomasa Tanaka, “Molecular basis for diversification of yeast prion strain conformation”, Proceedings of the National Academy of Science of the United States of America, 10.1073/pnas.1715483115.
発表者
理化学研究所
脳科学総合研究センター タンパク質構造疾患研究チーム
研究員(研究当時) 大橋 祐美子(おおは しゆみこ)
チームリーダー 田中 元雅(たなか もとまさ)
報道担当
理化学研究所 広報室 報道担当
産業利用に関するお問い合わせ
理化学研究所 産業連携本部 連携推進部
補足説明
-
- 核磁気共鳴(NMR)法
- 静磁場におかれた原子核の共鳴を観測し、分子の構造や運動状態などの性質を調べる分光法。溶媒に分子を溶解させて計測する「溶液NMR法」や固体状態の分子を計測する「固体NMR法」などがあり、幅広い状態の試料を計測することができる。NMRはNuclear Magnetic Resonanceの略。
-
- アミロイド
- βシート構造に富んだ線維状のタンパク質の凝集体。界面活性剤やタンパク質分解酵素に抵抗性があるため、組織に蓄積しやすく、アルツハイマー病やプリオン病などのヒト疾患の原因となる。
-
- プリオン病
- 脳内にプリオンタンパク質の凝集体(アミロイド)が沈着し、神経細胞が脱落することで生じる、これまでに有効な治療薬のない神経変性疾患。プリオンタンパク質の凝集体を感染源として感染症を引き起こすことが特徴である。
-
- 神経変性疾患
- 神経組織の変性を伴う病気で、異常なタンパク質の凝集などを伴い、神経細胞が損傷するために起きると考えられている。アルツハイマー型認知症やパーキンソン病、プリオン病、筋萎縮性側索硬化症(ALS)などが知られている。
-
- プリオンタンパク質
- プリオンとはタンパク質からなる遺伝因子のことであり、プリオンを構成するタンパク質のことをプリオンタンパク質と呼ぶ。哺乳動物では、狂牛病などの原因となる哺乳動物プリオンタンパク質(PrP)が、出芽酵母では、Sup35やUre2といったタンパク質がプリオンタンパク質として知られている。
-
- 天然変性領域
- α-へリックスやβ-シートなどの二次構造を取らないアミノ酸領域を示す。タンパク質内に存在する天然変性領域は、その構造の可塑性を利用して、標的分子との結合に重要な役割を果たす。
-
- 質量分析法
- 原子や分子などを何らかの方法で気体状のイオンにして、そのイオンの質量を分析することにより、原子や分子の種類を推定することができる。また、そのイオンの検出量から、原子や分子の含有量を調べることができる。
-
- 常磁性緩和促進法
- 常磁性プローブを部位特異的に導入し、その近傍に存在するアミノ酸残基のNMRシグナルが緩和効果によって大きくブロード化することを利用して、常磁性プローブ近傍の構造を調べる手法。
-
- 1H-15N異種核NOE測定
- 1Hと15N核の核オーバーハウザー効果を測定することで、観測している分子の動きを調べる手法。
-
- CLEANEX-PM
- 溶媒の水とタンパク質に存在するアミドプロトンとの交換速度を測定することで、観測しているアミノ酸残基がタンパク質の内部に埋もれているか、タンパク質表面に露出しているかを調べる方法。
-
- 飽和移動差NMR法
- ラジオ波の照射によって飽和させた磁化が、相互作用のある結合標的分子に飽和移動されることで、タンパク質と結合している分子の信号強度が減衰することを利用して、タンパク質間やタンパク質-リガンド間の結合部位を調べる手法。
-
- オリゴマー
- 数分子以上のモノマー(タンパク質、糖、核酸など)が会合した重合体。
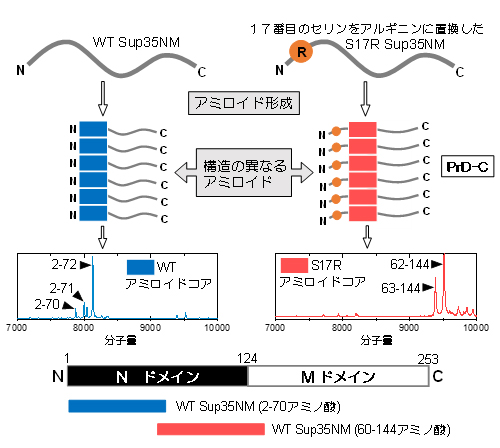
図1 Sup35NM S17R変異体のアミロイド構造の変化
Sup35NMの天然型(WT)とS17R 変異体のモノマー溶液から、それぞれ37℃でアミロイド形成させた。生成したアミロイドコア領域(アミロイドコア)を質量分析法により、分子量からコア領域の位置を調べた。その結果、WTのアミロイドコア(青)は2-70アミノ酸領域、S17R変異体のアミロイドコア(赤)は60-144アミノ酸領域の位置にあった。このように、WTとS17R変異体が、全く異なる領域にアミロイドコアを持つ(つまり異なる構造を持つ)ことを発見した。
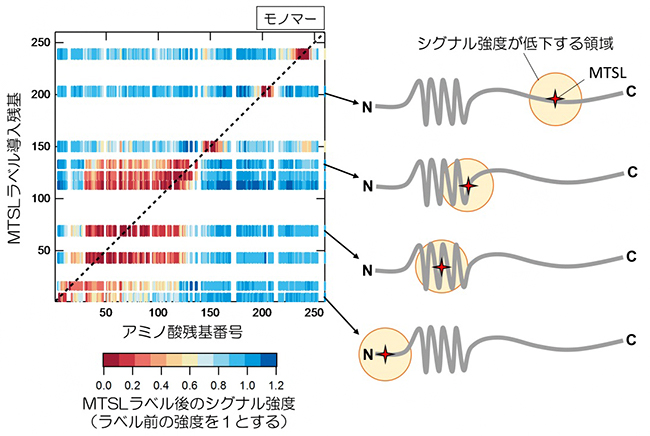
図2 天然変性Sup35NMにおける局所構造の発見
NMRの測定手法である常磁性緩和促進法を用いて、Sup35NMの構造を調べた。この方法は、常磁性体が近傍のアミノ酸残基のNMRシグナルの強度を下げることを利用し、Sup35NMの適当な箇所に入れた常磁性体(MTSL)ラベルによって、どのアミノ酸残基のシグナルが低下するかを観察することで、タンパク質の構造を調べることができる。その結果、30-120アミノ酸領域に常磁性体ラベルを入れたとき、広範囲のアミノ酸残基のシグナルが低下した。その結果、Sup35NMは、本来は特定の構造を取らない天然変性領域であるにもかかわらず、その領域に局所構造(コイル状構造)があることが分かった(右図上から3番目)。
図3 Sup35NMのWTとS17R 変異体のモノマー、オリゴマー構造の相違点
左)WTとS17R変異体のモノマーにおいて、アミド基の水素と溶媒中の水素の交換速度が、S17R変異体(赤線)で速くなっていることから、S17R変異体では局所構造に緩みが生じていることが示された。
右)飽和移動差NMR測定で、WTとS17R変異体のオリゴマーの構造を調べたところ、オリゴマー内で各モノマーは、それぞれ異なる領域で互いに相互作用していることが分かった(青四角と赤四角)。その相互作用領域(WTでは19-41アミノ酸領域、S17R変異体では91-118アミノ酸領域)がアミロイドコア領域(ATでは2-70アミノ酸領域、S17R変異体では60-144アミノ酸領域)に含まれることから、それらはアミロイド形成につながる初期接触領域であると考えられた。
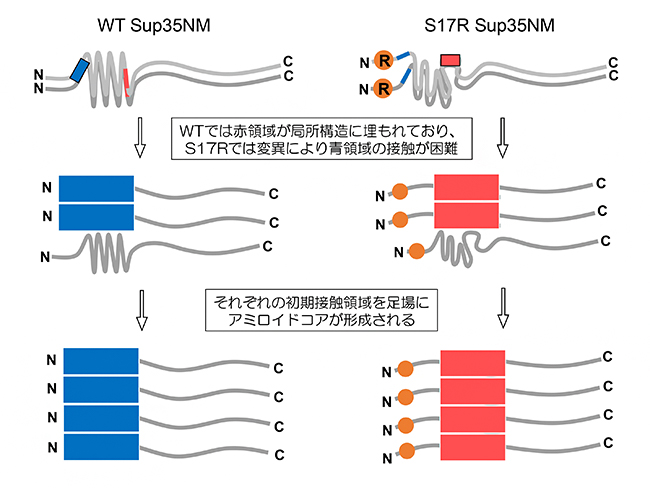
図4 Sup35NMのWTとS17R 変異体のアミロイド形成過程のモデル
Sup35NMのアミロイド形成は、初期接触領域を足場としてアミロイドのコア形成が進む。N末端の初期接触領域(青)はWTでは相互作用できるが、S17R変異体では変異により、相互作用が困難となっている。また、PrD-C接触領域(赤)は、WTでは局所構造に埋もれてアミロイド形成には使えないが、S17R変異体では局所構造に緩みが生じているため相互作用できる。このようにして、WTとS17R変異体で異なるアミロイド構造が生成する。