2022-02-17 東京大学
発表者
小田 有沙(東京大学 大学院総合文化研究科 広域科学専攻 助教)
太田 邦史(東京大学 大学院総合文化研究科 広域科学専攻 教授/東京大学 生物普遍性連携研究機構 教授)
発表のポイント
- 多くの遺伝子が関わる複雑な形質を高速で改良できるゲノム(注1)再編成誘発技術「TAQingシステム」(注2)を改良し、外来のDNAやRNAを一切導入せずにゲノム育種を行う新技術「TAQing2.0」を開発しました。
- 本技術では、細胞膜貫通ペプチドを用いてDNA切断活性をもつタンパク質を細胞内に導入し、大規模かつランダムにゲノム全体を再編成させることができます。
- 本技術により、生物の交配(有性生殖)が不能で通常の交配による育種が不可能な産業用微生物において、形質の効率的な改良が可能になりました。この技術を種々の生物に応用することで、従来得られなかった新しいタイプの作物品種や変異株を効率的かつ高速に取得することが可能になります。また、得られた改良株には外来DNAが一切含まれていないため、これまでの遺伝子組換え生物よりも社会的に受容されやすいと期待されます。
発表概要
東京大学は三菱商事ライフサイエンス株式会社と共同で、外部から遺伝物質を持ち込まずに生物のゲノムDNAを大規模に再編成し、生物機能の改良を加速する新技術「TAQing2.0」の開発に成功しました。
有用な農作物や発酵性微生物の改良には、古来有性生殖の仕組みを用いた交配という手法が用いられてきました。本技術では外部から遺伝子を導入することなく、既に存在する遺伝情報の組み合わせを変化させて新しい形質(性質や特徴)を生み出します。社会的にも受け入れられやすいゲノム改良技術であり人類の歴史で長く用いられてきた一方、掛け合わせによって子孫を残す能力を失った生物が一部に存在しこれらの生物では交配による育種は不可能でした。
本研究では、細胞にDNA切断酵素を直接導入することで、有性生殖能を欠く生物種でも、自然界の交配の際に生じるゲノム再編成過程と似た大規模ゲノム再編成を誘発することが可能になりました。
TAQing2.0を用いることで、これまで交配による育種が困難であった工業用微生物についても、高い効率で自然変異に近い形のゲノム改良が可能となります。また、感染症創薬研究という観点からは、抗生物質産生菌にTAQing2.0を適用することで、天然物の合成に関わる休眠遺伝子の活用が可能になり、新規の抗生物質リード化合物の創製が可能になります。これは将来の新興・再興感染症への対抗手段の一つとなることが期待されます。
発表内容
古来、人類は有性生殖の仕組みを利用して、人類にとって有用な性質をもつ品種を取得してきました。交配育種では、外部から遺伝子を導入することなくDNA(ゲノムDNA)を変化させ、有用形質を獲得していきます。この際のゲノムDNAの変化は世代交代を経て時間をかけて行われるため、生物の改良には非常に長い期間がかかるという根本的な課題がありました。また、有性生殖の機能を失った生物については、交配育種は用いることができず、放射線や薬剤処理によって多大な時間や労力をかけて有用形質を獲得するしかありませんでした。今回開発したTAQing2.0技術によって、これらの課題を克服することに成功しました。
本研究チームでは、これまでにゲノム再編成を誘発する技術であるTAQing技術を開発していました(Muramoto et al., Nature Commun. 2018)。この技術では、酵母細胞や植物(シロイヌナズナ)の細胞にDNA切断酵素を導入して同時多発的に細胞内のDNAを切断します。これにより、ゲノムDNAの大規模な再編成が誘発され、それに伴いバイオエタノール発酵性能などの有用形質を迅速に改良できることが明らかになりました。しかし、当初のTAQing技術では、DNA切断酵素の遺伝子をプラスミドDNAなどにより外部から導入する必要がありました。したがって、遺伝子導入系が確立されていない生物種に適用できないことや、得られた変異株は遺伝子組換え生物として取り扱う必要があるなど、実用化の際の課題がありました。
今回の実験では、有性生殖能を失った産業用酵母であるトルラ酵母(注3)の細胞を用いました。特定のDNA配列でDNAを切断する酵素(制限酵素(注4)TaqIやMboIなど)を、汎用的な膜透過性ペプチドと混合し、緩衝液中のトルラ酵母細胞に作用させるだけで、細胞内でゲノムDNA切断を引き起こし、大規模なゲノム再編成を誘発することに成功しました(図1)。さまざまな条件を検討し、細胞前培養の時期、緩衝液条件などの最適化(塩濃度、pHの範囲の絞り込みや双性イオン性緩衝液の利用など)により、細胞壁を維持したまま、種々のタンパク質や酵素を迅速に酵母細胞内に送致する方法も同時に開発しました。この技術を利用することで、凝集性などの表現型変化を伴う変異株が複数取得されています。得られた変異株について、1分子DNAシーケンサーなどを用いて全ゲノム解析を行ったところ、実際に導入したDNA切断酵素の切断配列部位で、ゲノム再編成が生じている証拠も得られました(図2)。また、この成果を得る過程で、これまで大まかにしか明らかになっていなかったトルラ酵母の全ゲノム配列を正確に決定することにも成功しました。
本研究チームはこの改良型技術をTAQing2.0と名付け、現在、他の生物種に適用を拡大しているところです。TAQing2.0により、新しい有用形質をもつ生物の育種の可能性拡大や、新規抗生物質の探索に寄与することが期待されます。
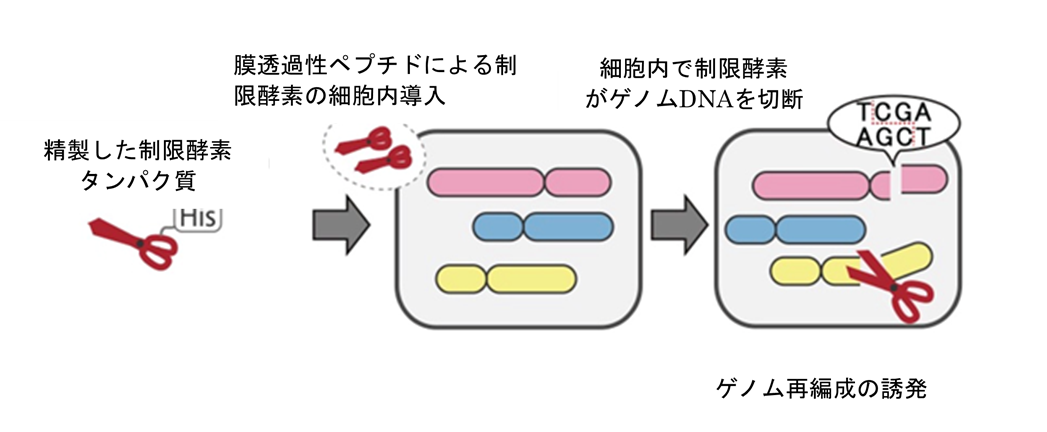
図1. TAQing2.0システムの概要
従来のTAQingシステムでは制限酵素遺伝子をもつプラスミドDNAを細胞に導入して細胞内で発現し、DNAの切断を細胞内で部分的に誘導します。TAQing2.0では精製した制限酵素タンパク質を膜透過性ペプチドと混合し、細胞内に直接送致します。そのため、外来DNAの取り込みを行わずにゲノム再編成を誘発することができます。また、遺伝子発現系が確立していない生物にも容易に適用できるため、適用範囲が広がります。
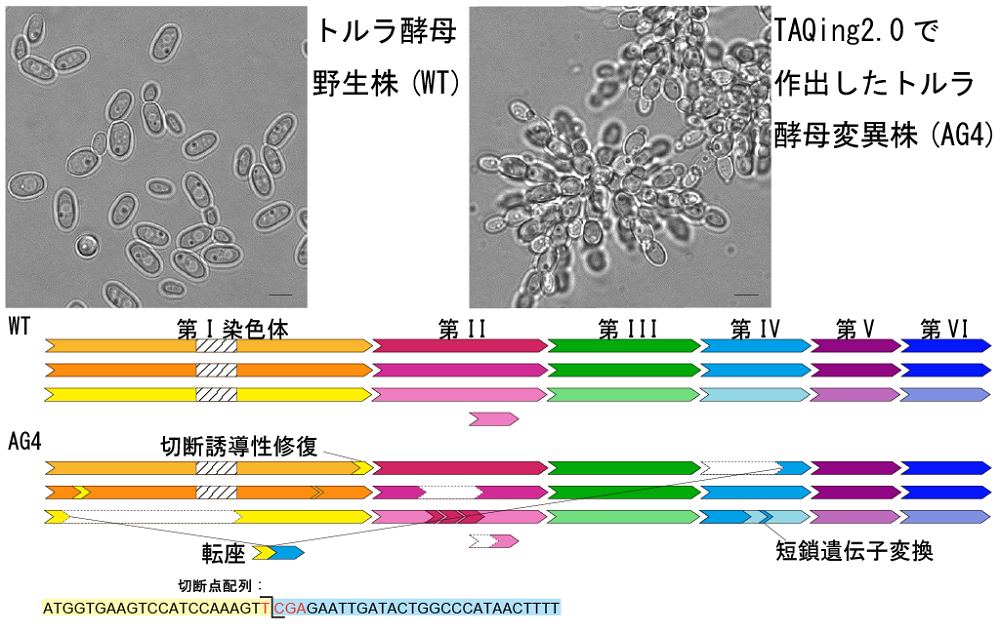
図2. TAQing2.0で作出したトルラ酵母変異株(左上、野生株;右上、変異株AG4)の顕微鏡像(写真、スケールバー:5 µm)と染色体構成(下段模式図)
今回開発した方法では、写真のように細胞壁を維持したまま任意のタンパク質を細胞内に導入できます。TAQing2.0をトルラ酵母に適用すると、凝集性やストレス耐性などの表現型が変化した細胞株が容易に取得できます。なお、写真で示したAG4では第I染色体と第IV染色体の間で転座(染色体の切断・再結合)が生じています。転座の切断点には、DNA切断酵素の認識配列が存在していることが示されています。
用語解説
(注1)ゲノム
ある特定の生物種を記述する最小単位のDNA情報。細胞一つ一つにゲノムの情報をもつDNA(ゲノムDNA)が格納されています。
(注2)TAQingシステム
2018年に報告された生細胞内に多部位でDNAを切断する制限酵素などのタンパク質を導入し、ゲノム再編成を同時多発的に誘発する技術(Muramoto et al., Nature Commun. 2018)。TAQingシステムでは、酵母細胞のバイオエタノール発酵性能や、シロイヌナズナなどの植物のストレス耐性やバイオマス量などの形質を迅速に改良することができます。
(注3)トルラ酵母
トルラ酵母(Candida utilis)は、第一次世界大戦中にタンパク質栄養源の確保のためにドイツで利用されるようになり、現在では食飼料用酵母や栄養補助食品として利用されています。また、その安全性は米国FDA(Food and Drug Administration:食品医薬品局)がGRAS(Generally Recognized As Safe)認定しており、世界各国で数多くの食品に利用されています。
(注4)制限酵素
4塩基あるいは6塩基、またはそれ以上の長さのDNA塩基配列を特異的に認識してDNAを切断する酵素。一部のバクテリアに存在し、バクテリアに感染するファージ(バクテリアに感染するウイルス)の増殖を制限する働きがあるため、制限酵素と呼ばれるようになりました。制限酵素は決まった配列でDNAを切断する性質があるため、組換えDNA技術の発展に不可欠な貢献を果たしました。
論文情報
Taishi Yasukawa, Arisa H. Oda, Takahiro Nakamura, Naohisa Masuo, Miki Tamura, Yuriko Yamasaki, Makoto Imura, Takatomi Yamada, and Kunihiro Ohta*, “TAQing2.0 for genome reorganization of asexual industrial yeasts by direct protein transfection,” Communications Biology: 2022年2月17日, doi:10.1038/s42003-022-03093-6.
論文へのリンク (掲載誌)