ヒト乳がん組織の3次元かつ高解像度な生物学的解釈が可能に
2022-03-09 東京大学,聖マリアンナ医科大学
発表のポイント
◆組織切片画像から空間上の遺伝子発現量を予測する深層学習モデルDeepSpaCEを開発し、ヒト乳がん組織切片での予測を実験的に証明しつつ、隠れた組織学的な特徴を見出すことに成功しました。
◆少数の空間トランスクリプトーム実験データを元にDeepSpaCEを用いて学習・予測することで、周辺の組織切片画像から3次元かつ高解像度な遺伝子発現解析が可能であるという新規概念を実証しました。
◆高額な実験コストを抑えて空間上の詳細な遺伝子発現プロファイリングを可能にしたことにより、世界中の研究者が組織切片の構造や機能をよりよく理解して、生物学的発見を加速させると期待されます。
発表概要
東京大学大学院新領域創成科学研究科の門城拓 博士課程学生(研究当時)、永澤慧 特任研究員、鈴木穣 教授、鎌谷洋一郎 教授、東京大学医科学研究所の小井土大 特任助教による研究グループは、組織切片画像から空間的な遺伝子発現量をコンピューター上で予測する、深層学習(注1)を応用した新規手法DeepSpaCEを開発しました。
病気の検体を採取・染色して顕微鏡でその特徴を見極める病理学的検査は、一般診療でがんの検査などに用いられています。近年、空間トランスクリプトーム(注2)と呼ばれる、これまではわからなかった病理画像上の場所ごとの遺伝子発現を網羅的に測定する手法が開発されました。この手法はNature Methods誌の2020年のMethod of the Yearにも選ばれるなど、がんなどの病気の本質に迫る測定技術として世界から注目されています。しかし、現在主流の方法では高価な試薬と高度な実験技術が必要とされ、組織切片全面の遺伝子発現量を測定できない(空間解像度が低い)といった課題が存在します。
研究グループはDeepSpaCEによる空間トランスクリプトームの超解像度化や3次元補完による課題解決法を考案し、実際のヒト乳がん組織切片を用いてその有用性を示しました。本手法により、組織学や病理学の専門家でない研究者も、遺伝子アノテーション(注3)を介して組織切片画像を詳細に解釈可能となり、がん研究をはじめとしたさまざまな分野への応用が期待されます。
本研究成果は、英国科学誌「Scientific Reports」に3月8日(火)に公開されました。
発表内容
研究の背景
ゲノム科学の進歩に伴い、以前は取得することができなかった病気に関する新たなデータを得ることができるようになり、将来的な医療への応用可能性が期待されています。空間トランスクリプトームはそのような技術の一つで、組織切片上の場所ごとの遺伝子発現量を網羅的に測定することのできる新しい技術です。しかし、以下の3つの課題がその活用の妨げになっています。
(1)1回の実験に100万円前後のコストがかかり、1つの研究室で何十枚もの組織切片に対して空間トランスクリプトームデータを取得するのは困難である。
(2)現在普及している技術では、組織切片上の遺伝子発現量を測定するスポット(直径55μmの円)と隣のスポットの間に隙間が空いており、空間解像度が限られる。
(3)組織切片を扱う熟練した手技があっても、稀に実験エラーにより発現量が測定できない領域が生じてしまう。
そこで研究グループは、ヘマトキシリン・エオジン(H&E)染色 (注4)画像が細胞の形態的特徴や状態を反映しうることに着目し、AI(人工知能)の一種である畳み込みニューラルネットワーク(CNN、注5)を用いてH&E染色画像上の任意の箇所における遺伝子発現量を予測する手法を考案し、上記課題の解決を目指しました(図1)。
図1:DeepSpaCEはH&E染色画像を入力として空間的な遺伝子発現量を予測する。応用事例1では遺伝子発現量の測定スポット間の隙間を予測により埋めて超解像度化(解像度の向上)する。応用事例2では連続組織切片の遺伝子発現量を3次元的にインピュテーション(補完)する。
研究内容
研究グループは、H&E染色画像を入力、空間的な遺伝子発現量を出力とした深層学習モデルDeepSpaCE(Deep learning model for Spatial gene Clusters and Expression)を開発しました。DeepSpaCEでは、空間トランスクリプトームにおいて実験スポットの隙間や実験エラーにより発現量が得られなかった箇所の遺伝子発現予測(超解像度化)と、空間トランスクリプトーム実験を行っていない周辺の連続切片全体の予測(インピュテーション)、この2つの応用解析が可能です(図1)。
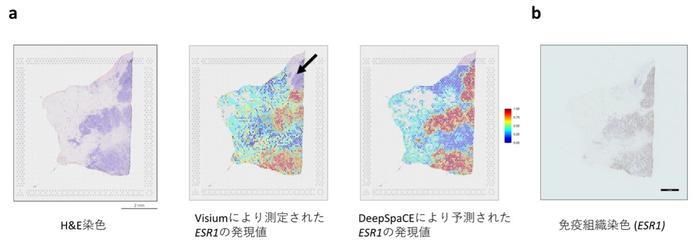
はじめに、ヒト乳がん組織の連続切片を用いた交差検証(注6)によりDeepSpaCEの予測精度を確認しつつ、実験エラーにより遺伝子発現量が測定されなかった領域の遺伝子発現量を補完できることを確認しました。実際に、乳がんで重要なESR1(エストロゲン受容体1)の予測された遺伝子発現パターンは、そのタンパクレベルの量を示す免疫組織化学染色(注7)を結果とよく一致しました(図2)。
また、がんの浸潤マーカー遺伝子であるSPARC(Secreted protein acidic and rich in cysteine)の発現プロファイルを予測した結果、空間トランスクリプトームの実験では空間解像度が低いために特定困難であった小さな腫瘍浸潤部位をはっきりと視認することが可能になりました(図3)。さらに、遺伝子発現情報のないH&E染色画像の情報も加えて学習する半教師あり学習(注8)を用いることによって、遺伝子発現量の予測精度が高まる(SPARCの例では相関係数が0.509から0.616に増加)ことを見出し、少数の空間トランスクリプトームデータを用いた際にもより正確な予測を実現できることを示しました。
図2:(a)左は組織切片のH&E染色画像。中央は空間トランスクリプトーム技術により測定されたESR1の発現値で、右上の黒矢印は、実験エラーが生じて発現量が測定されていない領域を示す。右はDeepSpaCEにより予測されたESR1の発現値。
(b) 組織切片の隣接切片におけるESR1の免疫組織染色によるタンパクレベルでの予測精度の検証。
図3:SPARCの超解像度化
(a)左列はH&E染色画像。中列は DeepSpaCEによるSPARC発現量の予測値。右列はDeepSpaCEの予測結果をH&E染色に重ねた画像で、上行は元のスポット、中行は予測されたスポット、下行は元のスポットと予測されたスポットの両方を示す。黒丸は、H&E染色画像では色調が類似しているが発現量は大きく異なる領域を示す。
(b)(a)右列の拡大画像。H&E染色の形態上の特徴から判断されるがん細胞の浸潤(青矢印)と微小環境(赤矢印)において、超解像度化されたSPARC発現パターンがその境界を明瞭に示した。
本研究成果が社会に与える影響
研究グループは、空間トランスクリプトーム技術における課題を克服するためにDeepSpaCEを提案・開発しました。応用事例としてヒト乳がん組織の発現パターンを超解像度化し、隠れた組織学的な特徴を見出し、実験的にその妥当性を証明しました。本手法により、未測定あるいは実験エラーの生じた空間トランスクリプトームを予測により補うことが可能になり、追加実験コストの大幅な削減が可能になりました。さらに、組織学や病理学の専門家でない研究者も、H&E 染色画像から得られる情報を、空間的遺伝子発現プロファイルと遺伝子アノテーションを介して解釈可能となり、生物学的発見に貢献することが期待されます。
DeepSpaCEは誰でも利用可能なツールとして、公開リポジトリであるGitHub上で公開しています(https://github.com/tmonjo/DeepSpaCE)。DeepSpaCEがさまざまな分野で広く使用されることにより、空間トランスクリプトーム研究が加速することが期待されます。
発表雑誌
雑誌名: 「Scientific Reports」(オンライン版:3月8日)
論文タイトル:Efficient prediction of a spatial transcriptomics profile better characterizes breast cancer tissue sections without costly experimentation
著者: Taku Monjo†,Masaru Koido†*,Satoi Nagasawa,Yutaka Suzuki,Yoichiro Kamatani*
†共同第一著者 *責任著者
DOI番号:10.1038/s41598-022-07685-4
アブストラクトURL:https://www.nature.com/articles/s41598-022-07685-4
発表者
門城 拓 (東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 生命システム観測分野 博士課程3年(研究当時))
小井土 大(東京大学医科学研究所 癌・細胞増殖部門 人癌病因遺伝子分野 特任助教)
永澤 慧 (東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 生命システム観測分野 特任研究員/聖マリアンナ医科大学 乳腺内分泌外科教室 非常勤講師)
鈴木 穣 (東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 生命システム観測分野 教授)
鎌谷 洋一郎(東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 複雑形質ゲノム解析分野 教授)
用語解説
(注1)深層学習
AI(人工知能)の一種で画像認識・機械翻訳等に広く使用される。生物の神経回路を模したニューラルネットワークを多層に重ねて学習・予測を行う。
(注2)空間トランスクリプトーム
組織切片上の空間的な遺伝子発現量を網羅的に測定する技術。 本研究では大量のスポットが等間隔に並んだチップを用いて、in situ capturing技術により組織標本内の位置情報と遺伝子発現量が対応できるVisiumプラットフォーム(10x Genomics社)を使用。
(注3)遺伝子アノテーション
これまでの知見に基づいてデータベースに登録されている遺伝子の構造や機能などのさまざまな注釈情報。
(注4)ヘマトキシリン・エオジン(H&E)染色
細胞核をヘマトキシリンが青色に、核以外の成分をエオジンが赤色に染める標準的な染色手法。
(注5)畳み込みニューラルネットワーク(CNN)
画像認識でよく用いられているニューラルネットワークの一種。画像の局所的な情報(例えば3×3ピクセル)の重み付き和を計算して新たな特徴量を作り出す”畳み込み”を画像全体に渡って実施する。この畳み込みを含む処理を何度も繰り返した深層CNNモデルは、しばしば人間と同等かそれ以上の画像認識に成功してきた。本研究では畳み込み層を含む合計16層からなる深層学習モデルVGG16を使用。
(注6)交差検証
データセットを学習用とテスト用に繰り返し分けて予測精度を検証する手法。本研究ではデータセットを5等分する5分割交差検証を使用。
(注7)免疫組織化学染色
抗体を用いて特定のタンパク質の組織上の分布を明らかにする染色手法。本研究ではESR1抗体を使用。
(注8)半教師あり学習
通常の教師あり学習では、すべての学習データに正解ラベルがついている必要がある。本研究では組織標本のスポットごとに測定された遺伝子発現量が、各スポットの画像に対応する正解ラベルに該当する。半教師あり学習とは、ラベルのない学習データもうまく活用することで、学習データが十分に存在しない場合でも高精度な機械学習モデルを目指す手法である。本研究で用いたNoisy student法と呼ばれる半教師あり学習法では、まず通常の教師あり学習を実施した後、ラベルのない画像に予測ラベルを付ける。正解ラベル付き画像と予測ラベル付き画像を一緒にして学習を行うことで、疑似的にサンプル数が増えて予測精度が向上することがある。
お問い合わせ
新領域創成科学研究科 広報室