2022-04-25 東京大学
寺川 瑛(生物科学専攻 博士課程3年生)
Yanhui Hu(ハーバード大学医学大学院 博士)
Norbert Perrimon(ハーバード大学医学大学院 教授)
黒田 真也(生物科学専攻 教授)
発表のポイント
- インスリン刺激下のショウジョウバエ細胞のマルチオミクスデータ(注1)から構築した分子間相互作用ネットワークを網羅的な単一遺伝子ノックアウト時の細胞増殖のデータと統合解析することで、インスリン刺激下で増殖制御に関与するネットワークを同定しました。
- 本研究で開発されたデータ解析手法により、先行研究では未解明の部分が多かった分子間相互作用ネットワークと表現型との関係について、大規模に評価することが可能になりました。
- 本開発手法は今後さまざまな生命現象や疾患の作用機序(注2)解明に利用されることが期待されます。
発表概要
増殖・成長などの細胞の表現型は、転写物、タンパク質および代謝物を含む大規模な分子間相互作用ネットワークにより制御されています。これまでに、オミクス計測技術(注3)やデータ解析手法の進歩に伴い、包括的な細胞内分子間相互作用ネットワークの同定が可能になってきました。しかしながら、そうして得られたネットワークと表現型との関係性を結びつける方法はこれまでになく、同定したネットワークの生理的意義の解明や、疾患との結びつきを考察する上での障壁になっていました。
東京大学大学院理学系研究科の寺川瑛博士課程学生、黒田真也教授、ハーバード大学医学大学院のYanhui Hu博士、Norbert Perrimon教授らは、奈良先端科学技術大学院大学データ駆動型サイエンス創造センターの小鍛治俊也助教、理化学研究所生命医科学研究センターの柚木克之チームリーダー、ハーバード大学医学大学院のJohn M. Asara准教授、Martha L. Bulyk教授らとの共同研究により、マルチオミクスデータの統合解析から構築した大規模な分子間相互作用ネットワーク(トランスオミクスネットワーク(注4))と、遺伝子ノックアウト時の表現型の計測データであるCRISPRノックアウトスクリーニングデータ(注5)(以下、CRISPRスクリーニングデータ)の統合解析手法を開発しました。特に本研究では、開発手法をショウジョウバエ細胞より取得したインスリン刺激下のマルチオミクスデータおよび細胞増殖のCRISPRスクリーニングデータに適用し、インスリン刺激下で細胞増殖制御に関与するトランスオミクスネットワークを明らかにしました。
本研究で得られた知見は、将来的にインスリンが関与する疾患の病態理解や治療に繋がる可能性があります。さらに、開発手法がその他のさまざまな生命現象や疾患の作用機序解明にむけて利用されていくことも期待されます。
発表内容
研究の背景・先行研究における問題点
増殖・成長などの細胞の表現型は、栄養条件や増殖因子などの細胞外部の環境や刺激に応じて、転写物、タンパク質および代謝物を含む大規模なトランスオミクスネットワークを変化させることで適切に制御されています。現在までに、オミクス計測技術の発展により、さまざまな実験条件下で上記分子群の存在量や相互作用の網羅的計測が可能になりつつあります。それに伴い、計測データから実験条件下で活性化または不活性化するトランスオミクスネットワークを推定する手法も盛んに開発されてきました。しかしながら、そのようにして計測・推定されてきたネットワーク中のどの部分ネットワークがどの表現型に関与しているかという点についてはほとんど評価されておらず、得られたネットワークと表現型とを紐づけることが当該分野における課題の一つでした。
インスリンは、ショウジョウバエから哺乳類まで進化的に高度に保存され、血糖値低下作用や細胞増殖・成長の促進作用をもつ重要なホルモンです。これまでに、本研究グループの先行研究も含め、インスリン刺激下で制御される大規模なトランスオミクスネットワークが同定されてきましたが、ネットワーク中の大部分について細胞増殖を含む表現型に対しての寄与は未解明でした。
そこで本研究グループは、網羅的な単一遺伝子ノックアウト時の細胞表現型の計測データであるCRISPRスクリーニングデータを、マルチオミクスデータ解析から構築したトランスオミクスネットワークと統合解析する手法を開発し、上記課題の解決を試みました。特に本研究では、ショウジョウバエ細胞におけるインスリン刺激下のマルチオミクスデータおよび細胞増殖のCRISPRスクリーニングデータに開発手法を適用し、インスリン刺激下で細胞増殖の制御に関与するトランスオミクスネットワークを明らかにしました。
研究内容
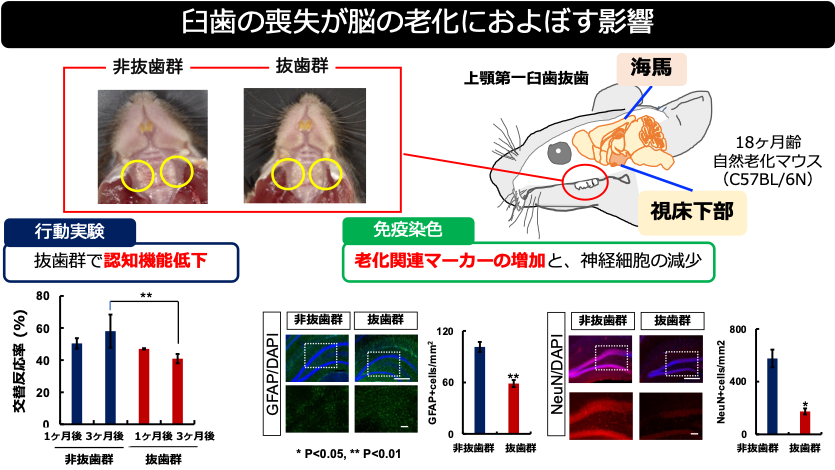
本研究グループは、1)インスリン刺激下のマルチオミクスデータを用いてインスリンに制御されるトランスオミクスネットワークを構築し、2)得られたトランスオミクスネットワークと細胞増殖のCRISPRスクリーニングデータの統合解析を行うことで、インスリン刺激下で細胞増殖制御に関与するトランスオミクスネットワークを明らかにしました(図1)。

図1:研究概要
ショウジョウバエS2R+細胞より取得したインスリン刺激下のマルチオミクスデータ(左上)と分子間相互作用データベース・ソフトウェア(左中)を使用し、インスリン作用のトランスオミクスネットワークを構築した(左下)。さらに、構築したトランスオミクスネットワークとショウジョウバエS2R+細胞より取得した細胞増殖のCRISPRノックアウトスクリーニングデータ(右上)を統合解析することで、インスリン刺激下の細胞増殖に必須な部分ネットワークを推定した(右下赤枠内)。
1)インスリン作用のトランスオミクスネットワークの構築
まず研究グループは、インスリン刺激下のショウジョウバエ胚由来S2R+細胞より取得した、タンパク質間相互作用、リン酸化タンパク質量、遺伝子発現量、代謝物量のオミクスデータを統合解析し、インスリン刺激下で活性化または不活性化するトランスオミクスネットワークを構築しました(図1左)。そのうち、代謝物量のデータは本研究で、その他のデータは本研究グループの先行研究で計測されました。ネットワーク構築では、各オミクスデータからインスリン刺激によって増加または減少する分子群(インスリン応答性分子群)および相互作用を同定し、さまざまな分子間相互作用に関するデータベースの情報を用いてインスリン応答性分子群の間の制御関係を推定しました。特に本研究では、既に開発済みであったトランスオミクスネットワーク構築手法を拡張し、タンパク質間相互作用のデータをネットワークに組み込むことで、先行研究よりも詳細かつ網羅性の高いネットワーク構築が可能になりました。
構築したネットワークを解析することで、インスリン刺激下のショウジョウバエ細胞で機能する分子間相互作用ネットワークが明らかになりました。具体的には、インスリンシグナル伝達経路(注6)の下流で転写因子Mycを含む14種類の転写因子群が制御され、これらの転写因子群を介してヌクレオチド代謝、転写および翻訳に関与する遺伝子群の発現が増加していました。そして、代謝物レベルでは転写および翻訳の基質であるヌクレオシド三リン酸やアミノ酸の量が減少することが明らかになりました(図1左下)。
2)インスリン作用のトランスオミクスネットワークと細胞増殖のCRISPRスクリーニングデータの統合解析
続いて本研究グループは、構築したトランスオミクスネットワークと、細胞増殖のCRISPRスクリーニングデータを統合解析し、インスリン刺激下で増殖制御に関与する部分ネットワークを同定しました(図1右)。CRISPRスクリーニングデータは、本研究グループの先行研究でショウジョウバエS2R+細胞を対象に計測され、このデータを用いることでトランスオミクスネットワーク中の各分子がどの程度細胞増殖に寄与しているか評価することができます。
本研究グループは、トランスオミクスネットワーク中に含まれる、各転写因子とその上流のシグナル伝達経路および下流のターゲット遺伝子群からなる部分ネットワーク(以下、部分ネットワーク)それぞれについて細胞増殖への寄与を評価しました。具体的には、細胞増殖の制御に関与する部分ネットワークには、ノックアウト時に大きな増殖低下を引き起こす分子群が多数含まれていると予想されるため、そのような条件を満たす部分ネットワークをGene set enrichment analysis(注7)と呼ばれる手法により同定しました。
解析結果から、転写因子Mycを介して、ヌクレオチド代謝や翻訳関連の遺伝子群の発現を促進する部分ネットワークが、インスリン刺激下の細胞増殖制御に関与することが明らかになりました(図1右下)。Mycはショウジョウバエおよび哺乳類のいずれでも、細胞増殖・成長を促進することが知られ、特にショウジョウバエではインスリンシグナル伝達経路による制御も知られています。このように、開発手法の信頼性の高さを示す結果が得られたことに加え、本研究で推定されたMycの上流および下流の分子群の応答とMycの活性化の因果関係を今後詳細に検証することで、インスリン作用の新規作用機序の解明が期待されます。
社会的意義・今後の予定
現在までに、計測技術の発展に伴い大量のオミクスデータが蓄積されてきましたが、オミクスデータは大規模でありデータを眺めるだけでは解釈が難しく、オミクスデータから表現型に関係するような重要な知見を抽出するデータ解析手法の発展が求められていました。本解析手法はそのような課題の解決に貢献できると考えられます。本解析手法の枠組みは、幅広い生物種や生命現象に適用可能であり、本研究の対象であるインスリン作用に加えて、さまざまな生命現象や疾患の作用機序解明に開発手法が使用されていくことが期待されます。
本研究は、科学技術振興機構における戦略的創造研究推進事業「多細胞間での時空間的相互作用の理解を目指した定量的解析基盤の創出」研究領域 研究課題名「時空間トランスオミクスを用いた多細胞・臓器連関代謝制御の解明」(課題番号:JPMJCR2123 研究代表者:黒田真也)、日本学術振興会における科学研究費助成事業の新学術領域研究(研究領域提案型)「2型糖尿病の代謝アダプテーション」(課題番号:JP17H06299, JP17H06300 研究代表者:黒田真也)の一環として得られました。
発表雑誌
- 雑誌名
iScience論文タイトル
Trans-omics analysis of insulin action reveals a cell growth sub-network which co-regulates anabolic processes著者
Akira Terakawa, Yanhui Hu, Toshiya Kokaji, Katsuyuki Yugi, Keigo Morita, Satoshi Ohno, Yifei Pan, Yunfan Bai, Andrey A. Parkhitko, Xiaochun Ni, John M. Asara, Martha L. Bulyk, Norbert Perrimon*, Shinya Kuroda*
用語解説
注1 マルチオミクスデータ
生体分子全体を網羅的に研究する学問のことをオミクスと呼ぶ。また、リン酸化タンパク質、転写物、代謝物などの複数種類のオミクスデータをマルチオミクスデータと呼ぶ。
注2 作用機序
タンパク質、転写物および代謝物などの生体分子の生化学的相互作用を介して当該作用を制御する仕組み。
注3 オミクス計測技術
タンパク質、転写物、代謝物などの各生体分子種を網羅的に計測する技術。転写物量の計測には主に次世代シーケンサー、タンパク質量、タンパク質間相互作用および代謝物量の計測には主に質量分析計が使用される。
注4 トランスオミクスネットワーク
シグナル経路・遺伝子発現・代謝物層にまたがる大規模ネットワーク。
注5 CRISPRノックアウトスクリーニングデータ
CRISPR-Casシステムを利用した遺伝子ノックアウト技術を用いて各遺伝子をノックアウトした際の、表現型の計測データ。本研究では、細胞増殖表現型のCRISPRスクリーニングデータを使用した。
注6 インスリンシグナル伝達経路
インスリンは細胞膜上のインスリン受容体に結合するとタンパク質のリン酸化やタンパク質間相互作用を介して、遺伝子発現や代謝を制御する。このような、分子群の応答の連鎖をインスリンシグナル伝達経路と呼ぶ。
注7 Gene set enrichment analysis
複数の遺伝子セットおよび各遺伝子に紐付けられた定量値を入力とし、特定の遺伝子セットが統計的に有意に高いまたは低い定量値に偏っているか判定する手法。本研究では、遺伝子セットは各転写因子の上流または下流の遺伝子群に、定量値はCRISPRスクリーニングデータに対応する。