2022-08-23 理化学研究所,東京大学
理化学研究所(理研)生命医科学研究センター自己免疫疾患研究チームの中野正博特別研究員、ヒト免疫遺伝研究チームの石垣和慶チームリーダー、自己免疫疾患研究チームの山本一彦チームリーダー、東京大学大学院医学系研究科アレルギー・リウマチ学の藤尾圭志教授らの共同研究グループは、全身性エリテマトーデス(SLE)の病態に関わる免疫細胞の遺伝子発現異常を多数同定しました。
本研究成果は、SLEのさらなる病態理解と新しい治療標的の発見に貢献すると期待できます。
SLEは、発症後に寛解[1]と増悪(重症化)を繰り返し、さまざまな臓器が侵される難治性自己免疫疾患[2]であり、詳細な病態は未解明です。
今回、共同研究グループは、SLE患者と健常人の血液から取り出した27種の免疫細胞(計6,386サンプル)を解析し、疾患に関わる遺伝子の発現パターンを詳しく調べました。さまざまな症状を持つSLE患者に対して、免疫細胞を細かく分けた高精度の遺伝子発現解析を行ったことで、SLEの発症時と増悪時に、異なる免疫細胞がそれぞれ異なるメカニズムで働いていることが初めて明らかになりました。発症時と増悪時を区別してSLEの病態を理解することにより、新たな治療標的の同定につながることが期待できるとともに、今後の遺伝子解析研究の新たな方向性を示すことに成功しました。
本研究は、科学雑誌『Cell』オンライン版(8月22日付:日本時間8月23日)に掲載されました。
本研究の概略
背景
全身性エリテマトーデス(SLE)は、免疫システムが自己の細胞や臓器を攻撃する自己免疫疾患の一つで、ひとたび発症すると寛解と増悪を繰り返し、治癒することが困難な慢性疾患です。(1)多くの遺伝子が疾患の病態に関与すること、(2)血液中の多様な免疫細胞が複雑に病態成立に関与すること、(3)疾患活動性[3]や臓器症状(皮膚・関節・腎障害など)が患者ごとに大きく異なることなどの理由から疾患の病態理解が進まず、効果的な治療薬があまり多く開発されていません。
SLEの病態を理解するには、患者に起きている免疫状態の異常を調べる必要があります。免疫細胞の遺伝子発現情報は、個人の免疫状態をリアルタイムに評価する有用な指標です。過去の遺伝子解析研究では、一度の研究で調べるSLE患者の数が少なかったため、異なる臨床症状の患者にどのような遺伝子発現の違いがあるかを十分に解明できませんでした。また、血液のあらゆる免疫細胞集団をひとまとめにして解析していたため、SLE患者のどの免疫細胞でどのような遺伝子発現異常が起きているか詳しく分かっていませんでした。
本研究では、さまざまな症状のSLE患者の血液から細胞集団を細かく分けて、それぞれの症状を特徴付ける遺伝子発現パターンを調べることで、SLEの複雑な病態メカニズムを詳細に解明することを目指しました。
研究手法と成果
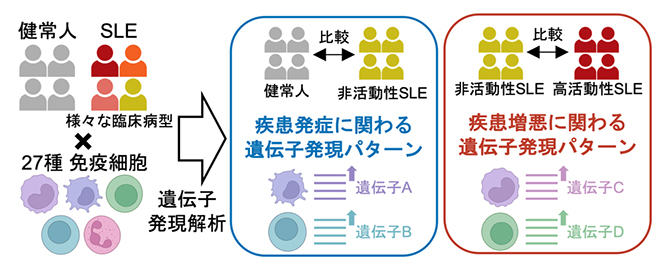
共同研究グループは、機能ゲノムコホートImmuNexUT[4]におけるSLE患者136例と健常人89例から27種に及ぶ免疫細胞6,386サンプルをセルソーター[5]で分取し、RNAシーケンス[6]で遺伝子発現量を網羅的に調べる過去最大規模の解析を行いました(図1)。今回あらゆる疾患活動性の患者が参加したことで、大きく二つのタイプの遺伝子発現パターンを定義することができました(図1)。
一つは寛解状態にある非活動性のSLE患者群と健常人を比較した発現変動遺伝子[7]からなる「疾患状態シグネチャー」と呼ばれるタイプであり、疾患の発症に関わる遺伝子群を反映します。もう一つは高疾患活動性と非活動性のSLE患者群を比較した発現変動遺伝子からなる「疾患活動性シグネチャー」と呼ばれるタイプであり、疾患の増悪に関わる遺伝子群を反映します。27の細胞種それぞれで解析を行い、疾患状態シグネチャー、疾患活動性シグネチャーともに1細胞種当たりおよそ2,000もの発現変動遺伝子を同定しました。
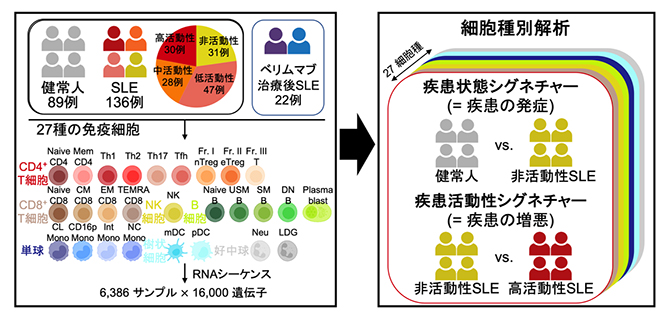
図1 研究の手法
SLE患者136例と健常人89例から27種の免疫細胞を分取し(計6,386サンプル)、RNAシーケンスで遺伝子発現量を網羅的に調べる大規模解析を行った。細胞種別に解析を行い、健常人と非活動性SLE患者群の発現変動遺伝子を「疾患状態シグネチャー」(=疾患の発症に関わる遺伝子群)、非活動性と高活動性SLE患者群の発現変動遺伝子を「疾患活動性シグネチャー」(=疾患の増悪に関わる遺伝子群)と定義した。
細胞種ごとに疾患状態シグネチャーと疾患活動性シグネチャーを比較してみると、多くの細胞で両者の遺伝子のメンバーが大きく異なることが分かりました(図2)。例えば、免疫シグナルの担い手であるサイトカイン[8]をコードする遺伝子に注目すると、疾患状態シグネチャーと疾患活動性シグネチャーではそれぞれ異なるサイトカイン群が同定されました(図3)。これらの結果から、SLEの発症と増悪では異なる病態メカニズムが働いている可能性が高いことが初めて明らかになりました。
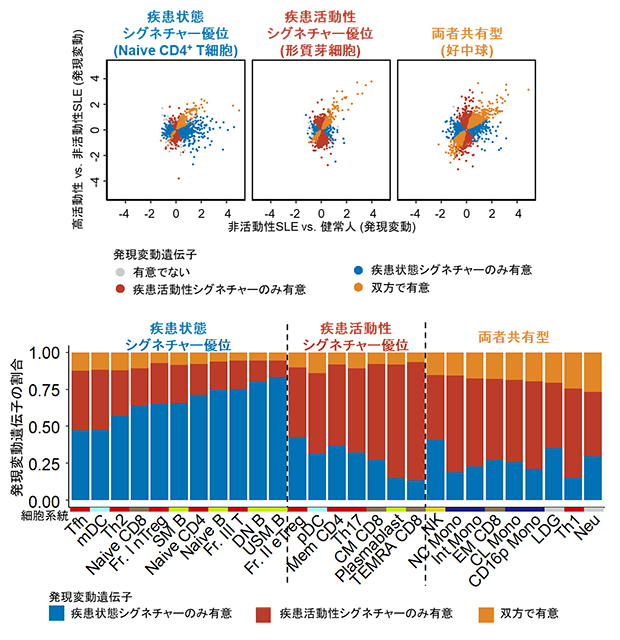
図2 SLEにおける疾患状態シグネチャーと疾患活動性シグネチャー
27の細胞種ごとに疾患状態シグネチャーの遺伝子群と疾患活動性シグネチャーの遺伝子群のメンバーを比較した。
(上)一つ一つの点が遺伝子を表し、点の色は二つの発現変動解析での有意性に基づく。横軸が疾患状態シグネチャーの発現変動、縦軸が疾患活動性シグネチャーの発現変動を表す。細胞種によって疾患状態シグネチャーが多いもの、疾患活動性シグネチャーが多いもの、両者が比較的共有されるものがあることが分かった。
(下)27の細胞種を疾患状態シグネチャーが多い群、疾患活動性シグネチャーが多い群、両者が比較的共有される群に分類できた。
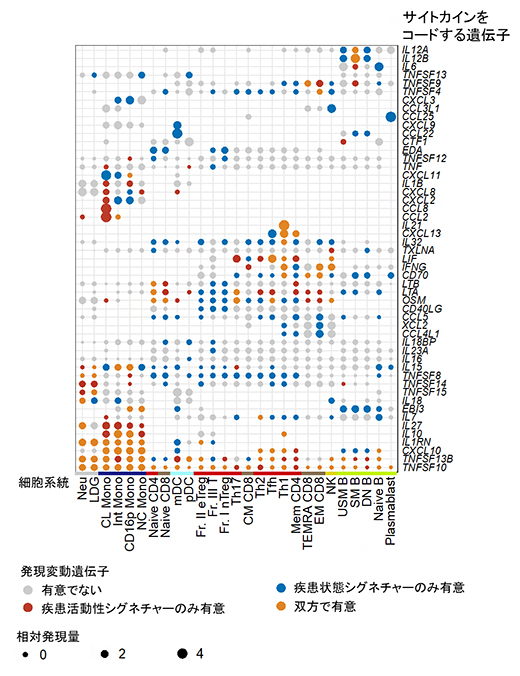
図3 サイトカイン関連遺伝子における疾患状態シグネチャーと疾患活動性シグネチャーの違い
サイトカインをコードする遺伝子に限定して、それぞれの遺伝子がどの細胞種で疾患状態シグネチャーと疾患活動性シグネチャーに該当するかを示した。疾患状態シグネチャーと疾患活動性シグネチャーでは、異なるサイトカインの変動パターンが生じていることが分かる。
次に、SLEの臓器症状の多様性に注目し、それぞれの臓器症状でどの免疫細胞が活性化しているかを調べました。主成分分析[9]と分散成分解析[10]を免疫細胞種ごとの遺伝子発現データに応用することで、皮膚症状を持つ患者ではヘルパーT細胞[11]の一種であるTh1細胞[11]、関節症状を持つ患者では単球系細胞[12]、腎症状を持つ患者では好中球系細胞[12]の遺伝子発現パターンが最も強い関連を示し、それぞれの症状で異なる免疫細胞が活性化していることが示唆されました(図4)。
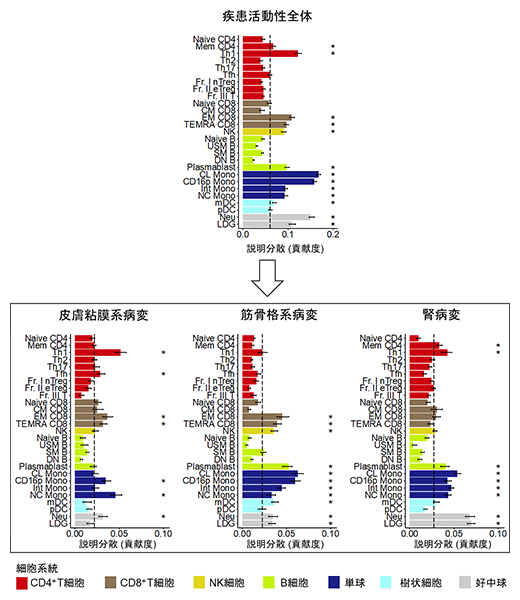
図4 臓器病変に対する細胞種ごとの貢献度の違い
主成分分析と分散成分解析を免疫細胞種ごとの遺伝子発現データに応用することで、細胞種ごとの臓器病変に対する貢献度を定量化した。皮膚粘膜病変(紅斑や皮疹など)ではTh1細胞が、筋骨格系病変(関節炎など)では単球系の細胞が、腎病変では好中球系の細胞が最も病態に寄与している可能性が示唆された。
さらに、既存のSLE治療薬が患者のどの免疫細胞に作用しているかを調べました。ベリムマブ[13]治療前後の発現変動遺伝子はB細胞系[14]細胞に集積し、ミコフェノール酸モフェチル[15]内服者と非内服者の発現変動遺伝子はTh1細胞、メモリーCD8陽性T細胞[11]、形質芽細胞[14]に集積することが明らかになりました(図5)。さらに、これらの治療薬で抑制された遺伝子群は疾患活動性シグネチャー遺伝子群と特に治療反応良好患者群でよく重複しており、現在のSLE治療薬が疾患活動性シグネチャーを抑制することで、臨床的な効能を発揮していることが確認できました。以上のことから、今回同定された27の細胞種ごとの疾患活動性シグネチャーは、新たなSLEの治療標的を探索する上でも有用である可能性が示されました。
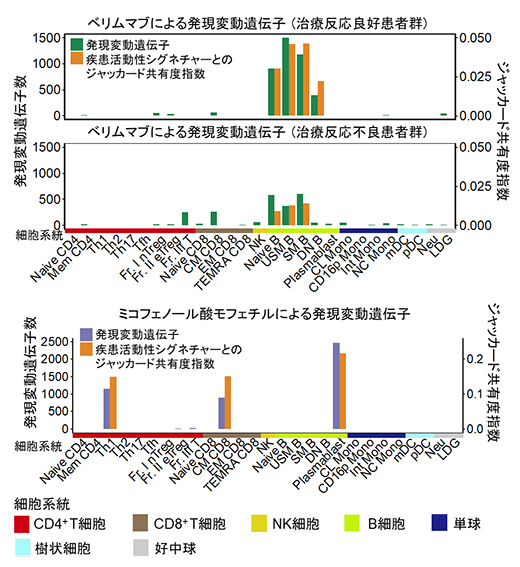
図5 SLEの治療薬による発現変動遺伝子と疾患活動性シグネチャー遺伝子との関連
(上)ベリムマブ治療前後の細胞種ごとの発現変動遺伝子数を、治療反応良好患者群と不良群に分けて示した。また、これらの発現変動遺伝子と疾患活動性シグネチャー遺伝子の共有度指数(ジャッカード指数)を細胞種ごとに示した。発現変動遺伝子はB細胞系細胞に集積し、また特に治療反応良好患者群で共有度指数が高いことが分かった。
(下)ミコフェノール酸モフェチル内服者と非内服者の細胞種ごとの発現変動遺伝子数と疾患活動性シグネチャー遺伝子との共有度指数を示した。発現変動遺伝子はTh1細胞、メモリーCD8細胞、形質芽細胞に集積することが分かった。
最後に、過去の大規模SLEゲノムワイド関連解析(GWAS)[16]結果との統合解析を実施しました。これまでのGWAS研究でSLEの発症リスクを高めるゲノム上の遺伝子多型[17](リスク多型[17])が100以上同定されましたが、これらの多くがどのよう機序を経てSLEの病態に関わっているか、十分に分かっていませんでした。統合解析において層別化LDスコア回帰法[18]を用いることで、ゲノム上のSLEのリスク多型が疾患状態シグネチャー遺伝子群をコードする領域の周囲に多く存在する一方、疾患活動性シグネチャー遺伝子群の周囲にはあまり多く存在していないことが分かりました(図6)。
この結果は、GWAS研究が主として「疾患対健常人」の症例対照研究に基づいて行われてきたことを考えると理に適った結果です。同時に、GWAS研究で得られた結果が疾患の増悪に関わる遺伝子群を十分に捕捉できていないことも示されました。つまり、今後のGWAS研究は従来の症例対照研究から症例内の多様性に注目した研究デザインにシフトする必要があると考えられます。
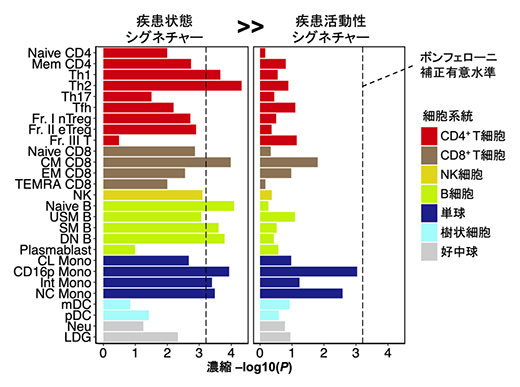
図6 本研究と過去のSLE GWASの統合解析
層別化LDスコア回帰法を用いて、ゲノム上で過去に同定されたSLEのリスク多型の分布と偏り(濃縮)を評価した。SLEのリスク多型は、疾患状態シグネチャー遺伝子群をコードするゲノム領域の周辺に多く存在する一方(9細胞種で有意)、疾患活動性シグネチャー遺伝子群をコードするゲノム領域の周辺にはあまり多く存在していない(有意な細胞種なし)ことが分かった。
今後の期待
SLEの治療目標は、疾患活動性をなるべく抑制することで再燃やダメージの蓄積を予防することと定められています。つまり、今回同定した疾患活動性シグネチャーに含まれる遺伝子情報を基点に、病態メカニズムに即した形で新たな治療標的が開発されることが期待できます。さらに、今後同一患者の遺伝子発現状態の推移を追う縦断的研究を行うことで、SLEの予後や再燃を予測する目安となるバイオマーカーの同定につながる可能性が期待できます。
補足説明
1.寛解
治療によって疾患の症状や検査異常が消失した状態を指す。治癒とは異なり、再燃のリスクがある。
2.自己免疫疾患
本来病原体やがんなどから自己を守るための免疫システムが暴走し、自己の細胞や組織を攻撃してしまう疾患群を指す。代表的疾患に関節リウマチやSLEがある。
3.疾患活動性
SLEによる疾患の勢いを表したもの。SLEは、長い経過の中で、疾患活動性が高い時期(高疾患活動性)と、治療により疾患が寛解状態となる時期(非活動性)を繰り返すことが多い。
4.ImmuNexUT
Immune cell gene expression atlas from the University of Tokyoの略称。代表的な10免疫疾患の患者および健常人(現時点で500例以上)から、全ゲノムシーケンスデータと最大28種類の免疫細胞RNAシーケンスデータを取得し、遺伝子の発現量やその調節機構を解析することを目的とした機能ゲノムコホート。
5.セルソーター
表面タンパクを蛍光抗体で標識した細胞を、蛍光を認識して分取する装置のこと。特定の表面タンパクの組み合わせによって、さまざまな細胞種を別々に集められる。
6.RNAシーケンス
次世代シーケンサーと呼ばれる一度に大量の塩基配列を読み取ることができる装置を用いて、網羅的に遺伝子の発現量を解析する手法。
7.発現変動遺伝子
異なる二つ以上の群において統計学的に有意に発現量の上昇(低下)を認めた遺伝子。
8.サイトカイン
主に免疫系細胞から分泌され、免疫シグナルを伝達する低分子タンパクの総称。炎症の重要な調節因子であり、SLEを含む自己免疫疾患の多くでサイトカインの調節異常が認められる。
9.主成分分析
多次元データの持つ情報をできるだけ損なわずに低次元空間に情報を縮約することで、データを理解しやすくする分析手法。
10.分散成分解析
データセットにおいて、ある変数Aのデータのばらつき(分散)が、異なる変数Bのデータのばらつきによってどの程度説明できるか(説明分散、貢献度)を定量化する解析。
11.ヘルパーT細胞、Th1細胞、メモリーCD8陽性T細胞
T細胞は胸腺という組織で分化、成熟したリンパ球の一種で、抗原提示細胞から抗原情報を受け取ることで、さまざまな機能を発揮する。ヘルパーT細胞は抗原情報に応答して、他の免疫細胞の機能を調整する役割を果たす。Th1細胞はヘルパーT細胞の一種であり、主にウイルスや細菌を攻撃する際に働くと考えられている。キラーT細胞はウイルス感染細胞やがん細胞を直接攻撃、排除する役割を果たし、メモリーCD8陽性T細胞はその代表的細胞と考えられている。
12.単球系細胞、好中球系細胞
単球、好中球ともに白血球の一種であり、感染に対する防衛の開始に重要な役割を果たす。単球はヘルパーT細胞などからの指示を受け、マクロファージに分化することで細菌などの異物を細胞内に取り込み、消化する(貪食作用)。好中球は細菌感染の最初に集まる細胞で、特に貪食作用と殺菌作用が強い。
13.ベリムマブ
B細胞活性化因子を阻害するヒトモノクローナル抗体製剤。SLEの治療薬として本邦でも、2017年に点滴静注と皮下注射製剤が承認された。
14.B細胞系細胞、形質芽細胞
B細胞は骨髄という組織で分化、成熟したリンパ球の一種で、ある抗原が体内に侵入すると、ヘルパーT細胞などにより活性化し、形質芽細胞、形質細胞へ分化する。この分化過程でその抗原に対する抗体(免疫グロブリン)を産生・分泌することで、抗原を攻撃する。
15.ミコフェノール酸モフェチル
細胞のプリン生合成経路を阻害することでDNA合成を抑制する免疫抑制剤。主にT細胞、B細胞に作用すると考えられており、SLEでは腎炎症状のある患者を中心に用いられている。
16.ゲノムワイド関連解析(GWAS)
疾患の発症や身長・体重などの量的な形質に影響を持つゲノム上の遺伝子多型を、網羅的に検索、同定する解析手法。GWASはGenome wide association studyの略。
17.遺伝子多型、リスク多型
ヒトゲノムを構成している塩基配列の個体差のことを遺伝子多型と呼ぶ。GWASで同定された疾患の発症に影響する遺伝子多型のことをリスク多型と呼ぶ。
18.層別化LDスコア回帰法
ゲノム全体に存在する多くの遺伝子多型が疾患発症に関わるというポリジェニック・モデルを前提とした解析手法。各遺伝子多型間の相関情報(LDスコア)とGWAS統計量の関連を利用することで、遺伝子多型の形質に対する説明分散(貢献度)を定量化、検定する手法をLDスコア回帰法と呼ぶ。層別化LDスコア回帰法は、LDスコア回帰法を拡張した手法であり、ゲノム上の領域を複数に分割(層別化)することで、それぞれの領域の形質に対する説明分散(貢献度)を定量化、検定することができる。
共同研究グループ
理化学研究所 生命医科学研究センター
自己免疫疾患研究チーム
特別研究員 中野 正博(ナカノ・マサヒロ)
チームリーダー 山本 一彦(ヤマモト・カズヒコ)
ヒト免疫遺伝研究チーム
チームリーダー 石垣 和慶(イシガキ・カズヨシ)
ゲノム解析応用研究チーム
チームリーダー 寺尾 知可史(テラオ・チカシ)
東京大学
大学院医学系研究科 アレルギー・リウマチ学
(医学部附属病院 アレルギー・リウマチ内科)
教授 藤尾 圭志(フジオ・ケイシ)
大学院医学系研究科 免疫疾患機能ゲノム学講座
特任助教 太田 峰人(オオタ・ミネト)
特任准教授 岡村 僚久(オカムラ・トモヒサ)
研究支援
本研究は、中外製薬株式会社との共同研究費、日本医療研究開発機構(AMED)ゲノム医療実現推進プラットフォーム事業「免疫担当細胞eQTLデータを用いた免疫介在性疾患ゲノム情報からの層別化および予後予測モデルの構築」(研究開発代表者:藤尾圭志)、同ムーンショット型研究開発制度「病気につながる血管周囲の微小炎症を標的とする量子技術、ニューロモデュレーション医療による未病時治療法の開発」(プロジェクトマネージャー:村上正晃)による支援を受けて行われました。
原論文情報
Masahiro Nakano, Mineto Ota, Yusuke Takeshima, Yukiko Iwasaki, Hiroaki Hatano, Yasuo Nagafuchi, Takahiro Itamiya, Junko Maeda, Ryochi Yoshida, Saeko Yamada, Aya Nishiwaki, Haruka Takahashi, Hideyuki Takahashi, Yuko Akutsu, Takeshi Kusuda, Hiroyuki Suetsugu, Lu Liu, Kwangwoo Kim, Xianyong Yin, So-Young Bang, Yong Cui, Hye-Soon Lee, Hirofumi Shoda, Xuejun Zhang, Sang-Cheol Bae, Chikashi Terao, Kazuhiko Yamamoto, Tomohisa Okamura, Kazuyoshi Ishigaki, and Keishi Fujio, “Distinct transcriptome architectures underlying lupus establishment and exacerbation”, Cell, 10.1016/j.cell.2022.07.021
発表者
理化学研究所
生命医科学研究センター 自己免疫疾患研究チーム
特別研究員 中野 正博(ナカノ・マサヒロ)
チームリーダー 山本 一彦(ヤマモト・カズヒコ)
ヒト免疫遺伝研究チーム
チームリーダー 石垣 和慶(イシガキ・カズヨシ)
東京大学
大学院医学系研究科 アレルギー・リウマチ学
(医学部附属病院 アレルギー・リウマチ内科)
教授 藤尾 圭志(フジオ・ケイシ)
報道担当
理化学研究所 広報室 報道担当
東京大学医学部附属病院
パブリック・リレーションセンター(担当:渡部、小岩井)