2024-01-19 京都大学iPS細胞研究所
ポイント
- 特定の機能をもつRNA配列を学習し、同等の機能を発揮する新規の配列を生成する深層生成モデル注1)“RfamGenアールファムジェン注2)“を構築した。
- 変分オートエンコーダ(VAE)注3)にRNAの数理モデルである共分散モデル注4)を統合することで、新規RNA生成の性能を高め、少数データでも安定的な性能を実現した。
- RfamGenは入力データの特徴を捉えながら情報を集約しており、RNA設計のカスタマイズに有用である。
- RfamGenによる人工RNAは、学習RNA群と同等の構造と機能を保持し、天然RNAよりも高い機能活性をもつ可能性がある。
- 創薬や基礎研究におけるRNA設計のコスト削減と高速化につながることが期待される。
1. 要旨
角俊輔 氏(京都大学iPS細胞研究所(CiRA)未来生命科学開拓部門 大学院生、早稲田大学理工学術院 研究室受け入れ)、浜田道昭 教授(早稲田大学理工学術院)、齊藤博英 教授(CiRA同部門)は、目的の機能と構造をもつ人工RNA設計を支援する世界初の深層生成モデル”RfamGen”を開発しました。
RfamGenは、深層生成モデルで広く用いられている手法の一つである変分オートエンコーダ(VAE)と、RNA配列と二次構造注5)の情報から機能性RNAを分類することのできる共分散モデルを組み合わせたもので、特定の機能と構造の特徴をもつRNA群の特徴を学習し、人工配列を生成することができます。
研究グループは、RfamGenが学習したRNA群と相同な構造と機能をもつRNA配列が安定的に生成できることをコンピュータ上の解析と生化学実験の両方で確認しました。また、このRfamGenの性能は、深層生成モデルに共分散モデルを適用した結果であることがわかりました。さらに、RfamGenによる生成配列のRNAを大規模に合成し、網羅的にその活性を検証したところ、生成配列のRNAは天然のRNAよりも高い活性を示す傾向もみられました。
RfamGenによる学習結果を調べたところ、入力データのRNA群の二次構造や機能性のモチーフなどのバリエーションを、入力データの特徴の分布として効果的に集約していました。これにより、研究者が利用したいRNAの特徴をカスタマイズして、配列を生成することが容易になります。
RfamGenにより人工知能支援型のRNA設計が可能となることで、従来のRNA設計と比較し、開発コスト削減と高速化が実現し、核酸医薬や遺伝子治療などのRNA創薬の研究開発に貢献することが期待されます。
この研究成果は、2024年1月18日に英科学誌「Nature Methods」で公開されました。
2. 研究の背景
RNA分子は、遺伝子の転写調節や酵素活性など、その配列に応じてさまざまな機能を発揮し、基礎研究から医療まで幅広い場面で利用されています。しかし、利用目的に適した機能をもつRNAの塩基配列を設計することは高度な専門性と労力を要するため、RNA配列の特徴を適切に捉えて、機能性RNAを効率的よく設計できる、コンピュータを活用した手法の開発が期待されています。
これまでに機能性RNAの設計法として、RNA逆フォールディング注6)が主に研究されています。しかし、この手法には、手法の性質上、その正確性や汎用性にいくつか課題がありました。今回、研究グループは、RNAの配列と二次構造を数理的に記述できる共分散モデルとVAEを統合した機能性RNA生成のための世界初の深層生成モデルRfamGenを開発しました。
共分散モデルは、配列から特定の二次構造を検出し、幅広い種類のRNAをRNAファミリーとして分類でき、ゲノム配列から多くの機能性RNAを発見することに長年使われてきました。これまでに人工RNAの設計に共分散モデルが活用された例はありませんでしたが、研究グループはRNA分類に有用な共分散モデルを利用することで、従来の技術的課題を解決することができるのではないかと考えました。
3. 研究結果
1)RNAファミリー配列を設計する深層生成モデルRfamGen
RfamGenは、機能性RNAの生成性能を高めるため、VAEにRNAの分類に用いられる共分散モデルを統合しています(図1)。
共分散モデルは、RNAの配列と二次構造に基づき、複数のRNA配列どうしを互いに揃うように並べること(マルチプルアラインメント)ができます。RfamGenでは、はじめに目的の機能をもつ既存のRNA配列群を用意し、これを一つのRNAファミリーと見立てて、それぞれの配列に対して共分散モデルによるアラインメントを行います(図1左)。
RNA群のアライメント結果を、VAEの入力データとして使用します。VAEでは、入力したデータ群の特徴を学習し、入力データの特徴を確率分布として表現する「潜在空間」を構築します。RfamGenでは、RNAファミリーとみなしたRNA群の特徴を確率分布として表現する潜在空間が構築されます(図1中央)。
この潜在空間から出力されるデータは、入力に用いたRNA群の共分散モデルによる特徴を示すように生成されます。出力データを共分散モデルを介して配列に再構築することで、最終的に目的の機能を獲得した人工RNA配列を得ることができます(図1右)。
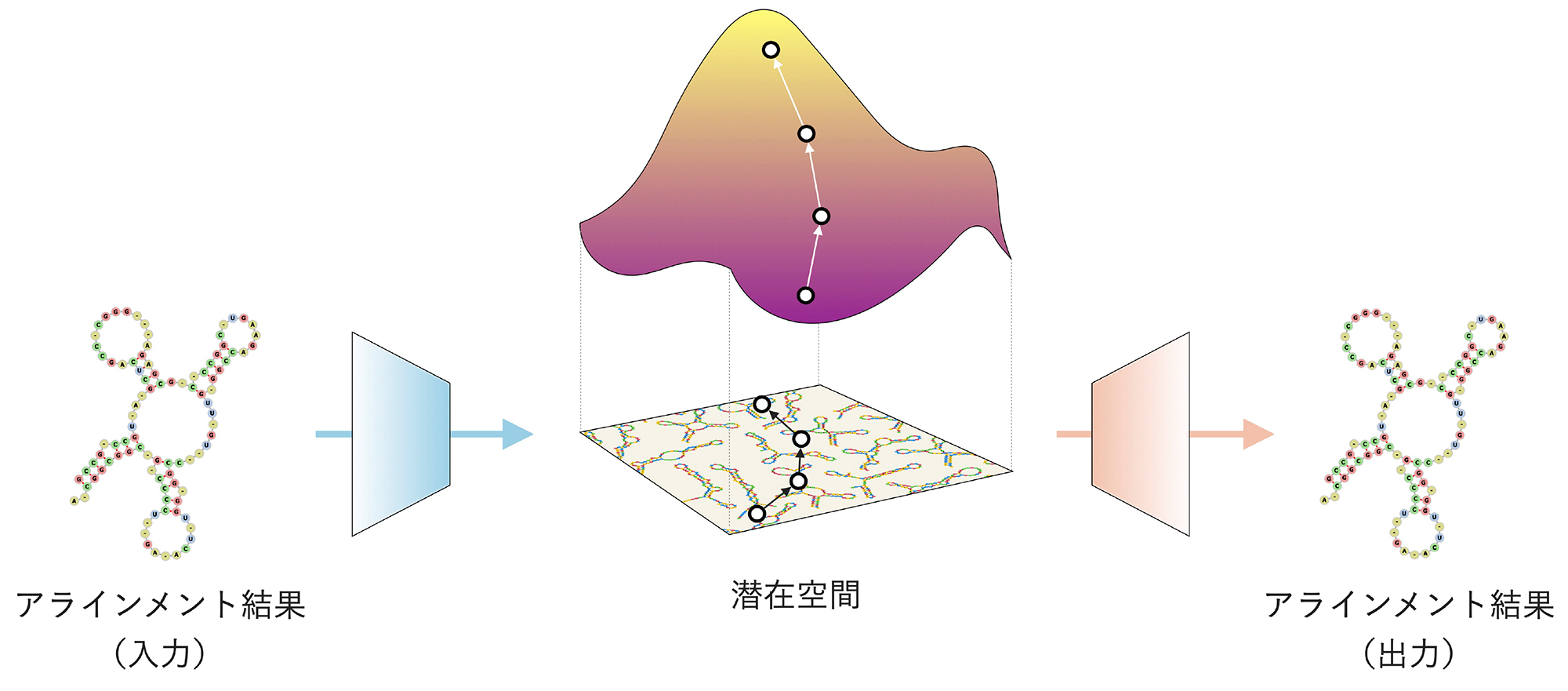
図1 RfamGenの概要
2)共分散モデルは深層生成モデルの性能と安定性を高める
RfamGenは共分散モデルを利用することで、アライメントと二次構造を学習します。それぞれの要素が生成能力にどのように関わるかを検証するため、比較対象として次の3種のモデルを用意しました。
① 共分散モデルによる入力データに含まれるアラインメントの情報を利用する深層生成モデル(GCVAE)
② 共分散モデルによる入力データに含まれる二次構造の情報を利用する深層生成モデル
③ 二次構造とアラインメントの情報をいずれも利用しない深層生成モデル
これら3種の深層生成モデルとRfamGenに、既知のRNAファミリー配列を学習させ、ランダムに1,000の配列を生成させました。そのうえで、学習に用いたRNAファミリーに共通の構造とどの程度、相同性をもつかをコンピュータ上で計算し比較しました。その結果、RfamGenが最も良いスコアを示すことがわかりました。
次に、学習に用いるRNA配列群のサンプル数の増減によりスコアがどのように変動するかについて研究グループは検証しました。まず、RfamGenに次いで良いスコアを出したアラインメント情報のみを利用する深層生成モデル(GCVAE)を比較対象としてRfamGenを検討した結果、多くの場合でRfamGenがGCVAEよりも高スコアを取ること、そして、サンプル数が少ない場合でもRfamGenの生成能力が安定して発揮されることが示唆されました(図2)。
これらの結果から、二次構造とアラインメント両方の学習が重要であり、共分散モデルを深層生成モデルと組み合わせることで、品質の高い人工RNA配列を安定的に生成できることが示唆されました。
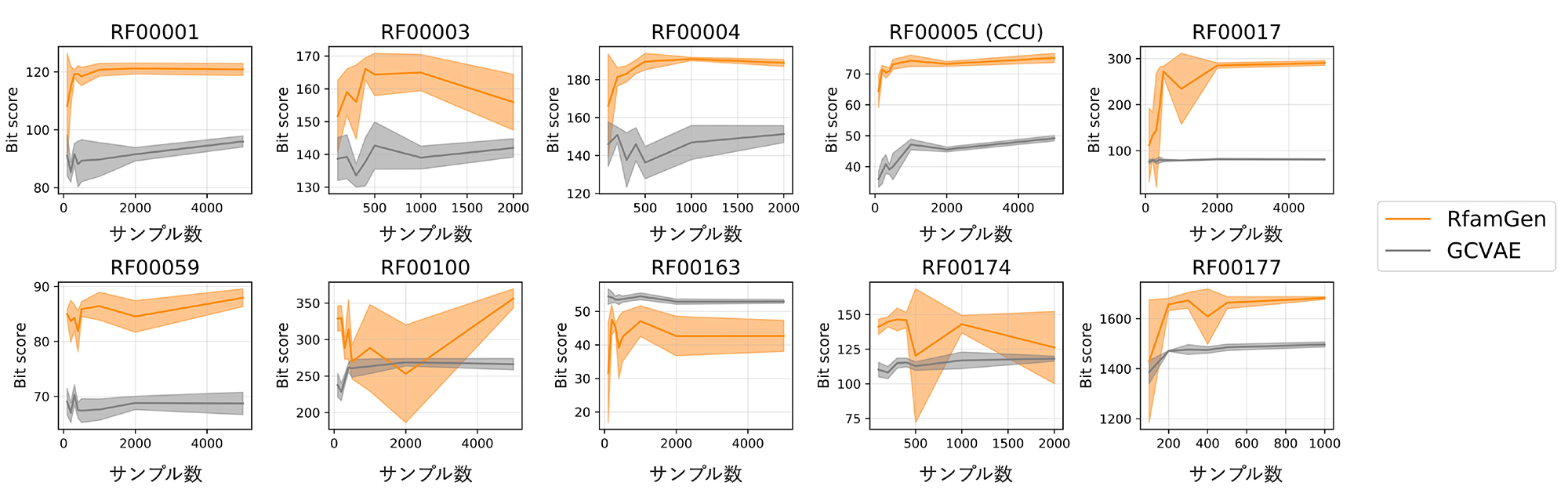
図2 サンプル数による生成性能の変動
表の縦軸 Bit Score:RNAファミリー配列らしさ
3)RfamGenによる潜在空間の学習
次に、VAEの潜在空間が、学習したRNA配列の特徴を的確に反映したものとなっているかを調べました。その結果、潜在空間を二次元に可視化したところ、RNAの二次構造にみられる多型領域や、標的タンパク質に結合する配列(モチーフ)など、入力データであるRNA配列の特徴の分布が、潜在空間に効果的に集約されていることがわかりました(図3)。このことから、RfamGenは、人工RNA設計支援ツールとして有用な、研究者が目的の機能と構造をもつ配列をより詳細にカスタマイズできる性能ももつことが示されました。
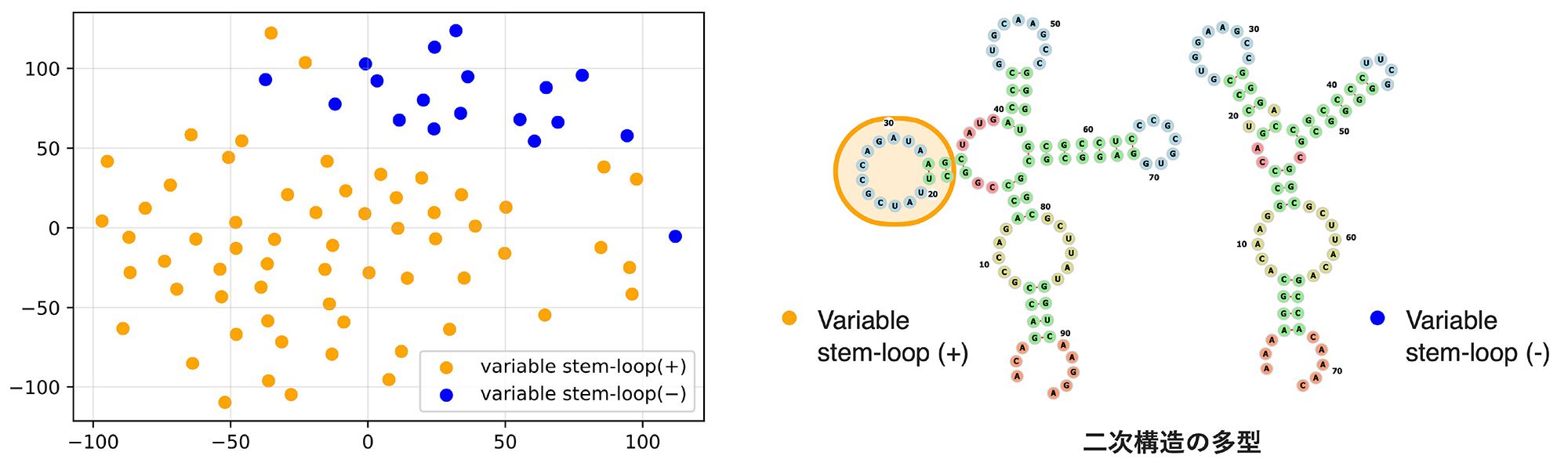
図3 潜在空間における配列情報の効果的な集約
4)RfamGenは高確率に活性配列を生成し、高活性を示す傾向がある
さらに、RfamGenを用いた人工RNAの性能を大規模な生化学実験によって評価しました。研究グループは、RNA分子のうち自己切断という酵素活性をもつ数百のRNA酵素(リボザイム)の配列情報を既存のデータベースから取得し、RfamGenの学習に使用しました。その結果、RfamGenから生成された配列が、実際に天然のRNAと相同な構造(図4左)と酵素活性をもつことがわかりました。この結果から、少数のデータで学習した場合も、RfamGenが期待した配列を生成できることを実験によって確認しました。
また、低分子に結合することで自己のRNA配列を切断する活性をもつRNA酵素であるglmSリボザイムを例に、RfamGenにより新規に1,000の配列を生成し、大規模に生成RNA配列の網羅的解析を行いました。その結果興味深いことに、RfamGenは酵素活性の高い配列を高確率に生成できることがわかりました(図4右)。
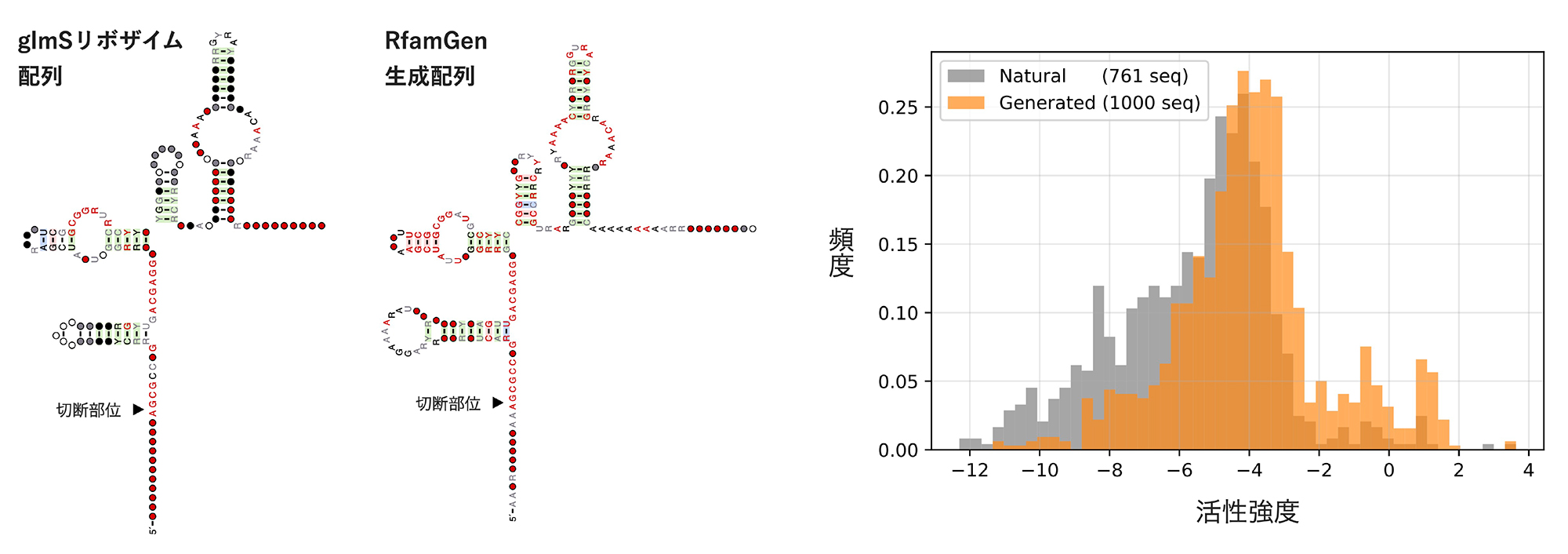
図4 RfamGen生成配列の構造と機能の評価
左:二次構造が相同な生成配列と天然配列
右:酵素活性の性能分布の比較(オレンジ:生成配列群、灰色:天然配列群)
4. まとめ
本研究では、RNA分類に利用される共分散モデルと深層生成モデルを統合し、人工RNA配列設計支援に用いることのできるRfamGenを構築しました。さらに、コンピュータ上と実験による性能評価によって、少数の入力データで学習した場合でも十分な性能が期待できることや、研究者が生成配列を詳細にカスタマイズ可能であること、入力データよりも高性能な人工RNA配列も生成しうることなど、RfamGenの有用性を示しました。今後、RfamGenを活用して、人工RNA設計を低コスト化、高速化し、生物学や医学など幅広い領域でRNAが活用されることに貢献することが期待されます。
5. 論文名と著者
〇論文名
Deep generative design of RNA family sequences
doi: 10.1038/s41592-023-02148-8
〇ジャーナル名
Nature Methods
〇著者
Shunsuke Sumi1,2,3, Michiaki Hamada3,4,5,*, Hirohide Saito1,*
*:責任著者
〇著者の所属機関
-
- 京都大学iPS細胞研究所(CiRA)
- 京都大学大学院 医学研究科
- 早稲田大学理工学術院
- 産業技術総合研究所 生体システムビッグデータ解析オープンイノベーションラボラトリ
(AIST CBBD-OIL) - 日本医科大学大学院医学研究科
6. 本研究への支援
本研究は、下記機関より支援を受けて実施されました。
- 科学技術振興機構(JST) 戦略的創造研究推進事業 CREST「イノベーション創発に資する人工知能基盤技術の創出と統合化」研究領域(研究総括:栄藤稔)「AIアプタマー創薬プロジェクト」(研究代表者:浜田道昭、主たる共同研究者:齊藤博英、グラント番号:JPMJCR21F1)
- 科学技術振興機構(JST) 戦略的創造研究推進事業 CREST「細胞操作」研究領域(研究総括:宮脇 敦史)「機能性RNA・RNP進化プラットフォームの構築と細胞制御技術の開発」(研究代表者:齊藤博英、主たる共同研究者:足立俊吾、浜田道昭、グラント番号:JPMJCR23B3)
- 日本学術振興会(JSPS)科学研究費補助金 特別推進研究
7. 用語説明
注1)深層生成モデル
コンピュータ上の多層化したニューラルネットワークにより情報の処理を行い、学習したデータの特徴をもったデータを新たに生成するモデルのこと。
注2)RfamGen
開発した深層生成モデルの名称。RNAファミリー(RNA family)配列の生成モデル(generator)であることから”RfamGen”と名付けた。
注3)変分オートエンコーダ(VAE)
深層生成モデルの手法の一つ。入力データを元にその特徴を確率分布として潜在空間にとらえ、入力データと似たデータを新たに生成(出力)することができる。VAEはVariational Autoencoderの略。
注4)共分散モデル
RNA配列の相同性を評価するアライメントに用いるモデル。ゲノム中の機能性RNAの探索に長年用いられている。共分散モデルにより、RNA配列は数千のRNAファミリーに分類されている。
注5)二次構造
1本鎖RNAの配列に応じて局所的に形成される塩基対構造。
注6)RNA逆フォールディング
RNAの構造から配列を計算する方法。配列から構造を計算するフォールディングの逆の流れのため、逆フォールディングと呼ばれる。