2024-07-05 理化学研究所,京都大学
理化学研究所(理研)生命医科学研究センター 理研-IFOMがんゲノミクス連携研究チームの小口 綾貴子 リサーチアソシエイト(京都大学 高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)特任研究員)、小松 秀一郎 客員研究員、村川 泰裕 チームリーダー(京都大学 高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)教授)、自己免疫疾患研究チームの鈴木 亜香里 上級研究員、山本 一彦 チームリーダー(生命医科学研究センター センター長)、ゲノム解析応用研究チームの寺尾 知可史 チームリーダーらの国際共同研究グループは、ヒトの多様なヘルパーT細胞[1]の遺伝子プロモーター[2]やエンハンサー[2]を1細胞レベルで調べることに成功しました。また、多様なヘルパーT細胞がさまざまな免疫疾患の発症にどのように関与するのかを系統的に解明しました。
本研究成果は、自己免疫疾患やアレルギー疾患の新しい治療法の開発に貢献すると期待されます。
今回、国際共同研究グループは、1細胞レベルでRNAの転写開始点を網羅的に同定し、遺伝子プロモーターやエンハンサーの活性を計測する新しい手法を開発し、約100万個のヒトの多様なヘルパーT細胞を解析しました。その結果、未報告の希少な亜集団を発見し、ヘルパーT細胞の思いがけない多様性を見いだしました。そして、これらのいろいろなヘルパーT細胞の亜集団で活性化しているエンハンサーと大規模な疾患ゲノムデータを統合することで、免疫疾患に関わり得る分子・細胞メカニズムを疾患横断的に解明しました。
本研究は、科学雑誌『Science』オンライン版(7月4日付:日本時間7月5日)に掲載されました。
エンハンサーを起点とした免疫疾患発症メカニズムの解明
背景
近年、大規模な疾患ゲノム解析が国際的に行われ、疾患発症に関わる遺伝的変異[3]が多く見つけられています。これらの多くは、タンパク質をコードしないノンコーディング領域[4]に位置しており、その領域の中でも特に、遺伝子発現のスイッチの構成要素であるエンハンサーにあることが分かってきました。しかし、ヒトゲノムにどれだけのエンハンサーが存在するかも、エンハンサーの機能的意義も、まだ十分に解明されていない段階です。
エンハンサーは、遺伝子遠方にあるDNA配列で、細胞種ごとに特異的に標的の遺伝子を活性化します。エンハンサーが活性化すると、その両端からエンハンサーRNA[2]が合成されます。このエンハンサーに遺伝的変異が生じると、その標的とする遺伝子の”質”ではなく”量”が変化します。それが疾患の発症に関わることが最近の研究で分かってきています。理研が中心となり結成されたFANTOM[5]コンソーシアムでは、エンハンサーRNAをヒトのさまざまな細胞・組織で解析し、エンハンサーの研究において世界をリードしてきました。しかし、これまでのRNA解析技術の限界から、1細胞レベルでの解析は十分に行えていませんでした。
ヘルパーT細胞は、免疫の司令塔としていろいろな機能を持っており、自己免疫疾患やアレルギー疾患に深く関与しています。ヘルパーT細胞には、Th1やTh2などさまざまな亜集団が存在しており、それぞれが異なる役割を担っています。ヘルパーT細胞の多様性を1細胞レベルで詳細に解き明かし、多様なヘルパーT細胞で活性化しているエンハンサーを網羅的に同定できれば、免疫疾患発症の遺伝的メカニズムの理解が深まることが期待されます。
研究手法と成果
国際共同研究グループは、RNAの先頭に付加されたキャップ構造[6]に由来するシグナルを高精度に捉える情報処理の手法に工夫をして、1細胞レベルでRNAの転写開始点を同定し、プロモーターとエンハンサーの活性を同時に解析できる独自の手法論、1細胞エンハンサー解析法(ReapTEC法)を開発しました。
ReapTEC法を、ヒトから採取した約100万個のヘルパーT細胞に適用することで、それぞれのヘルパーT細胞において、ダイナミックに変化しているRNAの転写開始点を同定してRNAの発現量を計測しました。その結果、報告されていない希少な亜集団を発見し、ヘルパーT細胞の多様性を見つけた上、それぞれのヘルパーT細胞の亜集団で活性化している遺伝子プロモーターおよびエンハンサーを網羅的に同定しました。その上で合計6万カ所を超えるヘルパーT細胞で活性化しているエンハンサーを同定し、ヘルパーT細胞の亜集団ごとのエンハンサー活性地図を作成しました(図1)。
図1 ヘルパーT細胞エンハンサー活性地図の作成
1細胞レベルでRNAの転写開始点を同定し、プロモーターとエンハンサーの活性を同時に解析できる独自の手法論(ReapTEC法)を開発し、ヒトの約100万個ものヘルパーT細胞にReapTEC法を適用することで、ヘルパーT細胞のエンハンサー活性地図を作成した。
次に、ある特定の免疫疾患に関連した遺伝的変異が、ヘルパーT細胞のどの亜集団のエンハンサーに存在しているのかを解析しました(図2)。
図2 エンハンサーを介した免疫疾患の分子・細胞メカニズムの解析
免疫疾患に関連した遺伝的変異が、ヘルパーT細胞のどの亜集団で特異的に活性化しているエンハンサーに重なっているのか、そしてそのエンハンサーの標的遺伝子はどれかを解析することで、免疫疾患の分子・細胞メカニズムを解析した。
大規模なゲノムワイド関連解析[7]によりいろいろな自己免疫疾患やアレルギー疾患に関連する遺伝的変異が数百カ所以上同定されています。こうした免疫疾患に関連した遺伝的変異の部位と、今回得られた多様なヘルパーT細胞で活性化しているエンハンサー領域を重ね合わせる解析を行いました(図3左)。まず、ヘルパーT細胞のエンハンサー領域には、全体としても、自己免疫疾患やアレルギー疾患に関連する遺伝的変異が強く濃縮していることが示されました(図3右)。また、免疫疾患に関連した遺伝的変異を持つヘルパーT細胞エンハンサー(疾患エンハンサー)を合計で約600カ所同定しました。
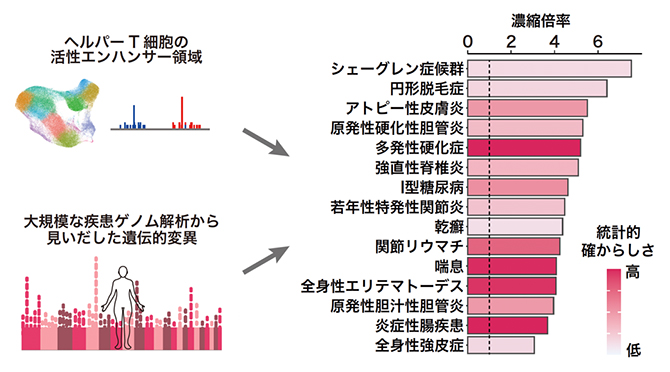
図3 ヘルパーT細胞のエンハンサー領域と大規模な疾患ゲノム解析との統合解析
ヘルパーT細胞のエンハンサー領域には、多発性硬化症や全身性エリテマトーデスなどの自己免疫疾患やアレルギー疾患に関連する遺伝的変異が強く濃縮していることが示された。
さらに、疾患エンハンサーの標的遺伝子を同定することで、疾患発症の関連分子を解析しました(図2)。エンハンサーは標的とする遺伝子に空間的に近接して働きかけます。そこで、国際共同研究グループは、ゲノムの3次元構造をヌクレオソーム[8]レベルの高い塩基解像度で決定することができるMicro-C法[9]を行いました。過去に前例のない深達度のMicro-CデータをヘルパーT細胞で作成して、どのゲノム領域が物理的に近接しているのかを解析しました。これにより、それぞれの疾患エンハンサーの標的遺伝子を推定しました(図4)。そして、自己免疫疾患やアレルギー疾患がどのような分子経路で発症に至るかを疾患系統的に解析し、新しい疾患関連分子を数多く同定しました。
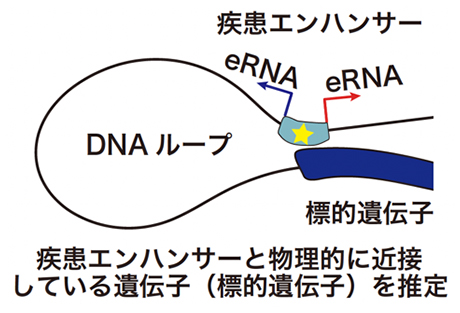
図4 疾患エンハンサーの標的遺伝子同定
エンハンサーは物理的に近接している標的遺伝子を活性化する。ゲノムの3次元構造を高い塩基解像度で決定できるMicro-C法を用いて、それぞれの疾患エンハンサーの標的遺伝子を網羅的に推定した。
今後の期待
自己免疫疾患やアレルギー疾患の多くは、どのような分子メカニズムで発症するのか十分に分かっておらず、治療が困難な場合も多くあります。本研究では、免疫疾患発症に関わる分子を疾患横断的に解析することに成功しました。その結果、既知の分子だけでなく、新規の分子を数多く見つけました。これにより、免疫疾患が発症する詳細な分子メカニズムの理解が加速的に進み、新しい治療標的分子が見いだされることが期待されます。
さらに、1細胞レベルで多様なヘルパーT細胞を解析し、これまで報告されていない新しい亜集団を数多く同定しました。こうした新規のヘルパーT細胞の発見により、新しい免疫システムの理解、さらには新しいがん免疫療法の開発につながる可能性があります。
本研究では、独自の1細胞解析法を開発し、ヘルパーT細胞の多様性を解きながら、自己免疫疾患やアレルギー疾患の理解に役立てました。同様のアプローチは、生活習慣病、心血管疾患、精神疾患などに応用することができ、今後いろいろな疾患に対して、新しい革新的医療を創出することが期待されます。
補足説明
1.ヘルパーT細胞
免疫応答に関与するリンパ球、T細胞の一種。抗原の情報をB細胞へ伝えて抗体の産生を誘導したり、免疫応答を誘導する液性因子を放出したりすることにより、免疫反応の司令塔として働く。
2.プロモーター、エンハンサー、エンハンサーRNA
プロモーターとエンハンサーはどちらも遺伝子発現を制御する機能を持つDNA配列。ゲノムDNA上で遺伝子の転写開始点の近くにあり、遺伝子を発現させる機能を持つ部分をプロモーターという。これに対し、遺伝子の上流や下流の遠方に位置し、遺伝子の転写効率を高める部分をエンハンサーという。活性化された状態のエンハンサーの両端からも転写が起きていることが分かっており、それをエンハンサーRNA(eRNA)という。
3.遺伝的変異
ヒトゲノムは30億塩基対のDNAから成るが、個々人を比較するとそのうちの0.1%の塩基配列の違いがある。これを遺伝子多型という。遺伝子多型のうち一つの塩基が、ほかの塩基に変わるものを一塩基多型と呼ぶ。ここでは、主に一塩基多型の意味で遺伝的変異という言葉を使用している。
4.ノンコーディング領域
ゲノム配列のうち、タンパク質を構成するアミノ酸配列をコードする領域と異なり、タンパク質をコードしない領域の総称。エピジェネティクス(塩基配列に依存しない遺伝子の調節機構)や転写、翻訳といった生物の活動の中枢を成す反応、幹細胞性の維持など、さまざまな働きに関与するノンコーディング領域が次々に報告されており、その重要性に注目が集まっている。
5.FANTOM
理研が中心となり、約20カ国、114の研究機関が参加する国際研究コンソーシアム。理研のマウスゲノム百科事典プロジェクトで収集された完全長cDNAの機能注釈(アノテーション)を行うことを目的に、理研ゲノム科学総合研究センター(当時)が中心となり2000年に結成された。役割は、トランスクリプトーム(転写産物)解析の分野を軸に発展・拡大してきた。また、プロジェクトの研究対象は、ゲノムの転写産物という「要素」の理解から、転写制御ネットワークという「システム」つまり「生命体のシステム」の理解へと発展し、得られた知見を基礎・応用の両面で有用なリソースとして公開している。FANTOMは、Functional ANnoTation Of Mammalian genomeの略。
6.キャップ構造
真核生物のmRNAの5’末端に特徴的な構造。核酸の重合は通常5’→3’で結合が進むが、mRNAの5’末端は修飾反応によりメチル化グアノシンが5′-5’結合となっている。mRNAの5’末端を保護してmRNAを安定化したり、タンパク質への翻訳効率を調整したりする。
7.ゲノムワイド関連解析
形質(疾患の有無や体質の特徴など)と関連する遺伝的特徴を知るための手法で、一塩基多型を用いて解析するのが一般的である。多人数のゲノム情報を調べて、形質と関連する遺伝的特徴を解析する。
8.ヌクレオソーム
細胞内におけるゲノムDNAの最小構造単位。ヒストンと呼ばれるタンパク質に、約150~200塩基対のDNAがおよそ1周半巻き付くことで形成される。
9.Micro-C法
全ゲノム解析手法の一つ。マイクロコッカルヌクレアーゼ(MNase)を用いてゲノムを切断することで、制限酵素を用いる従来のHi-C法より、高い塩基解像度でゲノムの三次元構造を予測できる。
国際共同研究グループ
理化学研究所 生命医科学研究センター
理研-IFOMがんゲノミクス連携研究チーム
チームリーダー 村川 泰裕(ムラカワ・ヤスヒロ)
(京都大学高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)教授、京都大学大学院医学研究科 システムゲノム医学分野 教授)
リサーチアソシエイト 小口 綾貴子(オグチ・アキコ)
(京都大学高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)特任研究員)
客員研究員 小松 秀一郎(コマツ・シュウイチロウ)
リサーチアソシエイト 孫 楽(ソン・ラク)
(京都大学高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)特任研究員)
客員研究員 竹内 一博(タケウチ・カズヒロ)
(京都大学高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)大学院生、京都大学大学院医学研究科 システムゲノム医学分野 大学院生)
大学院生リサーチ・アソシエイト 平井 公也(ヒライ・トモヤ)
(横浜市立大学 医学部 消化器・腫瘍外科学 大学院生)
テクニカルスタッフⅠ 佐野 浩美(サノ・ヒロミ)
自己免疫疾患研究チーム
チームリーダー 山本 一彦(ヤマモト・カズヒコ)
(生命医科学研究センター センター長)
上級研究員 鈴木 亜香里(スズキ・アカリ)
上級テクニカルスタッフ 明前 敬子(ミョウゼン・ケイコ)
上級テクニカルスタッフ 井上 尭(イノウエ・ギョウ)
ゲノム免疫生物学理研白眉研究チーム
理研白眉研究チームリーダー
ニコラス・パリッシュ(Nicholas Parrish)
基礎科学特別研究員 小嶋 将平(コジマ・ショウヘイ)
ゲノム解析応用研究チーム
チームリーダー 寺尾 知可史(テラオ・チカシ)
(静岡県立総合病院 臨床研究部 免疫研究部長、静岡県立大学 薬学部ゲノム病態解析講座 特任教授)
研究員 石川 優樹(イシカワ・ユウキ)
研究パートタイマーⅠ 田中 奈緒(タナカ・ナオ)
ヒト免疫遺伝研究チーム
チームリーダー 石垣 和慶(イシガキ・カズヨシ)
トランスクリプトーム研究チーム
チームリーダー ピエロ・カルニンチ(Piero Carninci)
(ミラノ Human Technopole 部門長)
東京都医学総合研究所 ゲノム医学研究センター
副センター長 川路 英哉(カワジ・ヒデヤ)
(理化学研究所 生命医科学研究センター 生命医科学大量データ技術研究チーム 客員主管研究員)
イタリア分子癌研究所(IFOM: Istituto Fondazione di Oncologia Molecolare ETS)
主任研究員 マッシミリアーノ・パガーニ(Massimiliano Pagani)
(ミラノ大学 教授)
研究員 ラウール・ジャン・ピエール・ボナール(Raoul Jean Pierre Bonnal)
東京大学医科学研究所 癌・細胞増殖部門
助教 小井土 大(コイド・マサル)
株式会社ダナフォーム
代表取締役社長 竹上 雄治郎(タケガミ・ユウジロウ)
代表取締役 林﨑 良英(ハヤシザキ・ヨシヒデ)
部員 金丸 愛(カネマル・アイ)
部員 山口 格(ヤマグチ・イタル)
京都大学高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)
特任助教 シュルティー・バーガット(Shruti Bhagat)
京都大学大学院医学研究科
腎臓内科学
教授 柳田 素子(ヤナギタ・モトコ)
血液・腫瘍内科学
教授 髙折 晃史(タカオリ・アキフミ)
免疫細胞生物学
教授 上野 英樹(ウエノ・ヒデキ)
准教授 吉富 啓之(ヨシトミ・ヒロユキ)
臓器連関研究チーム
特定准教授 河岡 慎平(カワオカ・シンペイ)
(東北大学 加齢医学研究所 生体情報解析分野 准教授)
特定助教 小西 理予(コニシ・リヨ)
放射線遺伝学分野
名誉教授 武田 俊一(タケダ・シュンイチ)
(深セン大学(中国)医学部 教授)
横浜市立大学医学部 消化器・腫瘍外科学
教授 遠藤 格(エンドウ・イタル)
三重大学大学院医学系研究科 腎泌尿器外科
教授 井上 貴博(イノウエ・タカヒロ)
大学院生 関戸 翔(セキト・ショウ)
(京都大学大学院 医学研究科 システムゲノム医学分野 特別研究学生)
九州大学大学院医学研究院 プレシジョン医療学分野
助教 平林 茂樹(ヒラバヤシ・シゲキ)
カロリンスカ研究所(スウェーデン)
教授 ユハ・ケーレ(Juha Kere)
(ヘルシンキ大学(フィンランド)教授)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業若手研究(A)「エンハンサー同定の新技術を開発し、腫瘍に認める遺伝子発現異常の原因を解明する(研究代表者:村川泰裕)」、基盤研究(A)「新規技術による白血病の包括的エンハンサー解析と分子病態解明(研究代表者:村川泰裕)」、挑戦的研究(萌芽)「1細胞エンハンサー解析法の確立による新たな機能性ゲノム学の開拓(研究代表者:村川泰裕)」、国際共同研究加速基金(国際共同研究強化(B))「発癌関連転写制御領域(エンハンサー)の網羅的同定の為の国際共同研究(研究代表者:村川泰裕)」、特別研究員奨励費「ゲノムエンハンサーの作動原理の解明(研究代表者:村川泰裕)」、基盤研究(S)「多因子疾患における疾患リスク遺伝子多型を用いた病態解析に関する新しい方法論の確立(研究代表者:山本一彦)、基盤研究(A)「エンハンサーの遺伝的発現制御の解明による免疫疾患解析(研究代表者:寺尾知可史)、若手研究「DNA配列機械学習モデルの予測根拠に基づく非翻訳RNAの発現特異性の解明(研究代表者:小井土大)、日本医療研究開発機構(AMED)「腎臓病において組織障害と修復を制御する微小環境の解明と医学応用(研究代表者:柳田素子)」「組織リンパ球分画解析に基づくヒト疾患ゲノム・薬剤情報を利活用した自己免疫疾患病態解明(研究代表者:吉富啓之)」「生体試料を用いた大規模機能ゲノミクス解析支援及びヒト免疫機能評価基盤の高度化(研究代表者:山本一彦)」「遺伝的多様性と機能に関するマルチオミックスを中心としたヒト免疫評価法の確立と支援の為のサポート機関(研究代表者:山本一彦)」「シングルセル統合ゲノミクス解析が解き明かす強皮症の病態基盤の開発(研究代表者:寺尾知可史)」「先天的/後天的構造多型に着目した免疫/精神疾患病態解明に関する研究開発(研究代表者:寺尾知可史)」「体細胞モザイクのがん発症および予後因子としての意義解明の開発(研究代表者:寺尾知可史)」「炎症性関節炎の統合ゲノミクス解析(研究代表者:寺尾知可史)」「先天的・後天的ゲノム情報と臨床情報を用いた関節リウマチの層別化基盤の構築(研究代表:寺尾知可史)」、科学技術振興機構(JST)戦略的創造研究推進事業(ERATO)「有田リピドームアトラスプロジェクト(研究代表者:有田誠)」、日本応用酵素協会成人病の病因・病態の解明に関する研究助成、武田科学振興財団生命科学研究助成を受けて行われました。
原論文情報
Akiko Oguchi†, Akari Suzuki†, Shuichiro Komatsu†, Hiroyuki Yoshitomi, Shruti Bhagat, Raku Son, Raoul Jean Pierre Bonnal, Shohei Kojima, Masaru Koido, Kazuhiro Takeuchi, Keiko Myouzen, Gyo Inoue, Tomoya Hirai, Hiromi Sano, Yujiro Takegami, Ai Kanemaru, Itaru Yamaguchi, Yuki Ishikawa, Nao Tanaka, Shigeki Hirabayashi, Riyo Konishi, Sho Sekito, Takahiro Inoue, Juha Kere, Shunichi Takeda, Akifumi Takaori-Kondo, Itaru Endo, Shinpei Kawaoka, Hideya Kawaji, Kazuyoshi Ishigaki, Hideki Ueno, Yoshihide Hayashizaki, Massimiliano Pagani, Piero Carninci, Motoko Yanagita, ITEC consortium, Nicholas Parrish, Chikashi Terao*, Kazuhiko Yamamoto*, Yasuhiro Murakawa*, “An atlas of transcribed enhancers across helper T cell diversity for decoding human diseases”, Science, 10.1126/science.add8394
発表者
理化学研究所
生命医科学研究センター 理研-IFOMがんゲノミクス連携研究チーム
チームリーダー 村川 泰裕(ムラカワ・ヤスヒロ)
(京都大学 高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)教授)
リサーチアソシエイト 小口 綾貴子(オグチ・アキコ)
(京都大学 高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)特任研究員)
客員研究員 小松 秀一郎(コマツ・シュウイチロウ)
自己免疫疾患研究チーム
チームリーダー 山本 一彦(ヤマモト・カズヒコ)
(生命医科学研究センター センター長)
上級研究員 鈴木 亜香里(スズキ・アカリ)
ゲノム解析応用研究チーム
チームリーダー 寺尾 知可史(テラオ・チカシ)
報道担当
理化学研究所 広報室 報道担当
京都大学高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)リサーチ・アクセラレーション・ユニット