新規の器官再生研究モデル生物イベリアトゲイモリ
2019-04-24 基礎生物学研究所
【本研究成果のポイント】
- 新規モデル生物#1イベリアトゲイモリ#2の遺伝子カタログを作成した。
- 遺伝子カタログをはじめとする様々なイベリアトゲイモリ研究情報を世界中の研究者が利用できるようにするためのポータルサイ“iNewt”(http://www.nibb.ac.jp/imori/main/)を開設した。
- 本研究の成果は、イモリの高い器官再生能力の解明をはじめとする様々な研究に不可欠なツールやヒントとなり、今後の再生医療研究を含む多様な分野への貢献が期待される。
【研究の概要】
両生類のイモリは、非常に高い再生能力を持っていることで知られ、再生医学や発生生物学における重要な実験動物として1世紀以上の研究の歴史を持っています。なかでも、繁殖や飼育が簡便なイベリアトゲイモリ(Pleurodeles waltl)は新しいモデル生物として脚光を浴びています。実験室での飼育や繁殖が容易なイベリアトゲイモリの有用性に着目した日本人の研究者でコンソーシアムを作り、飼育システムの確立、近交系の確立、高効率のゲノム編集法の開発など研究基盤の構築を推進してきました。現在では、イベリアトゲイモリは画期的な新興モデル生物として世界中の研究者から注目を浴びており、研究者人口が急速に増えています。しかし、イベリアトゲイモリには、ゲノムが巨大などの理由で研究の基盤となる遺伝子の情報がほとんど整備されていないという問題がありました。今回、基礎生物学研究所の重信秀治教授ら、鳥取大学医学部の林利憲准教授(現 広島大学教授)ら、琉球大学大学院医学研究科の松波雅俊助教ら、ほか広島大学、中央大学、産業技術総合研究所、九州大学、学習院大学の研究者から構成される研究チームは、イベリアトゲイモリの網羅的遺伝子カタログ作成に成功しました。各研究室から持ち寄った29種類もの多様なイベリアトゲイモリ試料からRNAを抽出し、次世代シーケンシング#3技術によるRNA-seq#4法により遺伝情報を解読しました。得られた配列情報を大型計算機で解析することにより、202,788個のイベリアトゲイモリの遺伝子モデル#5を構築しました。これらのモデルを検証したところ、イベリアトゲイモリが保有する全遺伝子の約98%をカバーする網羅性の高い高品質の遺伝子カタログであることが確認されました。さらに、研究チームは、この遺伝子カタログを世界中の研究者と共有するためのポータルサイト“iNewt”(http://www.nibb.ac.jp/imori/main/)を開設しました。iNewtでは遺伝子カタログに加えて、ゲノム編集のプロトコルなどイベリアトゲイモリの研究リソース情報を無料で提供しています。本研究によって作成された遺伝子カタログを利用することで、ゲノム編集や、次世代シーケンサーを用いた遺伝子の発現解析など、イベリアトゲイモリを用いた研究が格段にスピードアップすることが期待されます。これにより、再生医療や発生生物学はもちろん、癌研究、幹細胞生物学、生殖生物学、進化学、毒性学などの研究分野でイモリを活用した研究が大きく発展することが期待されます。
本研究成果は、2019年4月22日に国際科学誌「DNA Research」に掲載されました。

イベリアトゲイモリ
【研究の背景】
有尾両生類#6の仲間であるイモリは、発生生物学や再生医療のモデル生物として長い研究の歴史を持っています。脊椎動物の胚発生において重要な役割を担う部位として知られている「シュペーマンオーガナイザー」は、イモリの胚を用いた実験により発見されました。この業績によりシュペーマン博士は、1935年にノーベル生理学・医学賞を受賞しています。また、イモリは失われたり傷ついたりした組織や器官を元通りに修復する、非常に高い再生能力を持っています。1895年にドイツのウォルフ博士が、イモリの眼から水晶体(レンズ)を除去すると虹彩の細胞が変化してレンズが再形成されることを発見して以来、数多くの研究者がイモリを使って再生の研究に取り組んできました。イモリは四肢、心臓、脳など、ほとんどの器官を再生することができます。
このように、イモリは発生生物学や再生医学の研究に大きな貢献を果たしてきましたが、これまで研究に使用されてきたイモリの種(アカハライモリなど)は、飼育や繁殖に手間がかかり、性成熟するまで成長させるには多くの労力と時間が必要であり、必ずしも使い易い実験動物ではありませんでした。そこで研究チームは、実験室での飼育や繁殖が容易なイベリアトゲイモリ(Pleurodeles waltl)に着目し、飼育システムの確立、近交系の確立、高効率のゲノム編集法の開発など研究基盤の構築を推進してきました。現在では、イベリアトゲイモリは画期的な新興モデル生物として世界中の研究者から注目を浴びており、研究者人口が急速に増えています。
イベリアトゲイモリをモデル生物として確立し、それを利用した研究を加速するには、遺伝情報の整備が欠かせません。しかしながら、イモリの仲間はヒトよりも数倍以上大きなゲノムを持つことからゲノム研究が困難であり、遺伝情報の整備が立ち遅れていました。そこで今回RNA-seq法という新しい遺伝子解析技術を使い、イベリアトゲイモリの遺伝子のカタログ化を目指しました。イベリアトゲイモリを扱っている日本各地の研究室が協力してそれぞれのRNA-seqデータを持ち寄り、統合して解析することによって、極めて網羅性の高い遺伝子カタログの構築に成功しました。本研究は、基礎生物学研究所の重信秀治教授、内山郁夫助教、鳥取大学医学部の林利憲准教授(現 広島大学教授)、竹内隆教授、琉球大学大学院医学研究科の松波雅俊助教、広島大学の鈴木賢一特任准教授(基礎生物学研究所クロスアポイントメント)、中央大学の福井彰雅教授、産業技術総合研究所創薬基盤研究部門の原本悦和主任研究員、九州大学の田代康介准教授、学習院大学の阿形清和教授(現 基礎生物学研究所長)などから構成される研究チーム(語句説明#7参照)により行われました。
【研究の成果】
イベリアトゲイモリの様々な器官や発達段階の異なる胚など、のべ29種類の試料からRNAを抽出し、次世代シーケンシング技術を用いたRNA-seq法により、遺伝子情報の大量解読を行いました(合計で約12億塩基)(図1)。このデータから、大型計算機で解析することにより、202,788個のイベリアトゲイモリの遺伝子モデルを構築しました。検証の結果、研究チームが構築した遺伝子カタログは、イベリアトゲイモリの全遺伝子の約98%をカバーするであろう、高品質のものであることが確認されました。また、これらの遺伝子の組織ごとの発現量を定量しました。発現パターンをもとにデータマイニングを行ったところ、イベリアトゲイモリの遺伝子はその発現パターンから19のグループに分類することができ、それらは特定の組織・器官、発生時期や再生過程に特異的にはたらく機能単位であることが推定されました(図2)。
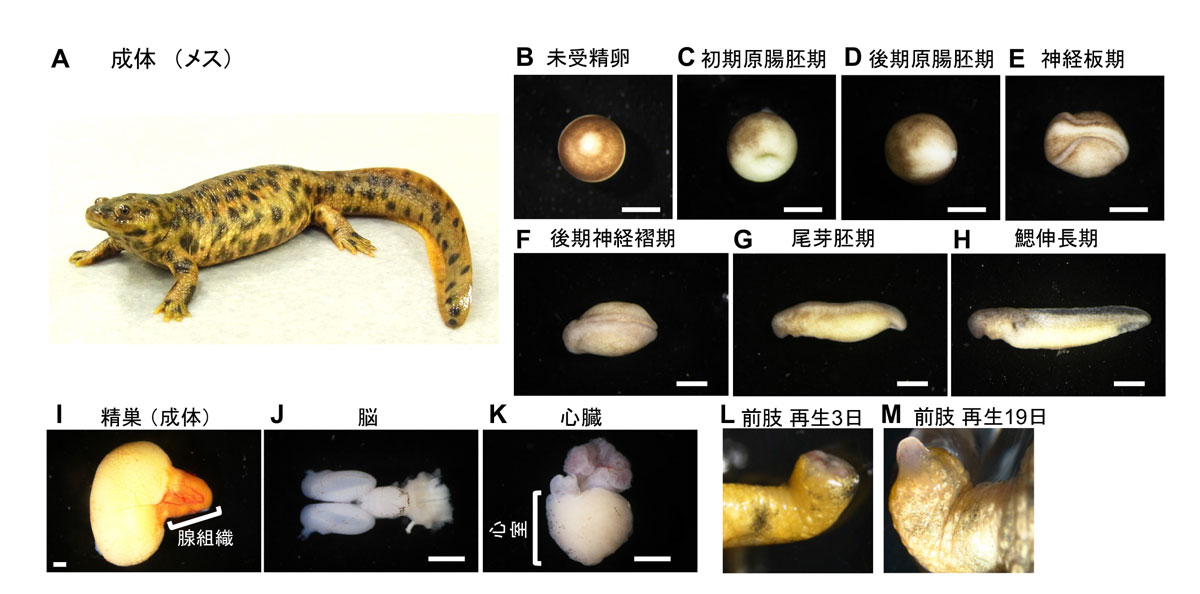
図1:イベリアトゲイモリの全身像(A)とRNAを採取した卵と胚(B-H)および組織(I-M)の例。白線は1 mmを示す。
得られたイモリの遺伝子カタログを他の脊椎動物と比較することで、イモリの遺伝子レパートリーの特徴や進化の過程が明らかになりました(図2)。イモリの遺伝子をその配列の類似性から15,923のグループに分類した上で、ヒトの遺伝子レパートリーと比較したところ、83%はヒトと共通であることがわかりました。一方、イモリに特徴的な遺伝子は660グループでした。これらはイモリの系統で独自に獲得された遺伝子である可能性が高く、イモリの驚くべき再生能力を理解する上で注目に値する遺伝子群であると言えます。
遺伝子カタログと発現量の情報からイモリの体作りに関わる遺伝子とその進化の一端も明らかになりました。得られたデータを使用して、hox#8やbmp#9など動物の体作りに重要な遺伝子の発現量のパターンや他の動物との遺伝子の相同性を解析した結果、脊椎動物間における進化的な対応関係を示すことができました。例えば、多くの脊椎動物で体作りについて重要な役割を担っているbmp4がイモリでは失われている可能性が示唆されました。このような進化がイモリの独自の形質に寄与しているのかもしれません。
さらに、四肢の再生時に発現している遺伝子について調べました。これまでの研究で既に四肢再生時に発現することが知られていた遺伝子群が確かに検出された一方で、今回の研究で初めて見出された再生特異的遺伝子もありました。中でも、再生期に多種類のリボソームタンパク質遺伝子群が発現していることは興味深い発見です。これらのリボソームタンパク質は、再生において重要な機能を担っている可能性があります。
以上の結果より、研究チームが作成したイベリアトゲイモリの遺伝子カタログとデータベースの有用性が確かめられました。これらの遺伝子カタログや関連データベースを世界中の研究者と共有するために、研究チームはポータルサイト“iNEWT”(http://www.nibb.ac.jp/imori/main/)を開設しました。iNEWTは遺伝子カタログなど研究チームが解析したデータを無料でダウンロードできるだけでなく、遺伝子検索の機能も備えています。遺伝子情報のみならず、ゲノム編集のプロトコルなど、イベリアトゲイモリの研究に必要な各種情報も提供しています。
【今後の展開】
本研究によって作成された遺伝子カタログやデータベースを利用することで、イベリアトゲイモリを用いた研究が格段にスピードアップされます。特にCRISPR-Cas9を用いたゲノム編集や、次世代シーケンサーを用いた遺伝子の発現解析に必要な遺伝子の情報が直ちに入手可能となるため、これらの技術と組み合わせることにより、イモリの器官再生能力に関与する遺伝子や疾患に関与する遺伝子を迅速かつ簡便に解析することが可能となりました。
このように本成果で得られたツールや知見は、再生医療をはじめとするイモリを活用した研究への大きな貢献が期待されます。
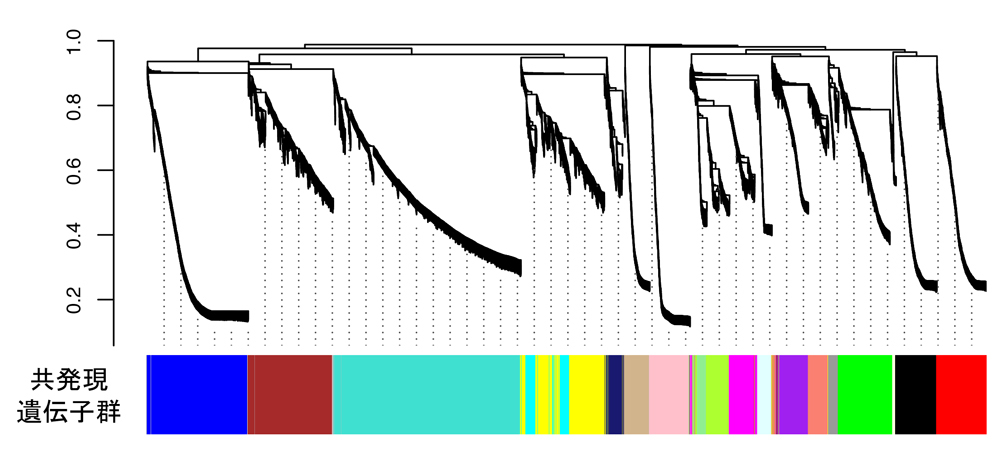
図2:遺伝子の共発現解析。発現パターンによって19のグループに分類された。
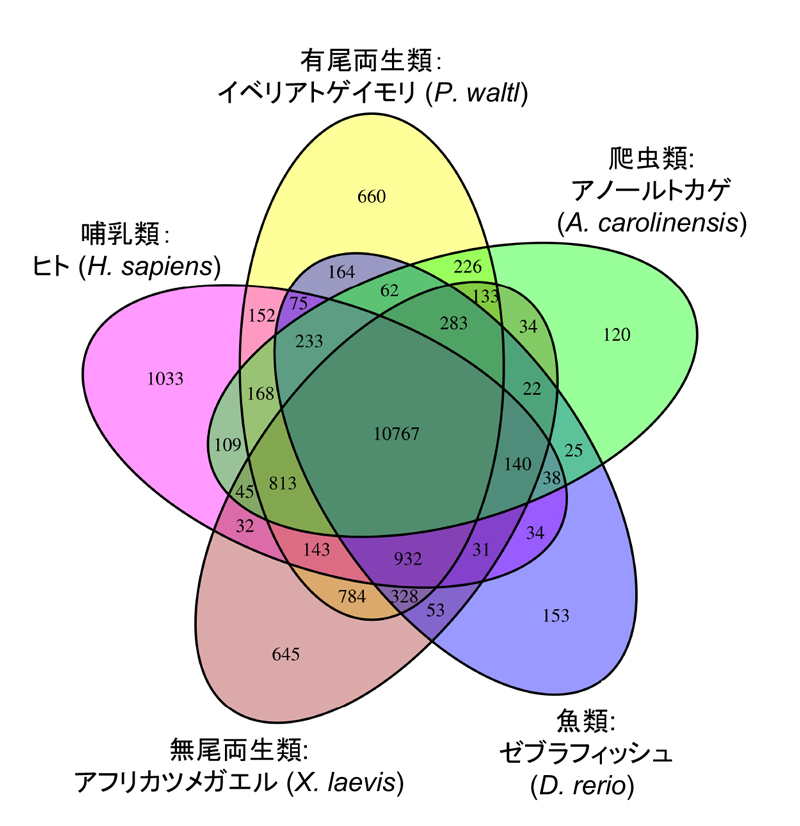
図3:イベリアトゲイモリとヒトなど他の脊椎動物との遺伝子レパートリー比較。
【語句説明】
#1 モデル生物
研究者が解明したい生命現象を研究するために、解析しやすい特徴を備えた生物。一般に、飼育や繁殖が容易、実験手法が確立されている、遺伝情報が整備されているなどの特徴を持つ。代表的なモデル生物として、大腸菌、酵母、マウス、メダカなどが知られている。特定のモデル生物を多くの研究者が共通して研究することにより、情報の共有や知見の統合が容易となる。
#2 イベリアトゲイモリ (Pleurodeles waltl)
スペインのイベリア半島原産の大型のイモリ(最大30cmになる)。一年以内に成体になり、年間を通じて大量の卵を産む。
#3 次世代シーケンシング
2000年代後半から発展してきた新規の塩基配列解読技術。超並列にシーケンス反応を行うことで、数億断片を一度に読むことができる画期的なDNAシーケンス技術である。従来の方法に比べて安価でより大規模な塩基配列が解読できる。
#4 RNA-seq
次世代シーケンシング技術によって網羅的に転写産物を解読する手法。得られた配列情報から遺伝子の同定やカタログ化ができるだけでなく、遺伝子発現量の定量も可能である。
#5 遺伝子モデル(の構築)
本研究では以下の方法で、RNA-seqの配列情報から遺伝子構造を推定(遺伝子モデルの構築)した。本研究で用いた次世代シーケンサーはショートリード型シーケンサーと呼ばれ、この手法で取得できる遺伝子情報はたかだか100塩基程度の長さの短い断片である。通常遺伝子の転写産物の長さは2〜4千塩基あるので、短い断片をつないで元の構造を再構築する作業が必要となる。この過程をアセンブルと呼び、再構築されたひとつながりの配列をコンティグと呼ぶ。次に、これらのコンティグから、タンパク質をコードする領域(オープンリーディングフレーム)を推定する。本研究では、次世代シーケンサーから得られた約12億個の配列断片から、1,395,387個のコンティグを構築し、さらにその中に、202,788種類のタンパク質をコードする遺伝子の構造を推定した。
#6 有尾両生類
サンショウウオやアホロートル(ウーパールーパー)のように尾を持った両生類のグループを指す。カエルのグループは無尾両生類と呼ばれる。
#7 イベリアリサーチコンソーシアム
本研究を契機にイベリアリサーチコンソーシアムを発足しました。イベリアトゲイモリの研究リソースの充実化と研究者コミュニティ育成を目的としています。
URL: http://www.nibb.ac.jp/imori/main/
#8 hox
動物の体を構成する各細胞が、体のどこに位置するかを決める遺伝子。脊椎動物のゲノムでは、約10個の連続したHox遺伝子からなる、4つの集団が保存されている。
#9 bmp
初期発生と組織再生の両方で重要な役割をもつ分泌性タンパク質で、bmp2、bmp4、bmp7などの複数の遺伝子が見つかっている。中でもBMP4タンパク質は、異なる動物種間でそのアミノ酸配列がたいへん似ているため、より重要ではないかと考えられていた。
【論文情報】
掲載誌:
国際科学誌 DNA Research
タイトル:
“A comprehensive reference transcriptome resource for the Iberian ribbed newt Pleurodeles waltl, an emerging model for developmental and regeneration biology”
著者名:
Masatoshi Matsunami1, Miyuki Suzuki2, Yoshikazu Haramoto3, Akimasa Fukui4, Takeshi Inoue5, Katsushi Yamaguchi6, Ikuo Uchiyama6, Kazuki Mori3, Kosuke Tashiro7, Yuzuru Ito3, Takashi Takeuchi8, Ken-ichi T Suzuki2, 6, Kiyokazu Agata5, Shuji Shigenobu6*, and Toshinori Hayashi8*
所属: 1. 琉球大学大学院医学研究科、2. 広島大学大学院理学研究科、3. 産業技術総合研究所、4. 中央大学理工学部生命科学科、5. 学習院大学理学部生命科学科、6. 基礎生物学研究所、7. 九州大学大学院、8.鳥取大学医学部生命科学科、*: 責任著者
DOI:
10.1093/dnares/dsz003 (https://doi.org/10.1093/dnares/dsz003)
【研究サポート】
本研究は文部科学省科学研究費助成事業(JP16H06376 to K.A., JP16H0125 and JP16K08467 to T.H., JP17J04796 to M.S., JP16K18613 to M.M., JP17K14980 to Y.H., JP16H04794 to T.T., and JP15K06802 to K.T.S.)のサポートを受けて実施されました。また、基礎生物学研究所のモデル生物・技術開発共同利用研究の一環として実施されました。
【お問い合わせ先】
基礎生物学研究所 生物機能解析センター
教授 重信 秀治(しげのぶ しゅうじ)
鳥取大学医学部生命科学科生体情報機能学講座生体情報学分野
准教授 林 利憲(はやし としのり)(現 広島大学両生類研究センター 教授)
琉球大学大学院医学研究科先進ゲノム検査医学講座
助教 松波 雅俊(まつなみ まさとし)
広島大学大学院統合生命科学研究科
基礎生物学研究所 新規モデル生物開発センター
特任准教授 鈴木 賢一(すずき けんいち)
中央大学理工学部生命科学科
教授 福井 彰雅(ふくい あきまさ)
産業技術総合研究所 創薬基盤研究部門
主任研究員 原本 悦和(はらもと よしかず)
学習院大学理学部生命科学科
教授 阿形 清和(あがた きよかず)(現 基礎生物学研究所長)
助教 井上 武(いのうえ たけし)
【報道担当】
基礎生物学研究所 広報室
鳥取大学 米子地区事務部総務課広報係
琉球大学 総務部総務課広報係
広島大学 広報グループ
中央大学 広報室
産業技術総合研究所 企画本部報道室
学習院大学 広報センター