2018-01-05 国立大学法人東京大学,国立研究開発法人日本医療研究開発機構
発表者
河岡 義裕(東京大学医科学研究所 感染・免疫部門 ウイルス感染分野 教授)
野田 岳志(京都大学 ウイルス・再生医科学研究所 微細構造ウイルス学 教授)
発表のポイント
- インフルエンザウイルスが子孫ウイルスにゲノム(遺伝情報)を伝える仕組みを解明した。
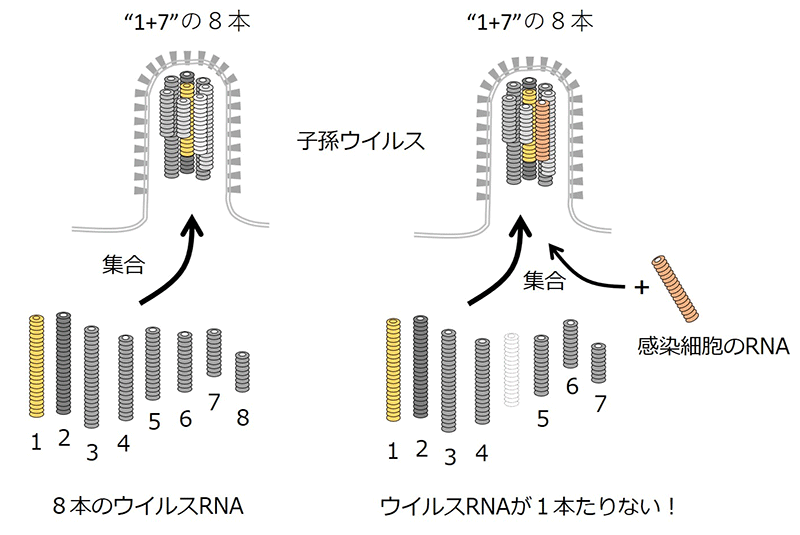
- 子孫ウイルスにゲノムを伝えるとき、8本のウイルスRNAを“1+7” という特徴的な配置(中心の1本のRNAを7本のRNAが取り囲む配置)に集合させる過程が重要であることがわかった。さらに、ウイルスRNAが足りないときには、”1+7” 配置にまとめるために感染細胞のRNAを奪う仕組みがあることがわかった。
- インフルエンザウイルスの遺伝に関する巧妙な仕組みを解明した点が重要であり、今後はウイルスRNAの集合を阻害する新規抗インフルエンザ薬の開発につながることが期待される。
発表概要
河岡義裕教授(東京大学 医科学研究所)と野田岳志教授(京都大学 ウイルス・再生医科学研究所)らの研究グループは、インフルエンザウイルスが子孫ウイルスに遺伝情報を伝える仕組みを明らかにしました。
あらゆる生物において、子は親からゲノム(遺伝情報)を受け継ぎます。ウイルスも同じく、子孫ウイルスは親ウイルスからゲノムを受け継ぎます。インフルエンザウイルス(注1)は8本のRNAをゲノムとして持っていますが、8本に分かれたRNAがどのように子孫ウイルス粒子に伝えられるか、その仕組みの詳細は明らかにされていませんでした。
本研究グループは以前、子孫インフルエンザウイルス粒子の中のRNAの解析を行い、子孫ウイルスが“1+7” という特徴的な配置(中心の1本のRNAを7本のRNAが取り囲む配置)をとった8本のRNAを取り込むことを明らかにしました(図1左)。今回はさらに、ウイルスRNAを1本欠き7本しかRNAを持たない変異子孫ウイルスにも、“1+7” 配置にまとめられた8本のRNAが取り込まれることを明らかにしました(図1右)。興味深いことに、8本目のRNAとして取り込まれたのはインフルエンザウイルスのRNAではなく、感染細胞のリボソームRNA(注2)でした(図1右)。
今回の発見から、インフルエンザウイルスが子孫ウイルスにゲノムを伝えるとき、8本のRNAを“1+7” に集合させる過程が重要であることがわかりました。さらに、ウイルスのRNAが足りないときには、“1+7” 配置にまとめるために感染細胞のRNAを奪う仕組みを持つことが明らかになりました。本成果は、ウイルスRNAの集合を標的とした新規抗インフルエンザ薬の開発に繋がることが期待されます。
本研究成果は、2018年1月4日(日本時間午後7時)に英国科学雑誌「Nature Communications」で公開されました。なお本研究は、東京大学、京都大学、米国ウィスコンシン大学が共同で行ったものです。本研究成果は、日本医療研究開発機構(AMED)新興・再興感染症に対する革新的医薬品等開発推進研究事業、AMED革新的先端研究開発支援事業、文部科学省新学術領域研究「ネオウイルス学:生命源流から超個体、そしてエコ・スフィアへ」「ウイルス感染現象における宿主細胞コンピテンシーの分子基盤」、科学技術振興機構さきがけ「ライフサイエンスの革新を目指した構造生命科学と先端的基盤技術」などの一環として得られました。
発表内容
➀研究の背景・先行研究における問題点
種を存続させるためには、ウイルスもゲノム(遺伝情報)を子孫ウイルスへと伝えなければいけません。インフルエンザウイルスは8本に分かれたRNAをゲノムとして持ちます。したがって、感染細胞から新たに作られるウイルス(子孫ウイルス)は、その粒子の中に8本に分かれたRNAをもれなく取り込む必要があります。しかしそのメカニズムの詳細はわかっておらず、8本のRNAを効率よく子孫ウイルスに取り込ませるような特別な仕組みがあるのかどうか、長年の論争となっていました。本研究グループは以前、電子顕微鏡解析(注3)によって、8本のウイルスRNAが“1+7”という特徴的な配置(中心に1本のRNAがあり、7本のRNAが中心のRNAを囲むような配置:図2)に集められ子孫ウイルス粒子に取り込まれることを明らかにしました(Noda et al. Nature, 2006: Noda et al. Nature Communications, 2012)。しかし、ウイルスRNAが“1+7” という配置をとることにどのような意義があるのか、よくわかっていませんでした。
➁研究内容
本研究では、次世代シークエンス解析(注4)と電子顕微鏡解析によって、子孫インフルエンザウイルス粒子の中に取り込まれているRNAを調べました。通常の子孫ウイルス粒子には8本のウイルスRNAが“1+7” という配置をとって取り込まれていました。また、ウイルスRNA以外のRNA(感染細胞のRNA)は、ほとんど取り込まれていませんでした。次にリバースジェネティクス法(注5)を用いて、ウイルスRNAを1本欠き7本しかウイルスRNAを持たない変異ウイルスを人工合成し、その変異ウイルス粒子の中に取り込まれたRNAを解析しました。予想に反して、7本しかウイルスRNAを持たない変異ウイルスにも、“1+7”に束ねられた8本のRNAが取り込まれていました。興味深いことに、この変異ウイルスに取り込まれた8本目のRNAは、インフルエンザウイルスのRNAではなく、感染細胞に存在するリボソームRNAでした。今回の発見から、インフルエンザウイルスが子孫ウイルスにゲノムを伝えるとき、8本のRNAを“1+7” に集合させるステップが重要であることがわかりました。さらに、ウイルスのRNAが足りないときには、“1+7” に集合させるために細胞のリボソームRNAを奪い取る仕組みを持つことが明らかになりました。
➂社会的意義・今後の予定
本成果は、インフルエンザウイルスの遺伝に関する巧妙な仕組みを明らかにしたという点で重要であり、インフルエンザウイルスの増殖機構の理解に大きな知見を与えました。今後、ウイルスRNAが“1+7” に束ねられるメカニズムがより詳細に明らかにされたら、インフルエンザウイルスのRNAの集合を標的とした(すなわちインフルエンザウイルスの8本のRNAの集合を阻害するような)新しい作用機序の抗インフルエンザ薬の開発へと繋がることが期待されます。
発表雑誌
- 雑誌名:
- Nature Communications(1月4日オンライン版)
- 論文タイトル:
- Importance of the 1+7 configuration of ribonucleoprotein complexes for influenza A virus genome packaging
- 著者:
- Takeshi Noda*, Shin Murakami, Sumiho Nakatsu, Hirotaka Imai, Yukiko Muramoto, Keiko Shindo, Hiroshi Sagara, Yoshihiro Kawaoka*
用語解説
- 注1:インフルエンザウイルス
- インフルエンザの原因となるウイルス。A型インフルエンザウイルスとB型インフルエンザウイルスが毎冬にヒトで流行する。A型ウイルスとB型ウイルスは、8つに分節化した一本鎖マイナス鎖RNAをゲノムとして持つ。
- 注2:リボソームRNA
- 細胞のタンパク質合成を担うリボソームを構成するRNA。通常はrRNAと表記される。真核生物の細胞には28S rRNA、5.8S rRNA, 5S rRNA, 18S rRNAが存在する。
- 注3:電子顕微鏡解析
- 光ではなく電子を用いて試料を観察する顕微鏡。光学顕微鏡よりも分解能が高く、直径が1/10,000mm程度のインフルエンザウイルスの微細な構造も詳細に観察することができる。
- 注4:次世代シークエンス解析
- DNAやRNAなどの塩基配列情報を網羅的に解析する手法。DNAやRNAの塩基配列を同定するだけでなく、どれくらいの量の遺伝子が存在するかを定量的に解析することもできる。
- 注5:リバースジェネティクス法
- インフルエンザウイルスの8種類の遺伝子をコードするプラスミドDNAを培養細胞に導入することでインフルエンザウイルスを合成する方法。1種類のプラスミドDNAを欠損させることで、7本しかウイルスRNAを持たない変異インフルエンザウイルスを合成することができる。
添付資料
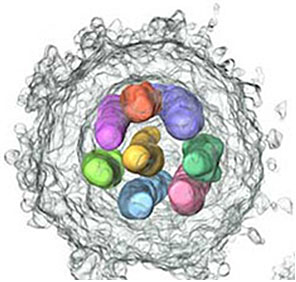
電子顕微鏡解析によって3次元的に作られた子孫ウイルス粒子とウイルスRNAのモデル図。
子孫ウイルス粒子の中には8本のウイルスRNAが存在する。
中心の1本のRNA(黄色)が7本のRNAに取り囲まれるように、“1+7“配置に束ねられている。
問い合わせ先
研究に関するお問い合わせ
東京大学医科学研究所 感染・免疫部門ウイルス感染分野
教授 河岡 義裕(カワオカ ヨシヒロ)
京都大学ウイルス・再生医科学研究所 微細構造ウイルス学分野
教授 野田 岳志(ノダ タケシ)
報道に関するお問い合わせ
東京大学医科学研究所 国際学術連携室 URA
国立研究開発法人日本医療研究開発機構(AMED)
経営企画部 企画・広報グループ
AMEDの事業に関するお問い合わせ
国立研究開発法人日本医療研究開発機構(AMED)
創薬戦略部 医薬品研究課
戦略推進部 感染症研究課