2021-12-02 理化学研究所,慶應義塾大学先端生命科学研究所
理化学研究所(理研)生命機能科学研究センター多階層生命動態研究チームの古澤力チームリーダー、慶應義塾大学先端生命科学研究所の福田真嗣特任教授らの共同研究チームは、糞便の顕微鏡画像から「腸内細菌叢[1]」の状態を推定する新たな手法を開発しました。
本研究成果は、腸内細菌叢の状態を調べ、その振る舞いを予測しコントロールする技術に応用可能なことから、健康維持や疾患の診断・予防・治療法の開発に貢献すると期待できます。
共同研究チームは、健康状態を反映して腸内細菌叢の状態が異なるマウスから糞便を採取し、その顕微鏡画像を入力データとして深層学習[2]に与えることで、糞便画像から腸内細菌叢の状態を推定する手法を開発しました。さらに、アンプリコンシーケンス解析[3]によって得られた腸内細菌叢の組成比を、糞便画像から高精度で予測することに成功しました。この手法によって、腸内細菌叢の状態を簡便かつ安価に推定することが可能になります。
本研究は、オンライン科学雑誌『iScience』(11月22日付)に掲載されました。
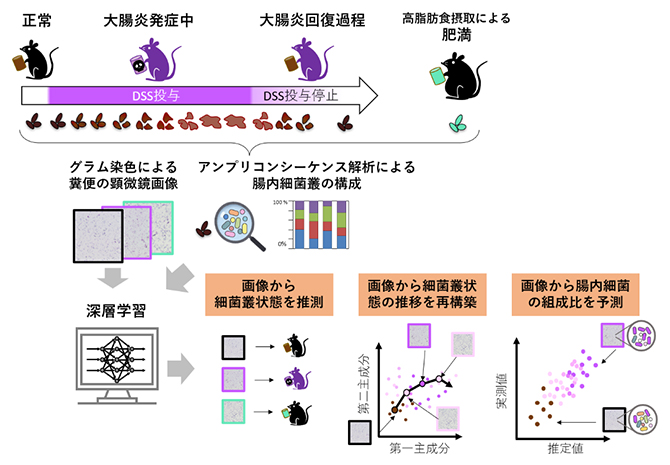
深層学習を活用した糞便画像から腸内細菌叢を推定する新手法
背景
哺乳類の腸内には、およそ1,000種類にも及ぶ多様な腸内細菌が生息しています。これら腸内細菌からなる生態系を「腸内細菌叢」と呼び、近年のさまざまな研究から、腸内細菌叢の組成は個人差が大きく、食生活や疾病などによって変化することが分かっています。また、腸内細菌叢がヒトの健康や生活の質に大きな影響を与えることも明らかになりつつあります。腸内細菌叢の状態を調べ、その変化を予測しコントロールする技術の開発は、医療やヘルスケア分野における重要な課題です。
腸内にどのような細菌がどれだけ存在するかを調べる手法として、腸内細菌ゲノムの特定の領域をPCR法[4]によって増幅し、そこに含まれる配列の種類とその数を調べる「アンプリコンシーケンス解析」が広く用いられています。ただしこの手法は、解析に次世代シーケンサー[5]を用いることから、比較的複雑な実験操作や長い分析時間、高いコストを要します。そのため、例えば個人の腸内細菌叢の日々の変化を調べるなど、多くのサンプルを迅速に解析する場合においては、必ずしも適した手法とはいえませんでした。
一方、機械学習・人工知能の技術の進展は、画像データからさまざまな情報を読み解くことを可能にしました。例えば、人間の目では同じように見える細胞の画像でも、適切に訓練された人工知能であればそれぞれの細胞がどのような状態にあるかを区別できます。実際に医療分野への応用も盛んに行われており、病理画像を深層学習などの人工知能に学習させることにより、さまざまながん組織の高精度な検出が可能になっています。近年、こうした機械学習・人工知能の技術を用いた腸内細菌叢の解析に期待が高まっています。
研究手法と成果
本研究では、深層学習を用いて糞便の顕微鏡画像から腸内細菌叢の状態を推定する新たな手法を開発しました。
共同研究チームはその有効性を示すために、腸内細菌叢の状態を変化させる処理を施したマウスを用意し、その糞便を採取しました。具体的には、大腸炎を誘発する薬剤であるデキストラン硫酸ナトリウム(DSS)[6]を5匹のマウスに投与し、大腸炎が進行していく過程(9日間)と、DSSの投与を停止して大腸炎から回復する過程(7日間)で糞便を採取しました。また別の処理として、高脂肪食を5週間与え、肥満を誘導した5匹のマウスから糞便を採取しました。これらの糞便サンプルに含まれる腸内細菌をグラム染色[7]により可視化し、低倍率の顕微鏡で画像を取得しました(図1)。

図1 マウス糞便の顕微鏡画像の例
大腸炎を誘発するデキストラン硫酸ナトリウム(DSS)投与前(a)とDSS投与後9日目(b)のマウス糞便をグラム染色し、顕微鏡画像を取得した。スケールは200マイクロメートル。図の画像は1360×1024ピクセルのサイズを持つが、ここから256×256ピクセルを切り出した低解像度の画像を入力データとして用いた。それぞれの状態を数百枚の画像から学習させた。
こうして得られた糞便画像を、深層学習に入力データとして与え学習させることにより、マウスの腸内細菌叢状態を推定できるか検証しました。大腸炎発症・回復マウスと肥満マウス、それぞれ5匹のうち4匹の画像を教師データ[8]として学習させ、学習に用いなかった残り1匹の画像を入力データとして、どのような腸内細菌叢状態にあるかを推定させたところ、おおよそ正しい状態の推定に成功しました(図2)。多少の「間違い」はありましたが、その多くは例えば大腸炎発症・回復マウスにおける「DSS投与後7日目」の状態を「DDS投与後8日目」の状態と間違うといった「近い状態の間違い」でした。従って、この結果は糞便画像から腸内細菌叢の連続的な変化が推定できていることを示しています。
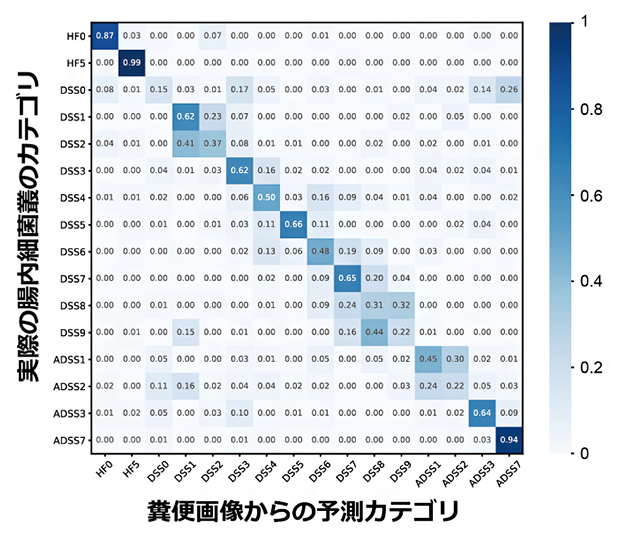
図2 糞便画像による腸内細菌叢のカテゴリの予測の例
HF0、HF5はそれぞれ高脂肪食を摂取前、摂取開始から5週間後のデータ、DSS0~9の数字はDSS投与後の日数、ADSS1~7はDSS投与停止後の日数を表す。数値と色の濃さで予測の正確さを表しており、対角線付近が濃い色をしているのは正確な予測ができていることを示す。例えば、DSS7の糞便画像を正しくDSS7カテゴリと判定した割合は65%であるが、誤判定の多くはカテゴリが1日違うだけのDSS6やDSS8といった「近い状態の間違い」であることが分かる。
さらに、アンプリコンシーケンス解析により同じ糞便サンプルの腸内細菌叢の組成(おおよそ属レベルの分類群に相当するOTU[9]とその存在比)を調べ、その組成を糞便の画像から予測できるかを検証しました。同じように、大腸炎発症・回復マウスと肥満マウス、それぞれ4匹の組成比のデータと糞便画像を正解セットとして学習させ、残り1匹の画像から組成比を予測させたところ、図3に示すように高い精度で予測することに成功しました。
これらの解析から、糞便画像の差異を深層学習によって適切に抽出し、腸内細菌叢の状態を推定できることが示されました。
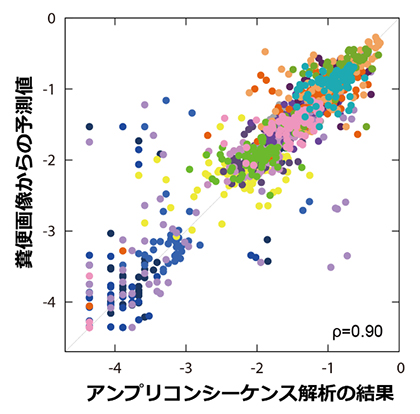
図3 糞便画像からアンプリコンシーケンス解析結果を予測した例
それぞれの点の色は、異なるOTU(おおよそ属レベルの分類群に対応)を表す。グラフのメモリは腸内細菌叢の組成比(10を底とした対数)を表し、数値が1違えば腸内細菌の個体数が10倍違うことを示す。横軸はアンプリコンシーケンス解析による実測値、縦軸は糞便画像からの予測値を表す。各点がほぼ正の相関を示すことから、高い精度で腸内細菌叢の組成比を予測できることが分かった。
今後の期待
本研究では、深層学習の技術を用いることで、糞便の顕微鏡画像から腸内細菌叢の状態を推定する手法を開発しました。広く用いられているアンプリコンシーケンス解析と比較して、今回開発した手法では糞便画像の入力データを低コストで取得できるため、簡便かつ安価に腸内細菌叢の状態を解析できます。
今回開発したこの技術を医療応用することで、例えば腸内細菌叢の乱れが素因となるような大腸がんや大腸炎を、糞便画像から病理検査室で診断できる可能性があります。また、簡単な光学系とデジタルカメラ、そして適切な流路をトイレに設置し、糞便の顕微鏡画像を自動で取得できれば、個人の腸内細菌叢が日々どのように変化するかを追跡し、疾患の発症を予測できる可能性があります。
今回開発した糞便画像と人工知能に基づいた解析は、腸内細菌叢という複雑な生態系を予測し、コントロールする技術に展開できることから、健康維持や医療に大きく貢献することが期待されます。
補足説明
1.腸内細菌叢
ヒトを含む動物の腸内に生息する多種多様な腸内細菌からなる生態系。腸内細菌は、宿主の免疫系の構築や栄養素の供給、消化の補助などの役割を担っている。近年の研究から、腸内細菌叢のバランスが崩れるとさまざまな疾患につながることが報告され、病気の予防や治療の重要なターゲットとして注目されている。
2.深層学習
多層のニューラルネットワークを用いた機械学習手法の一種。データ間の非自明な関係を非線形関数でモデル化し、入出力データの関係を学習することで、分類や予測などのタスクを可能とする。本研究で用いたのは深層学習の一つである畳み込みニューラルネットワーク(CNN)であり、画像の分類に広く用いられている。
3.アンプリコンシーケンス解析
PCR法を用いてDNAのうち特定のゲノム領域を増幅し、次世代シーケンサーで解析する手法。腸内細菌叢研究分野では、腸内細菌叢ゲノムの16S rRNA遺伝子を増幅して解析することで、サンプル中に含まれる細菌の種類と割合を分析する。
4.PCR法
DNAの特定の部分を増幅する手法。最初に、増幅対象のDNA、DNA合成酵素(DNAポリメラーゼ)、大量のプライマーと呼ばれるオリゴヌクレオチドを混合して、反応液を作る。反応液を加熱すると、2本鎖DNAが変性して1本鎖DNAになる。次に急速冷却すると、結合(アニーリング)したプライマーの3’端を起点としてDNAポリメラーゼが働き、1本鎖部分と相補的な2本鎖DNAが合成される。これで2倍量のDNAができたことになる。再び高温にしてDNA変性から繰り返す。このように、PCR法は、DNA鎖長の違いによる変性とアニーリングの違いを利用して、温度の上下を繰り返すだけでDNA合成を繰り返し、DNAを2倍、4倍、8倍、16倍…と増幅する。PCRはPolymerase Chain Reactionの略。
5.次世代シーケンサー
サンガー法を利用した蛍光キャピラリーシーケンサーである「第一世代シーケンサー」と対比させて使われる用語。機種によるが、一度に1,000万~100億個のDNAの配列を決定できる。以前は、ヒトのゲノム配列を決定するのに14年程度かかったが、次世代シーケンサーを用いると数日で決定できる。
6.デキストラン硫酸ナトリウム(DSS)
飲料水に添加することで、腸の上皮細胞を障害する作用を持つ化合物。腸炎モデルマウスの作製に利用される。DDSはDextran Sodium Sulfateの略。
7.グラム染色
細胞壁の構造の違いにより、細菌を染め分ける染色法。細菌を紫と赤の色素で染め、紫に染まるものをグラム陽性、紫に染まらず赤を呈するものをグラム陰性と分類する。大腸菌はグラム陰性である。
8.教師データ
機械学習において、学習に用いられる入力・出力のデータセット。与えられた教師データの入力に対して、対応する出力がなされるようにモデルが調整される。その後に、学習に用いていない入力データに対して適切な出力がなされるかを評価する。今回の研究では、一部のマウスの糞便画像データと対応する腸内細菌叢の状態(例:DSS投与後4日後)のペアを教師データとして用い、それを用いてニューラルネットワークの学習が行われた後に、学習に用いていないマウスの糞便画像データを入力したときに適切な出力がなされるかを評価している。
9.OTU
任意の閾値以上の類似度を持つDNA配列同士をクラスタリングし、便宜上一つの配列として扱うための分類単位。腸内細菌叢研究分野では97%以上の類似度を持つ配列を同じ菌種由来の配列として扱い、おおよそ属レベルの分類群に相当する。OTUはOperational Taxonomic Unitの略。
共同研究チーム
理化学研究所 生命機能科学研究センター 多階層生命動態研究チーム
チームリーダー 古澤 力(ふるさわ ちから)
テクニカルスタッフⅠ 田邊 久美(たなべ くみ)
慶應義塾大学 先端生命科学研究所
特任教授 福田 真嗣(ふくだ しんじ)
研究員 石井 千晴(いしい ちはる)
技術員 加賀田 紀子(かがた のりこ)
教授 冨田 勝(とみた まさる)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究、組織横断連携プロジェクト「ライフサイエンスの横断的取組による超高齢社会課題解決への貢献」)、科学技術振興機構(JST)戦略的創造研究推進事業総括実施型研究(ERATO)「深津共生進化機構プロジェクト(研究総括:深津武馬)」などによる支援を受けて行われました。
原論文情報
Chikara Furusawa, Kumi Tanabe, Chiharu Ishii, Noriko Kagata, Masaru Tomita, Shinji Fukuda, “Decoding gut microbiota by imaging analysis of fecal samples”, iScience, 10.1016/j.isci.2021.103481
発表者
理化学研究所
生命機能科学研究センター 多階層生命動態研究チーム
チームリーダー 古澤 力(ふるさわ ちから)
慶應義塾大学 先端生命科学研究所
特任教授 福田 真嗣(ふくだ しんじ)
報道担当
理化学研究所 広報室 報道担当
慶應義塾大学先端生命科学研究所 渉外担当 狩野、塩澤