新たな定量的アプローチにより、科学者や技術者がDNA編集プロセスをよりコントロールしやすくなった A new quantitative approach gives scientists and engineers more control over the DNA editing process
2022-07-20 ミネソタ大学
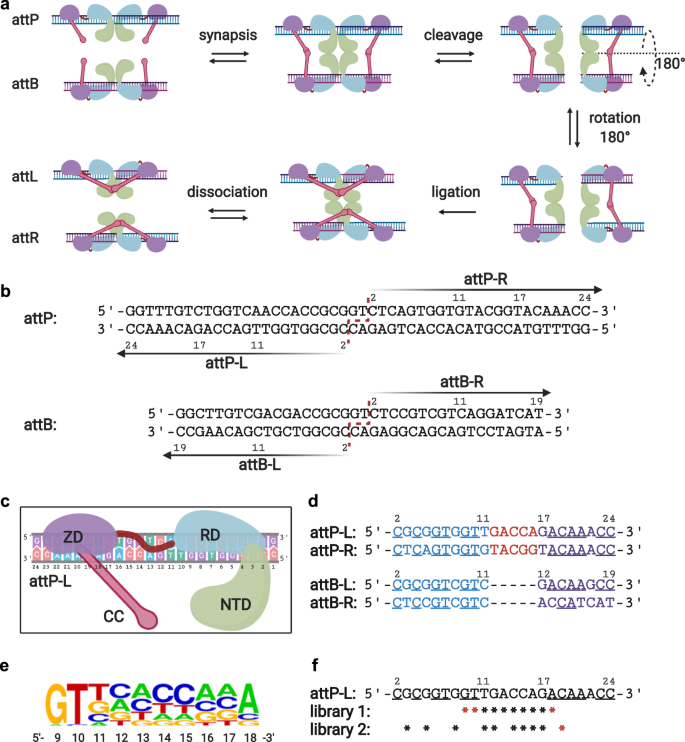
ミネソタ大学のエンジニアは、ハイスループット実験と機械学習モデルを組み合わせて、部位特異的組み換えプロセスをより効率的かつ予測可能にする方法を開発した。このモデルでは、研究者がDNAを編集する速度をプログラムすることができる。つまり、治療用細胞が環境に反応する速度を制御することで、薬物や治療用タンパク質を生成する速度と速度を制御することができるのだ。
チームはまず、部位特異的な組み換えの速度を計算する実験手法を開発し、その情報を使って機械学習アルゴリズムを訓練した。最終的には、DNA配列を入力するだけで、そのDNA配列がどの程度の割合で組み換わるかを予測するモデルを構築した。
さらに、細胞内で複数のタンパク質が同時に生成されることを予測し、制御するためのモデリングも可能であることがわかった。
<関連情報>
- https://cse.umn.edu/college/news/engineers-develop-new-tool-will-allow-more-personalized-cell-therapies
- https://www.nature.com/articles/s41467-022-31538-3
部位特異的な組換え率を予測可能なDNA配列のモデルガイドによる工学的研究 Model-guided engineering of DNA sequences with predictable site-specific recombination rates
Qiuge Zhang,Samira M. Azarin & Casim A. Sarkar
Nature Communications Published:20 July 2022
DOI:https://doi.org/10.1038/s41467-022-31538-3
Abstract
Site-specific recombination (SSR) is an important tool in synthetic biology, but its applications are limited by the inability to predictably tune SSR reaction rates. Facile rate manipulation could be achieved by modifying the DNA substrate sequence; however, this approach lacks rational design principles. Here, we develop an integrated experimental and computational method to engineer the DNA attachment sequence attP for predictably modulating the inversion reaction mediated by the recombinase Bxb1. After developing a qPCR method to measure SSR reaction rate, we design, select, and sequence attP libraries to inform a machine-learning model that computes Bxb1 inversion rate as a function of attP sequence. We use this model to predict reaction rates of attP variants in vitro and demonstrate their utility in gene circuit design in Escherichia coli. Our high-throughput, model-guided approach for rationally tuning SSR reaction rates enhances our understanding of recombinase function and expands the synthetic biology toolbox.