あまり理解されていない現象への重要な洞察を得た Study uncovers key insights into little-understood phenomenon
2022-08-23 カリフォルニア大学校アーバイン校(UCI)
トランスポゾンの比率はゲノムによって大きく異なる。ヒトでは50%、サンショウウオでは65%、フグではわずか6%という具合である。また、異なる種類のショウジョウバエの間でも、2%から25%までの幅がある。
「今回の新たな調査で、これらの副作用の強さや有害性に違いがあることがわかりました。我々は、時間とともに、副作用が隣接する遺伝子に特に有害である種は、トランスポゾンを除去する強い選択を経験したことを知りました。その結果、現在では彼らのゲノムに含まれるトランスポゾンの割合が低くなっています。」とリーは言う。
研究チームはまた、副作用の深刻さのばらつきが、化学修飾を製造・分配する遺伝子に由来している可能性を発見しました。生物学者らは、今後の研究でこの問題をさらに詳しく調べる予定である。
トランスポゾンは、すでにいくつかの稀な遺伝性疾患と関連している。さらに最近では、老化した脳やある種の癌細胞で活性化することが分かっている。
「これらの点におけるトランスポゾンの役割はまだ不明ですが、いずれは、これらの化学変化を生み出す遺伝子を変化させることによって、治療法を開発することが可能になるかもしれません。我々はまた、細胞が化学修飾を分配する方法に影響を与えることが知られている、食事や環境といった考慮事項が、トランスポゾンに影響を与えるかどうかを調べたいと思います。」とリーは述べています。
<関連情報>
- https://news.uci.edu/2022/08/23/uci-biologists-track-dna-parasites-in-the-hunt-for-disease-treatments/
- https://elifesciences.org/articles/81567
種特異的なクロマチンランドスケープがトランスポーザブルエレメントのゲノム進化を形成する方法を決定する Species-specific chromatin landscape determines how transposable elements shape genome evolution
Yuheng Huang,Harsh Shukla,Yuh Chwen G Lee
eLife Published:Aug 23, 2022
DOI:https://doi.org/10.7554/eLife.81567
Abstract
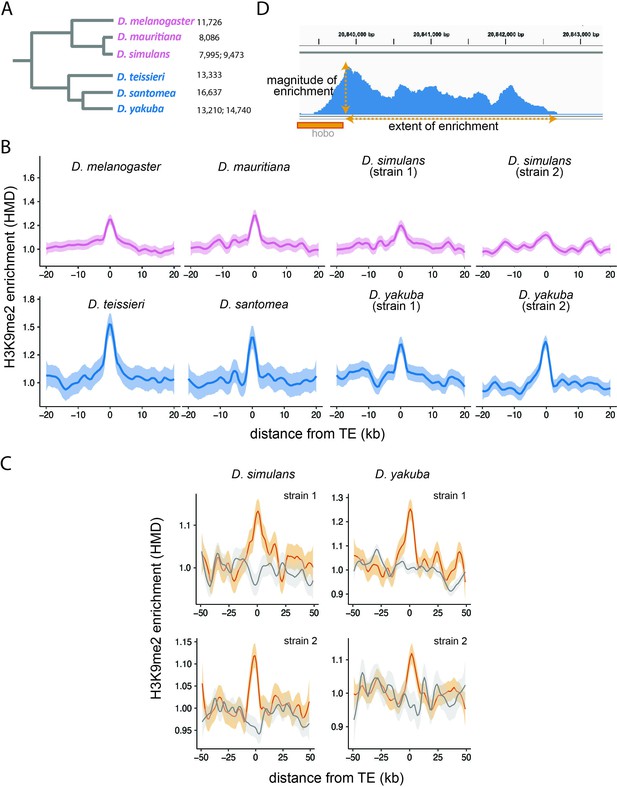
Transposable elements (TEs) are selfish genetic parasites that increase their copy number at the expense of host fitness. The ‘success’, or genome-wide abundance, of TEs differs widely between species. Deciphering the causes for this large variety in TE abundance has remained a central question in evolutionary genomics. We previously proposed that species-specific TE abundance could be driven by the inadvertent consequences of host-direct epigenetic silencing of TEs—the spreading of repressive epigenetic marks from silenced TEs into adjacent sequences. Here, we compared this TE-mediated local enrichment of repressive marks, or ‘the epigenetic effect of TEs’, in six species in the Drosophila melanogaster subgroup to dissect step-by-step the role of such effect in determining genomic TE abundance. We found that TE-mediated local enrichment of repressive marks is prevalent and substantially varies across and even within species. While this TE-mediated effect alters the epigenetic states of adjacent genes, we surprisingly discovered that the transcription of neighboring genes could reciprocally impact this spreading. Importantly, our multi-species analysis provides the power and appropriate phylogenetic resolution to connect species-specific host chromatin regulation, TE-mediated epigenetic effects, the strength of natural selection against TEs, and genomic TE abundance unique to individual species. Our findings point toward the importance of host chromatin landscapes in shaping genome evolution through the epigenetic effects of a selfish genetic parasite.