2020-01-22 理化学研究所,沖縄科学技術大学院大学
理化学研究所(理研)生命医科学研究センター疾患遺伝研究チームの山本雅チームリーダー(沖縄科学技術大学院大学細胞シグナルユニット教授)、鈴木亨上級研究員らの共同研究グループは、メッセンジャーRNA(mRNA)[1]を分解する酵素(タンパク質複合体[2])に存在する複数の活性因子の中には、mRNA分解やマウス・細胞の生存に必須なものと代替可能なものがあることを発見しました。
本研究成果は、さまざまな組織・細胞で不可欠な役割を果たしているmRNA分解の分子機構を解明するものであり、今後、生命現象や疾患のメカニズムのさらなる理解に貢献すると期待できます。
不要なタンパク質が産生されたり、必要なタンパク質であっても過剰に産生されたりすると、生体の恒常性維持に支障を来し、疾患の要因となります。それを防ぐ分子機構の一つが、mRNAの分解です。
今回、共同研究グループは、mRNA分解酵素の「CCR4-NOT複合体」に存在する四つの酵素活性因子(CNOT6、CNOT6L、CNOT7、CNOT8)を欠損させたマウス胎児の繊維芽細胞[3]を用いて、それぞれの因子の機能を調べました。その結果、CNOT7とCNOT8は、マウス・細胞の生存、mRNA分解に必須であることが分かりました。一方、CNOT6とCNOT6Lの両方を欠損しても、マウス・細胞の生存、mRNA分解は大きく損なわれなかったことから、CNOT6とCNOT6Lは他の因子に代替可能であると考えられます。
本研究は、英国の科学雑誌『RNA biology』のオンライン版(1月10日付)に掲載されました。
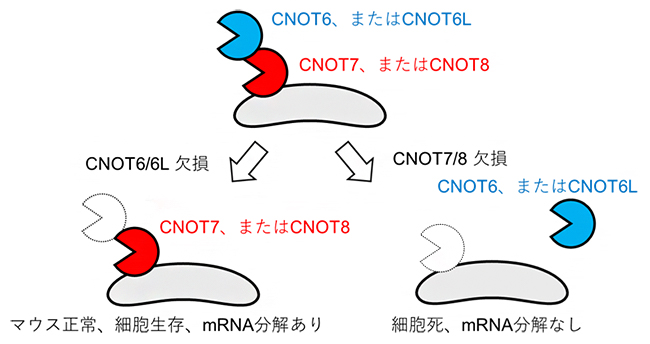
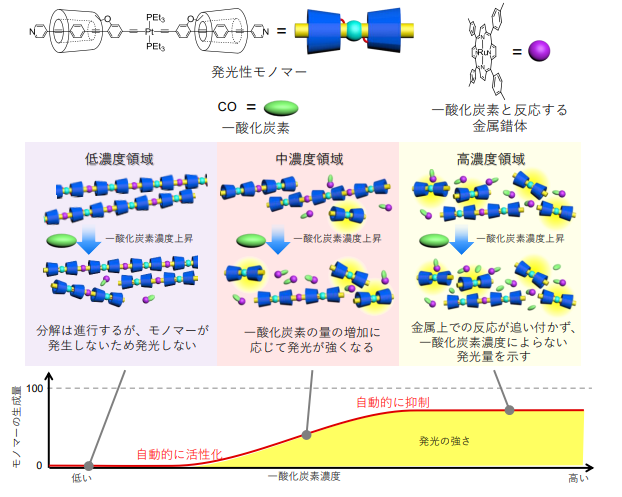
図 CCR4-NOT複合体に存在する酵素活性因子の異なる役割
背景
不要なタンパク質が産生されたり、必要なタンパク質であっても過剰に産生されたりすると、生体の恒常性維持に支障を来し、疾患の要因となります。それを防ぐ分子機構の一つとして、メッセンジャーRNA(mRNA)の分解が知られています。mRNA分解は、mRNAのポリA鎖[4]を削る反応で始まります。この反応の活性を持つタンパク質複合体「CCR4-NOT」は、哺乳動物のほぼ全ての細胞種に存在することから、主要なmRNA分解酵素であると考えられています。
CCR4-NOT複合体には、酵素活性を持つ四つの因子(CNOT6、CNOT6L、CNOT7、CNOT8)が存在します。CNOT6とCNOT6L、CNOT7とCNOT8は類似した構造と役割を持ち、それぞれどちらか一方だけが複合体に存在しています。そして、CNOT6またはCNOT6Lは、CNOT7またはCNOT8との結合を介して複合体に存在することが分かっています(図1)。しかし生体において、四つの酵素活性因子がそれぞれどのような役割を果たすのかについてはよく分かっていませんでした。
図1 CCR4-NOT複合体に存在する四つの酵素活性因子
CCR4-NOT複合体には、CNOT6、CNOT6L、CNOT7、CNOT8の四つの酵素活性因子が存在する。CNOT6とCNOT6L、CNOT7とCNOT8のそれぞれどちらか一方だけが複合体に存在し、かつCNOT6またはCNOT6Lは、CNOT7またはCNOT8との結合を介して複合体に存在する。
研究手法と成果
共同研究グループは、マウス繊維芽細胞でCNOT6、CNOT6L、CNOT7、CNOT8を欠損させるとどのような影響があるか調べました。各酵素活性因子を欠損した細胞は、因子の遺伝子を改変したマウス胎児から調製しました。
CNOT6、CNOT6L、CNOT7、CNOT8をそれぞれ単独で欠損した細胞は、その生存への影響はほとんど見られませんでした(図2)。CNOT6とCNOT6Lの両方を欠損した細胞も同様の結果でした(図2上)。また、CNOT6とCNOT6Lを欠損したマウスは正常に生まれ、成体まで成長しました。しかし、CNOT7とCNOT8を欠損した細胞は、徐々に死んでいくことが分かりました(図2下)。
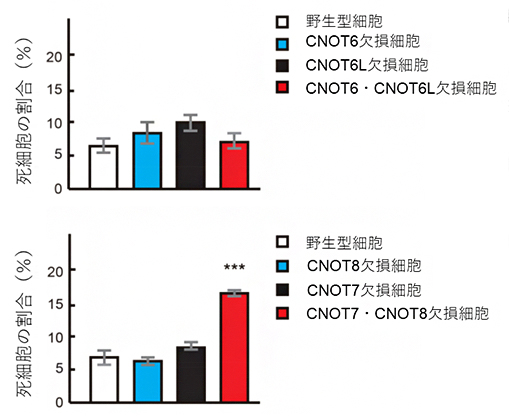
図2 各酵素活性因子の欠損細胞で誘導される細胞死
細胞をヨウ化プロピジウムで染色して、死亡している細胞の割合を定量した。CNOT6欠損細胞、CNOT6L欠損細胞、CNOT7欠損細胞、CNOT8欠損細胞、CNOT6・CNOT6L欠損細胞の死細胞の割合は、野生型細胞とほとんど変わらないが、CNOT7・CNOT8欠損細胞の死細胞の割合は高くなった。
次に、四つの酵素活性因子がCCR4-NOT複合体のmRNA分解機能にどのような影響を与えるのか調べました。その結果、CNOT7とCNOT8の両方を欠損した細胞ではポリA鎖を削る活性が大きく失われたのに対し、CNOT6とCNOT6Lの両方を欠損した細胞では、野生型細胞と同程度の活性が残っていることが分かりました(図3)。
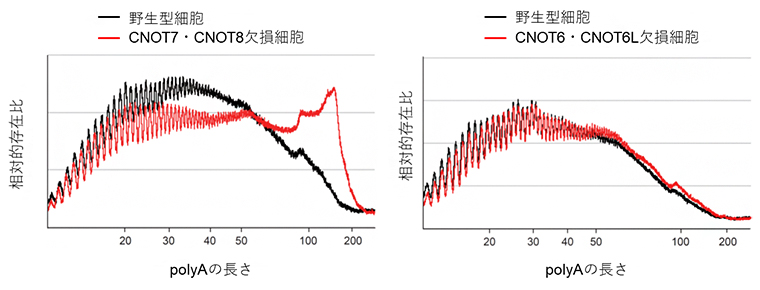
図3 CNOT7とCNOT 8の欠損細胞におけるポリA鎖分解の異常
細胞から抽出したtotal RNAを放射性同位元素で標識し、ポリA部位以外を分解する処理をした。反応産物を電気泳動し、ポリAの長さごとに放射性標識の強度を定量した。2検体ごとの平均を算出し、全体の強度に対する比の値をグラフに示した。左側のCNOT7・CNOT8欠損細胞では、長いポリA鎖の存在比が野生型よりも高いのに対し、右側のCNOT6・CNOT6L欠損細胞では、存在比は野生型と変わらなかった。
また、CNOT7とCNOT8の両方を欠損した細胞に、CNOT7を再導入すると細胞死は起こらなくなりました。しかし、ポリA鎖を削る活性を持たない変異を持つCNOT7を導入し、CNOT6とCNOT6Lが存在できる状況を作っても、細胞死を止めることはできませんでした。
さらに、RNA-seq[5]の手法を用いてmRNAの発現量と半減期[6]を網羅的に調べたところ、CNOT7とCNOT8の両方を欠損した細胞では、非常に多くの種類のmRNAの発現量が野生型よりも増加し、それぞれのmRNAの半減期も野生型よりも延びていることが分かりました。一方、CNOT6とCNOT6Lの両方を欠損した細胞におけるmRNAの発現量や半減期の変化は、CNOT7とCNOT8の両方を欠損した細胞ほど大きくありませんでした。
以上の結果は、ポリA鎖を削ってmRNAを分解する機能の有無が、マウス繊維芽細胞の生死を左右する可能性を示しています。
今後の期待
本研究の結果から、CCR4-NOT複合体に存在する酵素活性因子のうちCNOT7とCNOT8は、mRNA分解に必須な役割を果たしていること、CNOT6とCNOT6Lは代替可能であることが分かりました。mRNA分解機能が果たせない影響は、細胞死という現象に反映されていました。
今後、各酵素活性因子を欠損した細胞を調製するために作製した遺伝子改変マウスを利用し、より詳しく調べれば、さらに高次な機能における活性因子の機能解析が可能になります。それにより、組織の特異的な機能や外部環境の変化に対する応答などにおいて、CCR4-NOT複合体のつかさどるmRNA分解の果たす役割が解明されると期待できます。
補足説明
1.メッセンジャーRNA(mRNA)
伝令RNAとも呼ばれる。DNA上のアデニン、グアニン、シトシン、チミンからなる塩基配列に基づいて合成される一本鎖のヌクレオチド。mRNAを使ってタンパク質が合成される。
2.タンパク質複合体
複数の異なるタンパク質が結合した状態のもの。各タンパク質は異なる役割を担っているが、結合した状態で一つの機能(CCR4-NOT複合体の場合はmRNA分解)を果たす。
3.マウス胎児繊維芽細胞
繊維芽細胞は結合組織を構成する最も主要な細胞で、膠原線維や弾力線維を産生する。紡錘形を呈する。マウス胎児から調製したものを特にマウス胎児繊維芽細胞という。
4.ポリA鎖
mRNAやその他いくつかのRNAの3’末端に付加されるAMP(アデノシン一リン酸)の重合体。
5.RNA-seq
次世代シークエンシングの技術を利用して転写物の発現を解析する手法。RNA sequencingの略。
6.半減期
特定の時間(測定開始時間)から、量が半分になるまでの時間。
共同研究グループ
理化学研究所
生命医科学研究センター 疾患遺伝研究チーム
チームリーダー 山本 雅(やまもと ただし)
(沖縄科学技術大学院大学 細胞シグナルユニット 教授)
上級研究員 鈴木 亨(すずき とうる)
生命機能科学研究センター 生体ゲノム工学研究チーム(研究当時)
チームリーダー 古田 泰秀(ふるた やすひで)
研究員 阿部 高也(あべ たかや)
沖縄科学技術大学院大学 細胞シグナルユニット
研究員 栁谷 朗子(やなぎや あきこ)
研究員 高橋 明格(たかはし あきのり)
研究員 呉羽 拓(くれは たく)
大学院生 ディナ・ムスタファ(Dina Mostafa)
東京慈恵会医科大学 総合医科学研究センター 基盤研究施設
准教授 鐘ケ江 裕美(かねがえ ゆみ)
秋田大学大学院 医学系研究科 分子機能学・代謝機能学講座
教授 久場 敬二(くば けいじ)
助教 山口 智和(やまぐち ともかず)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金基盤研究(S)「CCR4-NOTデアデニレース欠損に伴う病態解析と新たな遺伝子発現制御機構(研究代表者:山本雅)」、同基盤研究(C)「細胞間相互作用を介した肝再生機構の解明と再生肝細胞の起源の同定(研究代表者:鈴木亨)」「Poly(A) 鎖の長さによる遺伝子発現制御の破綻が導く致死性肝炎・脂肪委縮症(研究代表者:高橋明格)」「膵β細胞でのグルコース応答性インスリン分泌における転写後調節の分子機構の解析(研究代表者:栁谷朗子)」による支援を受けて行われました。
原論文情報
Dina Mostafa, Akinori Takahashi, Akiko Yanagiya, Tomokazu Yamaguchi, Takaya Abe, Taku Kureha, Keiji Kuba, Yumi Kanegae, Yasuhide Furuta, Tadashi Yamamoto and Toru Suzuki, “Essential functions of the CNOT7/8 catalytic subunits of the CCR4-NOT complex in mRNA regulation and cell viability”, RNA Biology, 10.1080/15476286.2019.1709747
発表者
理化学研究所
生命医科学研究センター 疾患遺伝研究チーム
チームリーダー 山本 雅(やまもと ただし)
(沖縄科学技術大学院大学 細胞シグナルユニット 教授)
上級研究員 鈴木 亨(すずき とうる)
報道担当
理化学研究所 広報室 報道担当
沖縄科学技術大学院大学 広報メディアセクション