ライス大学の研究者が、細胞に「非正規の」経路を植え付け、自ら薬を作るように仕向ける。 Rice University scientists imbue cells with ‘noncanonical’ pathway to make own drugs
2022-09-16 ライス大学
この種の鳥は、非正規アミノ酸(つまり、ほとんどのタンパク質をコード化するのに必要な20種類のアミノ酸の中にないアミノ酸)を生成する酵素を自然に作り出すことが知られている唯一の鳥である。
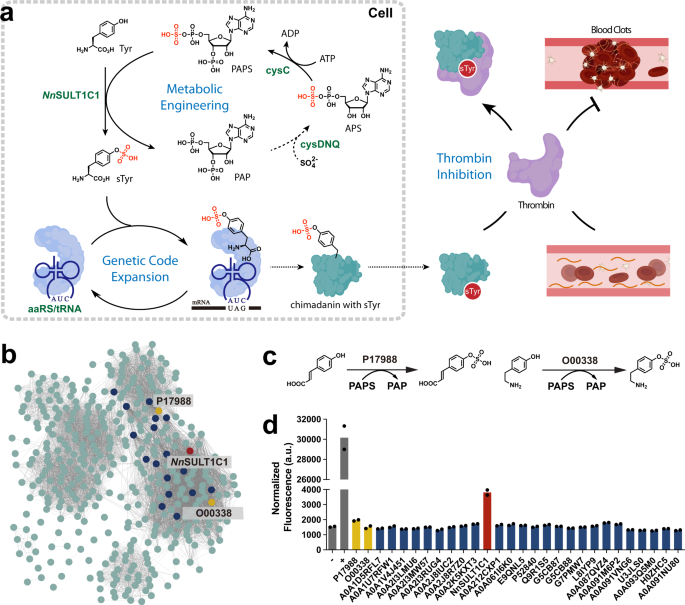
新しい研究によれば、標準アミノ酸であるチロシンの変異体であるスルホチロシン(sTyr)というアミノ酸が、治療用タンパク質を発現する生きた細胞をプログラムするための重要な構成要素になることが明らかになった。これにより、細胞が環境をモニターし、必要な治療を行うセンサーとして機能する可能性がある。
トキがsTyrを合成してタンパク質に組み込む能力を模倣するには、細胞のDNAに変異コドンを加え、トキが持つ転移酵素である硫酸転移酵素1C1を作らせる必要がある。
この概念実証研究により、初めてsTyrを合成する哺乳類細胞が作製された。
血液凝固を防ぐための抗凝固剤であるトロンビン阻害剤の効能を増強する細胞も作製し、実験を行った。
<関連情報>
- https://news.rice.edu/news/2022/birds-enzyme-points-toward-novel-therapies
- https://www.nature.com/articles/s41467-022-33111-4
非正規アミノ酸生合成の可能性を引き出し、チロシン硫酸化を精密に行う細胞を創製する Unleashing the potential of noncanonical amino acid biosynthesis to create cells with precision tyrosine sulfation
Yuda Chen,Shikai Jin,Mengxi Zhang,Yu Hu,Kuan-Lin Wu,Anna Chung,Shichao Wang,Zeru Tian,Yixian Wang,Peter G. Wolynes & Han Xiao
Nature Communications Published:16 September 2022
DO:Ihttps://doi.org/10.1038/s41467-022-33111-4
Abstract
Despite the great promise of genetic code expansion technology to modulate structures and functions of proteins, external addition of ncAAs is required in most cases and it often limits the utility of genetic code expansion technology, especially to noncanonical amino acids (ncAAs) with poor membrane internalization. Here, we report the creation of autonomous cells, both prokaryotic and eukaryotic, with the ability to biosynthesize and genetically encode sulfotyrosine (sTyr), an important protein post-translational modification with low membrane permeability. These engineered cells can produce site-specifically sulfated proteins at a higher yield than cells fed exogenously with the highest level of sTyr reported in the literature. We use these autonomous cells to prepare highly potent thrombin inhibitors with site-specific sulfation. By enhancing ncAA incorporation efficiency, this added ability of cells to biosynthesize ncAAs and genetically incorporate them into proteins greatly extends the utility of genetic code expansion methods.