2022-10-27 理化学研究所,東京大学,京都大学,東京医科歯科大学
理化学研究所(理研)開拓研究本部 渡邉分子生理学研究室の渡邉 力也 主任研究員、安藤 潤 研究員、飯田 龍也 テクニカルスタッフⅠ、東京大学 先端科学技術研究センターの西増 弘志 教授、京都大学 医生物学研究所の野田 岳志 教授、東京医科歯科大学の武内 寛明 准教授らの共同研究グループは、新型コロナウイルス(SARS-CoV-2)由来のウイルスRNAを「1分子」レベルで識別し、世界最速で検出できる安価な小型装置を開発することに成功しました。
本研究成果は、臨床現場即時検査(POCT)に対応した、新型コロナウイルス感染症(COVID-19)を含む次世代の感染症診断法の核心技術として実用化されるものと期待できます。
今回、共同研究グループは、2022年度に開発した世界最速のSARS-CoV-2全自動検出装置「opn-SATORI装置」の検出部を大幅に改善した安価な小型装置「COWFISH」を開発しました。COWFISHは、従来のopn-SATORI装置と比較して、設置面積比で約5分の1以下まで小型化、構成部品の総額を120万円程度と約30分の1以下まで低コスト化することに成功しました。また、テレセントリックレンズ[1]と市販の一眼レフカメラを用いることで、大面積の視野を高精細に撮像することが可能となるとともに、最短3分の迅速検出を実現しました。一方、SARS-CoV-2の検出感度は、opn-SATORI装置の検出部である共焦点顕微鏡[2]とほぼ同等であり、また、臨床検体を用いた実証実験では、陽性判定において95%の正解率を達成しました。
本研究は、科学雑誌『Lab on a Chip』オンライン版(10月18日付)に掲載されました。

COWFISHの外観
背景
現在、新型コロナウイルス感染症(COVID-19)の感染診断では、主にウイルスのタンパク質抗原を検出する方法(抗原検査法)とウイルスRNAを増幅して検出する方法(PCR検査法[3])が利用されており、それぞれスクリーニング、確定診断など用途に応じて使い分けられています。抗原検査法は30分程度で迅速かつ簡便にウイルスを検出できるため、スクリーニングには適していますが、検出感度や特異度[4]の低さに起因する検出エラーの多さが問題になっています。一方、PCR検査法は感度が優れ、確定診断に適していますが、検出の前処理に最短で1時間程度かかるため、大量の検体を迅速に解析し、診断につなげることが困難です。そのため、PCR検査法の「感度の高さ」と、抗原検査法の「迅速・簡便さ」を両立させた新しいウイルス検出法の開発が期待されています。
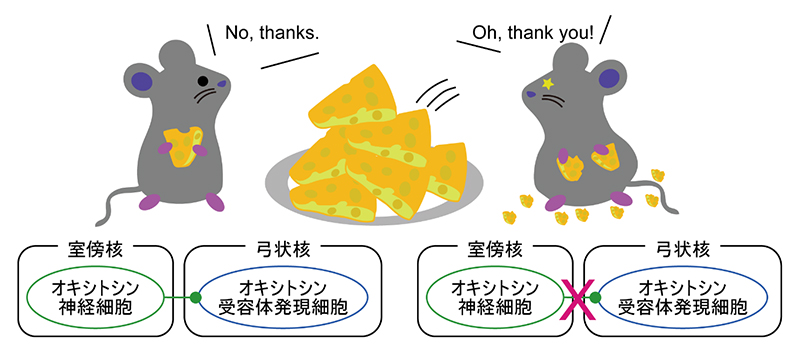
この背景も踏まえ、2022年度、共同研究グループは世界最速の新型コロナウイルス(SARS-CoV-2)の全自動検出装置「opn-SATORI装置」を開発しました注1)。opn-SATORI装置は、CRISPR-Cas13a[5]と呼ばれる酵素と微小試験管を集積したマイクロチップを用いることで、SARS-CoV-2由来のウイルスRNAを「1分子」レベルで識別し、9分以内の世界最速の迅速検出を可能とする全自動装置です(図1)。検出感度は1.4コピー/マイクロリットル(μL、1μLは100万分の1リットル、PCR検査法とほぼ同等)で、臨床検体を用いた検証実験では、陽性判定および変異株判定において98%以上の正解率を達成しており、従来技術と検出原理の異なる革新的な装置でした。
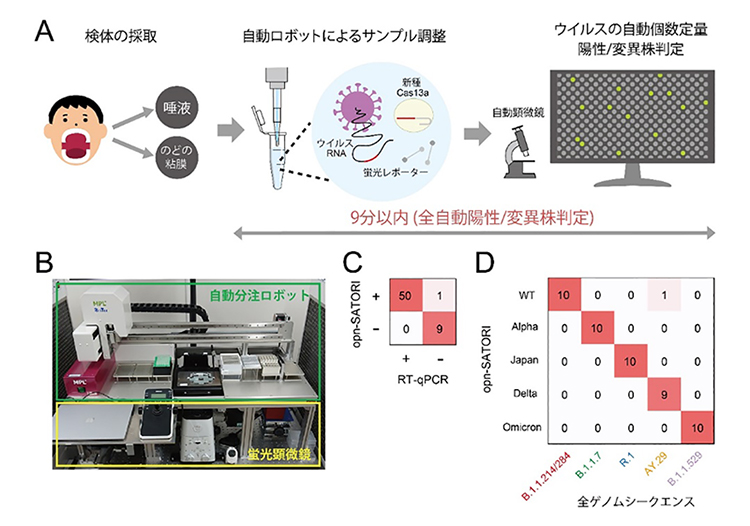
図1 独自の新型コロナウイルスの世界最速検出装置(opn-SATORI装置)
(A)感染診断の模式図。サンプル調製、顕微鏡観察、ウイルスの個数定量、陽性/変異株判定の全ての工程が自動化され、9分以内で完結できる。
(B)opn-SATORI装置写真。
(C)(D)臨床検体を用いた陽性判定(C)と変異株判定(D)の結果。ともに正解率98%以上。
しかし、opn-SATORI装置は横幅120cm、奥行70cmと大きく、検出部が共焦点顕微鏡で構成されるため非常に高価であるなどの課題を抱えていました。社会実装を想定すると、大規模な検査センターや大病院には設置できる可能性はあるものの、迅速検出の利点を最大限に活用できる市中のクリニックや検疫所に設置することは難しく、小型化および低コスト化した新装置の開発が期待されていました。
注1)2022年5月26日プレスリリース「新型コロナウイルスの超高感度・全自動迅速検出装置の開発」
研究手法と成果
今回、共同研究グループでは、opn-SATORI装置の検出部を小型化・低コスト化し、その装置を「Compact Wide-field Femtoliter-chamber Imaging System for High-speed digital bioanalysis:COWFISH」と名付けました(図2A)。COWFISHは、従来のopn-SATORI装置の検出部を一から再設計するとともに、COVID-19診断検査の律速過程の一つであった「蛍光画像の撮像時間」の大幅な短縮に成功しました。詳しい改善点は以下の通りです。
1)opn-SATORI装置と比較して、設置面積比で約5分の1以下まで小型化
(横幅35cm、奥行45cm、高さ30cmの卓上サイズ)
2)市販品の組み合わせにより、検出部の原価を約30分の1以下まで低コスト化
(共焦点顕微鏡が不要となり、構成部品の総額が120万円以下になった)
3)一眼レフカメラとテレセントリックレンズを利用することで、大面積の視野を高解像度で撮像することが可能
(視野面積:11.8mm×7.9mm[約60万個の微小試験管を1回で撮像可能]、空間分解能:3.4マイクロメートル(μm、1μmは100万分の1メートル)[緑色]、5.6μm[赤色])注2)
4)マイクロチップの蛍光画像の撮像時間を約10分の1まで短縮
(1回の撮像時間が約10秒)
COWFISHはopn-SATORI装置と同様に、ウイルスRNAを1個ずつ識別し、検体中の個数を定量できます(図2B)。検出感度は共焦点顕微鏡を検出部として用いた場合と同等であり、PCR検査法でCt[6]30相当の検体であれば、観察視野の大面積化により、従来装置よりも速く、検出の全工程を含めてわずか3分程度で検出できます。また、COVID-19臨床検体を用いた検証実験では、陽性判定において95%の正解率を達成しました(図2C、D)。
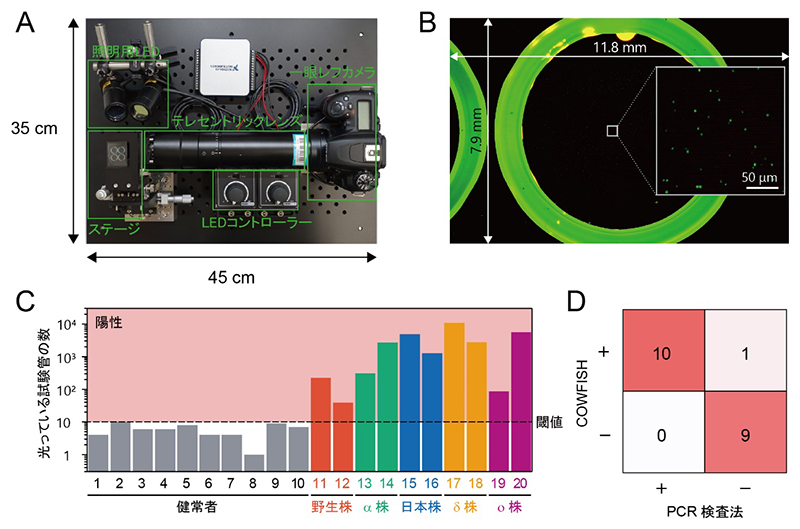
図2 COWFISHによるCOVID-19感染診断
(A)COWFISHの内部写真。35cm×45cmの中に全ての要素(照明用LED、コントローラー、テレセントリックレンズ、一眼レフカメラなど)が収められている。
(B)1回に撮像される蛍光画像。視野の大きさは11.8mm×7.9mm。空間分解能は緑色:3.4μm、赤色:5.6μm。約60万個の微小試験管の蛍光画像を1回で撮影できる。緑色に光っている試験管は、そこにSARS-CoV-2由来のウイルスRNAが内包されていることを示している。
(C)臨床検体を用いたときの光っている試験管の数。
(D)臨床検体を用いた陽性判定(正解率95%)。
注2)opn-SATORI装置では検出部が共焦点顕微鏡であるため、60万個の微小試験管の全体を撮像するためには、20倍の対物レンズを用いて64視野に分割して撮像し、それらをタイリングする必要があった。
今後の期待
COWFISHは、ウイルスRNAを1分子レベルで識別して世界最高速度で自動定量し、さらには、陽性判定へとつなげることができる小型かつ安価なウイルス感染症診断装置です。60万個の微小試験管を一度に撮像できるため、検出時間の大幅な短縮が実現し、安価で素早く多種のウイルス感染症を正確に診断できる次世代の感染症診断装置となることが期待できます。
また、従来のopn-SATORI装置から小型化・低コスト化することにより、COWFISHは大規模検査センターだけでなく、迅速検出のニーズの高い市中のクリニックや検疫所に設置され、臨床現場即時検査(POCT)を実施するための基盤装置となることも併せて期待できます(図3)。
図3 感染症診断におけるCOWFISHの将来展望
COWFISHはSARS-Cov-2だけでなく、多種の感染症を迅速に診断できる装置であり、臨床現場即時検査(POCT)を実現する。
補足説明
1.テレセントリックレンズ
主光線がレンズ光軸に対して平行になるように設計されたレンズ。主に、産業用途のマシンビジョンシステムの一部として用いられる。
2.共焦点顕微鏡
焦点面のみからの蛍光画像を取得できる顕微鏡。主に、高解像度の蛍光画像を取得するために用いられる。
3.PCR検査法
ポリメラーゼ連鎖反応法(PCR法)によってウイルスに特徴的な塩基配列を増幅してウイルスの有無を検出する方法。最初に、増幅対象のDNA、DNA合成酵素(DNAポリメラーゼ)、大量のプライマーと呼ばれるオリゴヌクレオチドを混合して、反応液を作る。反応液を加熱すると、2本鎖DNAが変性して1本鎖DNAになる。次に急速冷却すると、結合(アニーリング)したプライマーの3端を起点としてDNAポリメラーゼが働き、1本鎖部分と相補的な2本鎖DNAが合成される。これで2倍量のDNAができたことになる。再び高温にしてDNA変性から繰り返す。このように、PCR法は、DNA鎖長の違いによる変性とアニーリングの違いを利用して、温度の上下を繰り返すだけでDNA合成を繰り返し、DNAを2倍、4倍、8倍、16倍……と増幅する。PCRはpolymerase chain reactionの略。SARS-CoV-2のようなRNAウイルスを検出する場合は、ウイルスRNAの塩基配列をDNAに写し取ってからPCR法で増幅する。
4.特異度
検査の性能を表す指標の一つ。陰性のものを正しく陰性と判定した割合。
5.CRISPR-Cas13a
多くの細菌は、「CRISPR-Casシステム」と呼ばれる獲得免疫システムを備えている。VI型CRISPR-Casシステムに関与するCRISPR-Cas13aは、ガイドRNAと複合体を形成し、ガイドRNAと相補的な1本鎖RNAと結合すると活性化し、1本鎖RNAを切断するRNA依存性RNA切断酵素である。
6.Ct
PCR検査法で陽性と判断したときの増幅サイクル数。CtはThreshold cycleの略。
共同研究グループ
理化学研究所
開拓研究本部 渡邉分子生理学研究室
主任研究員 渡邉 力也(ワタナベ・リキヤ)
研究員 篠田 肇(シノダ・ハジメ)
研究員 安藤 潤(アンドウ・ジュン)
テクニカルスタッフⅠ 飯田 龍也(イイダ・タツヤ)
テクニカルスタッフⅠ 牧野 麻美(マキノ・アサミ)
テクニカルスタッフⅠ 吉村 麻実(ヨシムラ・マミ)
東京大学 先端科学技術研究センター
教授 西増 弘志(ニシマス・ヒロシ)
京都大学 医生物学研究所
教授 野田 岳志(ノダ・タケシ)
東京医科歯科大学大学院 医歯学総合研究科
准教授 武内 寛明(タケウチ・ヒロアキ)
研究支援
本研究は、科学技術振興機構(JST)戦略的創造研究推進事業CREST「細胞外微粒子に起因する生命現象の解明とその制御に向けた基盤技術の創出(研究総括:馬場嘉信、JPMJCR19H5)」、三菱財団自然科学研究特別助成「新型コロナウイルス等感染症に関する学術研究助成」、日本学術振興会(JSPS)科学研究費助成事業学術変革領域研究(A)「未開拓タンパク質の1分子計測技術・デバイス開発(研究代表者:渡邉力也、20H05931)」、同基盤研究(A)「デジタル検出技術を基盤としたウイルスの新規感染診断法の開発(研究代表者:渡邉力也、21H04645)」、日本医療研究開発機構(AMED)「ウイルス等感染症対策技術開発事業(20he0622032h0001)」「新興・再興感染症に対する革新的医薬品等開発推進研究事業(20fk0108529h0001)」、新エネルギー・産業技術総合開発機構(NEDO)「官民による若手研究者発掘支援事業(JP22he0422018)」をはじめとする諸機関からの支援によって行われました。
原論文情報
Tatsuya Iida, Jun Ando, Hajime Shinoda, Asami Makino, Mami Yoshimura, Kazue Murai, Makiko Mori, Hiroaki Takeuchi, Takeshi Noda, Hiroshi Nishimasu and Rikiya Watanabe, “Compact wide-field femtoliter-chamber imaging system for high-speed and accurate digital bioanalysis”, Lab on a Chip, 10.1039/d2lc00741j
発表者
理化学研究所
開拓研究本部 渡邉分子生理学研究室
主任研究 員渡邉 力也(ワタナベ・リキヤ)
研究員 安藤 潤(アンドウ・ジュン)
テクニカルスタッフⅠ 飯田 龍也(イイダ・タツヤ)
東京大学 先端科学技術研究センター
教授 西増 弘志(ニシマス・ヒロシ)
京都大学 医生物学研究所
教授 野田 岳志(ノダ・タケシ)
東京医科歯科大学大学院 医歯学総合研究科
准教授 武内 寛明(タケウチ・ヒロアキ)
報道担当
理化学研究所 広報室報道担当
東京大学 先端科学技術研究センター 広報・情報室
京都大学 総務部 広報課 国際広報室
東京医科歯科大学 総務部 総務秘書課 広報係