タンパク質の塩基配列の変化を伴わない遺伝子の変化でも、タンパク質の機能が変化することをモデルで示す Modeling shows how genetic changes that don’t lead to changes in protein sequence can still alter protein function
2022-12-05 ペンシルベニア州立大学(PennState)
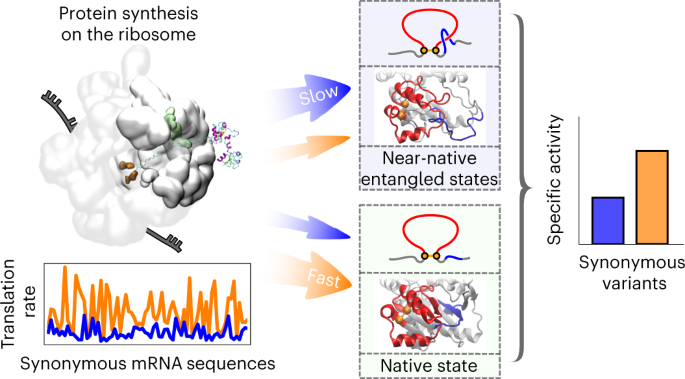
研究チームは、タンパク質を構成するアミノ酸の配列ではなく、タンパク質合成の速度を変える遺伝子の変化が、タンパク質の活性レベルを変えるミスフォールディングにつながることをモデル化し、そのモデルを実験的に裏付けた。この結果は、タンパク質の構造や機能を決定する上で、配列に加えてタンパク質の合成速度であるカイネティクスが重要であることを示しており、合成されたタンパク質の活性を微調整する生物薬剤学などの分野に示唆を与えるものと期待される。
研究チームは、理論と計算を駆使して、タンパク質合成時に分子レベルで起こっていることをシミュレーションするマルチスケールモデリング手法を用い、同義語変異によって起こりうるタンパク質構造の変化を予測し、それによってタンパク質の活性を変化させることに成功した。
3種類の酵素(生化学反応を触媒する特殊なタンパク質)について、高速翻訳コドンからなるmRNAと低速翻訳コドンからなるmRNAをシミュレーションし、新生タンパク質の生成、翻訳後の折り畳み方、活性をモデル化した。
モデルから予測されるタンパク質の構造と折り畳み経路を調べ、活性の変化をもたらしたと思われる分子レベルの変化を特定しようとした。
『非共有結合性投げ縄のもつれ』と呼んでいる、新しいタイプのタンパク質の折り畳み失敗を発見しました。基本的には、タンパク質の一部が閉じたループを形成し、タンパク質の一端が誤ってループを通過してしまい、長い間捕捉されてしまう。
研究者らは、このようなミスフォールディングがタンパク質の活性を低下させる理由として、2つの可能性を指摘している。まず、ミスフォールディングが酵素の活性部位付近で発生し、その活性が阻害される可能性がある。第二に、細胞にはミスフォールディングしたタンパク質を再フォールディングまたは除去するシャペロンという機構があるが、この特定のミスフォールディング構造はシャペロンシステムに認識されないほど微妙なものかもしれず、観察された変化を修正するためにはタンパク質の大部分をアンフォールドする必要があるため、細胞内にとどまることができるのである。
<関連情報>
- https://www.psu.edu/news/eberly-college-science/story/why-synonymous-mutations-are-not-always-silent/
- https://www.nature.com/articles/s41557-022-01091-z
同義性突然変異が酵素の構造と機能を長い時間スケールでどのように変化させるか How synonymous mutations alter enzyme structure and function over long timescales
Yang Jiang,Syam Sundar Neti,Ian Sitarik,Priya Pradhan,Philip To,Yingzi Xia,Stephen D. Fried,Squire J. Booker & Edward P. O’Brien
Nature Chemistry Published:05 December 2022
DOI:https://doi.org/10.1038/s41557-022-01091-z
Abstract
The specific activity of enzymes can be altered over long timescales in cells by synonymous mutations that alter a messenger RNA molecule’s sequence but not the encoded protein’s primary structure. How this happens at the molecular level is unknown. Here, we use multiscale modelling of three Escherichia coli enzymes (type III chloramphenicol acetyltransferase, D-alanine–D-alanine ligase B and dihydrofolate reductase) to understand experimentally measured changes in specific activity due to synonymous mutations. The modelling involves coarse-grained simulations of protein synthesis and post-translational behaviour, all-atom simulations to test robustness and quantum mechanics/molecular mechanics calculations to characterize enzymatic function. We show that changes in codon translation rates induced by synonymous mutations cause shifts in co-translational and post-translational folding pathways that kinetically partition molecules into subpopulations that very slowly interconvert to the native, functional state. Structurally, these states resemble the native state, with localized misfolding near the active sites of the enzymes. These long-lived states exhibit reduced catalytic activity, as shown by their increased activation energies for the reactions they catalyse.