マウスとヒトのデータを統合した新たな解析手法の開発
2018-08-06 理化学研究所,東京医科歯科大学,東北大学東北メディカル・メガバンク機構
理化学研究所(理研)生命医科学研究センター医科学数理研究チームの角田達彦チームリーダーらの共同研究グループ※は、マウスとヒトのデータを統合的に解析することで、アルツハイマー病の原因遺伝子を新たに同定しました。
本研究成果は、アルツハイマー病の発症メカニズムのさらなる解明、そして疾患関連遺伝子探索のためのヒトとモデル動物とのトランスレーショナル研究[1]の発展に貢献すると期待できます。
今回、共同研究グループは、アルツハイマー病の原因となる未知の遺伝子を同定するために、マウスの遺伝子発現と表現型の関連データと、ヒトのアルツハイマー病のゲノムワイド関連解析(GWAS)[2]のデータとを統合的に解析しました。その結果、これまでにアルツハイマー病との関係が知られていない遺伝子を新たに検出しました。このように、ヒトとモデル動物からの異なるタイプのデータを統合解析することで、これまでヒトのGWASだけでは検出されなかった新しい疾患関連遺伝子を発見できる可能性があることが明らかになりました。今回開発した手法は、他の疾患の研究にも応用できると考えられます。
本研究は、国際科学雑誌『Human Genetics』(7月号)の掲載に先立ち、オンライン版(7月13日付け)に掲載されました。
※共同研究グループ
生命医科学研究センター
医科学数理研究チーム
チームリーダー 角田 達彦(つのだ たつひこ)
(東京医科歯科大学 難治疾患研究所 ゲノム応用医学研究部門 医科学数理分野 教授)
研究員(研究当時) 山口(加畑) 由美(やまぐち(かばた) ゆみ)
(現 東北大学 東北メディカル・メガバンク機構 助教)
客員研究員 重水 大智(しげみず だいち)
統計解析研究チーム
客員主管研究員 高橋 篤(たかはし あつし)
統合生命医科学研究センター(研究当時)
副センター長 久保 充明(くぼ みちあき)
大阪大学大学院 医学系研究科
精神医学教室
寄付講座教授 森原 剛史(もりはら たかし)
助教(研究当時) 林 紀之(はやし のりゆき)
名誉教授 武田 雅俊(たけだ まさとし)
教授 池田 学(いけだ まなぶ)
九州大学大学院 医学研究院
衛生・公衆衛生学分野
教授 二宮 利治(にのみや としはる)
助教 小原 知之(おはら ともゆき)
名古屋市立大学大学院 医学研究科
特任教授 赤津 裕康(あかつ ひろやす)
福祉村病院 神経病理研究所
所長 橋詰 良夫(はしず めよしお)
※研究支援
本研究は、文部科学省「脳科学研究戦略推進プログラム」課題F「革新的技術を活用し、加齢による脳機能低下と異常蛋白蓄積につながる病理過程の上流を追及・解明し、認知症の血液診断マーカーと治療薬を開発する(ヒトGWASとモデル動物トランスクリプトームの統合的解析による新規アルツハイマー病関連遺伝子の同定)」による支援を受けて行われました。
背景
ヒトのある表現型[3]に関わる遺伝子を同定する際に、マウスなどの動物モデルを利用すると、遺伝的背景をそろえるなど実験条件をコントロールできるというメリットがあります。しかし、同定された遺伝子がヒトの表現型にどのように影響するかについてまでは確かめることはできません。一方で、ヒトのゲノムワイド関連解析(GWAS)は、多因子疾患や形質の関連遺伝子の同定において強力な手法で、現在、盛んに行われており、同定された疾患関連座位(ゲノム領域)の知見が蓄積されています。しかし、低頻度のゲノム変異の効果を統計的に検出しにくいなどの限界があり、また、同定された遺伝子がどのように発病メカニズムや形質に影響するかについては、多くの場合、さらなる研究が必要です。したがって、ある疾患の原因遺伝子をさらに同定する方法として、ヒトを対象とした研究と動物モデルを用いる研究、それぞれのメリットを生かした「トランスレーショナル研究」が考えられます。
認知症の大部分を占めるアルツハイマー病は、一部は家族性ですが、多くの場合は孤発性の多因子疾患です。孤発性のアルツハイマー病の遺伝的リスク要因として、アポリポタンパク質E(APOE)[4]が知られていますが、他の遺伝的な要因についてはまだよく分かっていません。脳内にアミロイドβペプチド(Aβ)[5]が出現することが、発病につながる初期の現象として知られています。しかし、Aβ蓄積の原因となるさまざまな要因についての全体像はまだ明らかになっていません。
そこで、アルツハイマー病研究をさらに推進するため、ヒトのデータとモデル動物のデータの両方を活用した有効なアプローチに期待がかかります。ヒトとマウスのトランスレーショナル研究において、トランスクリプトーム[6](全遺伝子発現)を活用することは種間の保存性を活用できるというメリットがあります。大阪大学による先行研究では、アルツハイマー病への感受性が異なるマウスの複数の系統を用いたトランスクリプトームデータと、遺伝子改変マウスを用いた遺伝子発現量とAβ蓄積との関係性のデータが得られています注1)。
今回、共同研究グループは、アルツハイマー病に関わる遺伝子をさらに同定するために、マウスのデータとヒトのデータを統合的に解析する新しい手法の開発を試みました。
注1)Morihara et al. 2014. Transcriptome analysis of distinct mouse strains reveals kinesin light chain-1 splicing as an amyloid-beta accumulation modifier. Proc Natl Acad Sci U S A 111: 2638-43.
研究手法と成果
共同研究グループは、この統合解析に向けて、マウスについては2段階の解析から得られたデータを用いました。まず、アルツハイマー病感受性が異なる複数の非遺伝子組換えマウス系統を比較し、遺伝子発現レベルに有意差を示す373の遺伝子を検出しました。アルツハイマー病の二次的な影響を受けていないため、これらには疾患の原因遺伝子の候補が含まれている可能性があります。次に、Aβ蓄積というアルツハイマー病理を再現する疾患モデル動物としてアミロイド前駆体タンパク質(APP)[5]をコードするAPP遺伝子の改変マウスを、アルツハイマー病感受性が異なる複数のマウス系統と交配させ、背景遺伝子を混合させたアルツハイマー病モデル動物を用意しました。このマウスにおいて上記の遺伝子発現量とAβ蓄積量との相関を調べ、相関係数のP値[7]を出しました。また、ヒトについては、日本人におけるアルツハイマー病のGWASの統計量のSNP単位のP値を、遺伝子単位の統計量のP値に変換しました。次に、マウスとヒトとの同祖遺伝子[8]ペアで結合し、逆正規法[9](StoufferのZ-スコア法)により、マウスとヒトからのP値から統合P値を計算しました。そして候補遺伝子を統合P値により評価し、統計的に有意と判断された遺伝子を検出するというアプローチを提案しました(図1)。
このアプローチの有効性を確かめるために、既報の大規模なヨーロッパ系集団のアルツハイマー病のGWAS(アルツハイマー病ゲノミクス国際プロジェクトコンソーシアム(IGAP)[10]:患者17,008人と対照者37,154人)での一塩基多型(SNP)[11]単位の統計量データを用いました。有意性で上位のSNPを持つ遺伝子を調べたところ、マウスで得られた373遺伝子のうち11遺伝子がヒトと共通であることが分かりました。これらの遺伝子について、マウスのデータとの統合解析を行ったところ、大部分の遺伝子において有意性が増加しました。これにより、提案したアプローチが有効であると判断しました。
次に、日本人のアルツハイマー病(患者811人と対照者7,504人)のGWASデータ注2)を活用し、SNP単位の統計量をP値に変換しました。そして、マウスのデータとの統合解析を行ったところ、統合されたP値から統計的に有意である5つの遺伝子が検出できました(図2)。
この5遺伝子について、ヒトの剖検脳(患者10人と対照者13人)を用いて、アルツハイマー病患者群と対照群との間で遺伝子発現量に差があるか否かを調べました。その結果、二つの遺伝子(LBH遺伝子とSHF遺伝子)について、対照群に比べアルツハイマー病患者群において有意に発現量が低くなることが分かりました(図3)。この傾向はマウスのデータとも一致し、Aβ蓄積量と遺伝子発現量は負の相関を示しました。これは、LBHとSHFの遺伝子産物が、疾患発症のメカニズムにおいて「抑制的な効果」を持つことを示しています。
LBH遺伝子は、転写因子[12]をコードしており、マウスの相同遺伝子は、肢芽と心臓の発生に関わることが知られています。また、LBH遺伝子はWntシグナル[13]のターゲットであること、自己免疫疾患[14]の関節リウマチとの関連があることが報告されています。また、SHF遺伝子は、血小板由来増殖因子(PDGF)[15]応答において細胞死を調節する役割を持つことが示されています。SHF遺伝子産物は、未分化リンパ腫キナーゼ[16]と相互作用することで、神経芽細胞腫[17]における下流のシグナルを負に調節することが知られています。
このように、ヒトとモデル動物からの異なるタイプのデータを統合解析することで、これまでヒトのGWASだけでは検出されなかった新しい疾患関連遺伝子を発見できる可能性があることが明らかになりました。また、新たな遺伝子を同定できたことで、これまでに蓄積した様々な知見に基づき、アルツハイマー病の発病メカニズムを解明できる可能性が示されました。
注2)Hirano et al. 2015. A genome-wide association study of late-onset Alzheimer’s disease in a Japanese population. Psychiatr Genet 25: 139-46
今後の期待
疾患に関わる遺伝子を同定するための、異なるデータを組み合わせた統合解析の手法は、まだまだ発展の余地があります。今後、P値だけでなく、効果サイズ[18]の活用や、遺伝子発現に影響するSNPの情報を統合することにより、さらに有効な手法の確立が期待できます。
アルツハイマー病は多因子疾患であるにもかかわらず、現状では研究対象となる分子は限られており、効果的なアルツハイマー病の治療法の開発もいまだ成功していません。今後、今回のようなアプローチをさらに展開することにより、複雑な疾患の発症メカニズムに関わる遺伝子がさらに見つかる可能性があり、これまでにない診断法や治療法開発につながることが期待できます。
原論文情報
Yumi Yamaguchi-Kabata, Takashi Morihara, Tomoyuki Ohara, Toshiharu Ninomiya, Atsushi Takahashi, Hiroyasu Akatsu, Yoshio Hashizume, Noriyuki Hayashi, Daichi Shigemizu, Keith A. Boroevich, Manabu Ikeda, Michiaki Kubo, Masatoshi Takeda, Tatsuhiko Tsunoda, “Integrated analysis of human genetic association study and mouse transcriptome suggests LBH and SHF genes as novel susceptible genes for amyloid-beta accumulation in Alzheimer’s disease.”, Human Genetics, 10.1007/s00439-018-1906-z
発表者
理化学研究所
生命医科学研究センター 医科学数理研究チーム
チームリーダー 角田 達彦(つのだ たつひこ)
(東京医科歯科大学 難治疾患研究所 医科学数理分野 教授)
報道担当
理化学研究所 広報室 報道担当
東京医科歯科大学 総務秘書課 広報係
東北大学東北メディカル・メガバンク機構 報道担当 長神 風二(ながみ ふうじ)
補足説明
-
- トランスレーショナル研究
- 主に医学や生物学の分野において、基礎研究からの有望な知見を選び、臨床研究に至るまでの工程に関わる研究。
-
- ゲノムワイド関連解析(GWAS)
- ゲノムワイド関連解析は、人のゲノム全体をほぼカバーする場所の、一塩基多型(SNP)を代表とする配列多様性の遺伝子型を決定し、遺伝子型の頻度の集団的偏り(特に患者と一般集団との間の)と、病気や表現型との関連を統計的に調べる方法。GWASはGenome Wide Association Studyの略。
-
- 表現型
- ある生物の持つ遺伝子型が形質として表現されたもの。その生物の形態、構造、行動、生理的性質などを含む。
-
- アポリポタンパク質E(APOE)
- リポタンパク質の一種。ヒトでは、アミノ酸配列の違いによって、三つのアイソフォームE2、E3、E4が知られている。なかでも、アイソフォームE4はアルツハイマー病のリスク因子として知られている。遺伝子は19番染色体上に存在し、アルツハイマー病の関連解析において強いシグナルを示す。
-
- アミロイドβペプチド(Aβ)、アミロイド前駆体タンパク質(APP)
- アルツハイマー病脳の最大の特徴は老人斑(アミロイドプラーク)の沈着であり、その構成成分がアミロイドβと呼ばれる40アミノ酸程度のペプチドである。健常者でも加齢とともに脳内にアミロイドβは蓄積するが、アルツハイマー病患者ではその量が著しく多い。アルツハイマー病の脳病理形成はまずアミロイドβが蓄積していき、その後タウタンパク質病理が出現、さらに神経脱落が生じると考えられている。認知症の臨床症状は最終段階である神経脱落がある程度進行した時点で出現する。アミロイドβ蓄積抑制は、開発が待ち望まれているアルツハイマー病の根治的治療法のターゲットでもある。アミロイドβは、ヒトの21番染色体上にあるAPP遺伝子から作られる長いポリペプチドのアミロイド前駆体タンパク質(APP)の部分断片である。
-
- トランスクリプトーム
- ゲノムDNAを鋳型としてRNA分子が合成されることを転写(トランスクリプション)と呼び、転写の結果合成されたRNA分子のことを転写産物と呼ぶ。細胞中にはさまざまなRNA分子がそれぞれの量で存在している。このようなRNA分子の総体を、転写産物を表す「トランスクリプト」に、総体を表す接尾語「オーム」をつなげて「トランスクリプトーム」と呼ぶ。
-
- P値
- 偶然にそのようなことが起こる確率のことで統計学的有意差を示す指標。数値が低いほど有意性が高いことを表す。
-
- 同祖遺伝子
- 異なる生物種の比較において、共通祖先由来の進化的に機能の保存されている遺伝子で、種分化によって分岐したもの。
-
- 逆正規法
- 複数のP値を統合する方法の一つ。独立に観測された複数のP値を統合する際に、まずそれぞれのP値に対応するZ-スコアを、標準正規分布累積関数の逆関数から求める。統合されたZ-スコアを計算し、それに対応するP値を標準正規分布累積関数から得る。
-
- アルツハイマー病ゲノミクス国際プロジェクトコンソーシアム(IGAP)
- アルツハイマー病の原因となる遺伝要因の同定を推進するため、ヨーロッパと米国の研究機関で構成された組織。IGAPはInternational Genomics of Alzheimer’s Projectの略。
-
- 一塩基多型(SNP)
- 集団のゲノム配列中でみられる一塩基の違い(個人差、多様性)で、特に、集団の1%以上に違いが認められる場合に、一塩基多型と呼ぶ。SNPはSingle Nucleotide Polymorphismの略。
-
- 転写因子
- DNAに配列特異的に結合するタンパク質で、プロモーターやエンハンサーといった転写制御領域に結合し、RNAポリメラーゼによる遺伝子の転写を活性化あるいは不活性化する。
-
- Wntシグナル
- Wntタンパク質が細胞膜受容体に結合したした後に引き起こされる細胞内でのシグナル伝達。ショウジョウバエで翅のない変異体(wingless)が見いだされたことから、研究が進んだ。多くの生物種によって保存されている。ウィントシグナルを読む。
-
- 自己免疫疾患
- 自己免疫疾患は、何らかの免疫異常によって自分の体や組織を異物のように認識し、自己抗体や自己に反応するリンパ球を作り、自分の体を攻撃する疾患のこと。代表的なものに関節リウマチがある。
-
- 血小板由来増殖因子(PDGF)
- 血小板由来増殖因子。細胞の遊走および増殖などの調節に関与する増殖因子。チロシンキナーゼ関連型受容体の一種PDGFRを介して、生理学機能を発揮する。PDGFはplatelet-derived growth factorの略。
-
- 未分化リンパ腫キナーゼ
- 2番染色体上に遺伝子があり、その産物はそのままの形ではレセプターチロシンキナーゼである。脳の発生や特異的なニューロンに重要な役割を持つ。染色体の転座や遺伝子融合などにより、がんを起こすことが知られている。
-
- 神経芽細胞腫
- 小児がんの一つ。交感神経節や副腎髄質などから発生する。
-
- 効果サイズ
- GWASにおいての意味は、ゲノム上のSNPなどの遺伝子多型の遺伝子型の違いと、疾患群と対照群の間の表現型の差との、関連の強さを表したもの。
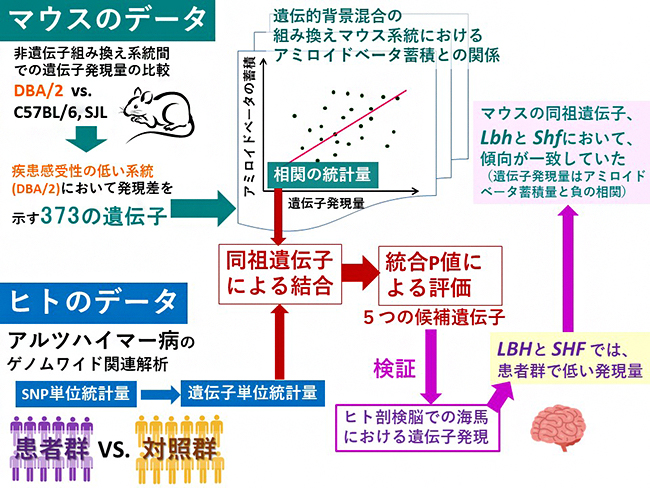
図1 アルツハイマー病の原因遺伝子を同定するためのマウスとヒトのデータの統合解析
マウスについては2段階の解析から得られたデータを用いた。まず、アルツハイマー病低感受性の複数の非遺伝子組換えマウス系統を比較し、遺伝子発現レベルに有意差を示す373の遺伝子を検出した(左上)。次に、遺伝的背景を混合したアミロイド前駆体タンパク質(APP)をコードするAPP遺伝子の改変マウスを用いて、上記の遺伝子発現量とAβ蓄積量との相関を調べ、相関係数のP値を出した(中央上)。ヒトについては、日本人におけるアルツハイマー病のGWASの統計量をP値(遺伝子単位の統計量)に変換した(左下)。マウスとヒトとの同祖遺伝子ペアで結合し、逆正規法により、マウスとヒトからのP値から統合P値を計算した(中央)。そして候補遺伝子を統合P値により評価し、統計的に有意と判断された遺伝子を検出した。ヒトの剖検脳を用いて、アルツハイマー病患者と対照群でこれら遺伝子の発現量に差があるかどうかを調べた(右下)。
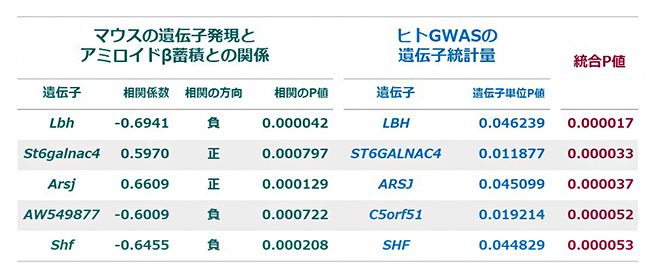
図2 マウスとヒトのデータの統合解析の上位遺伝子
統合P値により、373遺伝子から統計的に有意と判断された5つの遺伝子の結果を示している。
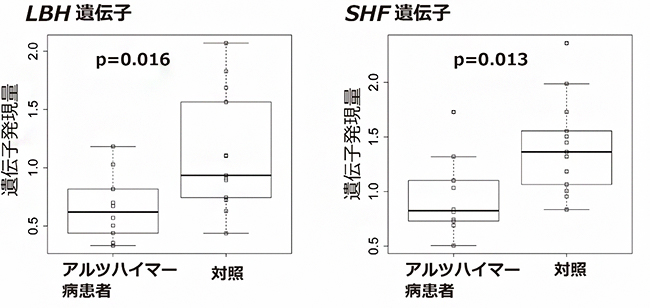
図3 ヒト剖検脳における遺伝子発現量の比較
統合解析において統計的に有意と判断された5つの遺伝子について、ヒト剖検脳における遺伝子発現量が患者群(10人)と対照群(13人)で異なっているか否かを調べた。5つの遺伝子の中で、LBH遺伝子とSHF遺伝子においては、患者群では対照群よりも遺伝子発現量が低いことが分かった。