RNA合成を一時的に停止させる構造変化を低温電子顕微鏡で解明 Researchers use cryo-electron microscopy to reveal structural changes that temporarily shut down RNA synthesis
2023-02-09 ペンシルベニア州立大学(PennState)
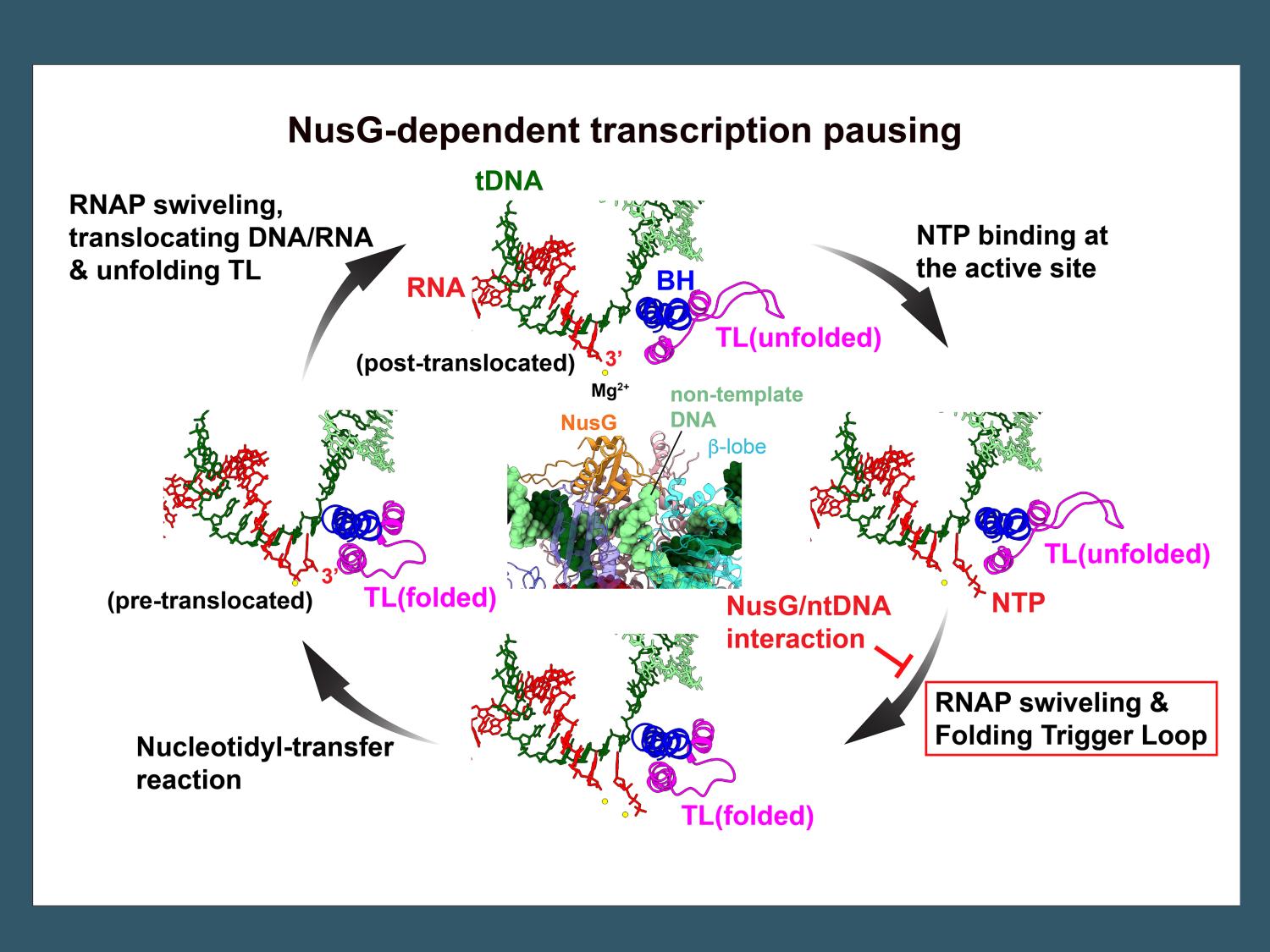 Researchers have worked out the mechanism of transcription pausing in some bacteria by determining the structures of RNA polymerase in a complex with a DNA template, an RNA product, and a protein factor using cryo-electron microscopy (cryo-EM). The nucleotide addition cycle of bacterial RNA polymerase (RNAP) is depicted by cartoon models of template DNA (tDNA, green), RNA (red), trigger loop (TL, magenta), bridge helix (BH, blue), incoming NTP (red) and Mg2+ (yellow spheres) together with the conformational changes of RNAP (swiveling, folding Trigger loop) associated with each step of the nucleotide addition cycle. NusG and non-template DNA (ntDNA, light green) interaction (center panel) inhibits the RNAP swiveling, the Trigger loop folding allosterically thus pauses RNA synthesis. Credit: Murakami Laboratory / Penn State. Creative Commons
Researchers have worked out the mechanism of transcription pausing in some bacteria by determining the structures of RNA polymerase in a complex with a DNA template, an RNA product, and a protein factor using cryo-electron microscopy (cryo-EM). The nucleotide addition cycle of bacterial RNA polymerase (RNAP) is depicted by cartoon models of template DNA (tDNA, green), RNA (red), trigger loop (TL, magenta), bridge helix (BH, blue), incoming NTP (red) and Mg2+ (yellow spheres) together with the conformational changes of RNAP (swiveling, folding Trigger loop) associated with each step of the nucleotide addition cycle. NusG and non-template DNA (ntDNA, light green) interaction (center panel) inhibits the RNAP swiveling, the Trigger loop folding allosterically thus pauses RNA synthesis. Credit: Murakami Laboratory / Penn State. Creative Commons
◆このたび、研究チームは、低温電子顕微鏡(クライオ電子顕微鏡)を用いて、RNAポリメラーゼがRNA合成を停止する前、停止中、停止直後の構造を原子レベルで明らかにし、いくつかの細菌で転写が停止する機構を解明した。転写を一時停止するメカニズムを解明することは、細胞の基本的な機能を理解する上で極めて重要である。
◆今回の研究で明らかになった転写の一時停止の仕組みは、地球上のすべての生物の遺伝子制御を理解するために広く応用できる可能性がある。また、この知見は、転写の一時停止を標的として阻害し、適切な遺伝子発現と細胞機能を阻害する新しい抗菌剤の同定に利用できる可能性がある。
◆ペンシルベニア州立大学の科学者チームによるこの研究は、2月6日発行の米国科学アカデミー紀要に掲載されます。
◆ペンシルベニア州立大学の村上和彦教授(生化学・分子生物学)は、「細胞が正しく機能するためには、遺伝子の発現を正確に制御して、選択したタンパク質や機能性RNAを適切な量、適切なタイミングで作る必要があります」と語っている。「RNAポリメラーゼが活発にRNAをつくっているときにも、転写の一時停止など、さまざまな方法で遺伝子発現が制御されることがあるのです。ここでは、低温電子顕微鏡による構造決定と構造に基づく生化学的アッセイにより、RNAポリメラーゼ、DNA、NusG間の相互作用、およびRNAポリメラーゼが転写中に一時停止したときに起こるRNAポリメラーゼの構造変化を明らかにしました」と述べている。
◆RNAポリメラーゼは、いくつかの機能ドメインからなる分子機械である。また、他の多くのタンパク質補因子と相互作用し、RNAが生成されるタイミングと量を調節するのに役立っている。ペンシルベニア州立大学のスタンレー・パーソン教授(分子生物学)の研究室で、研究チームのリーダーを務めるポール・バビツケは、これらの補因子の1つであるNusGというタンパク質が、転写を一時停止するのに重要であることを明らかにしている。
◆研究チームは、NusGの存在下で転写を一時停止させるDNAとRNAの配列を特定し、DNA、RNA、NusGをRNAポリメラーゼと共に用いて、転写複合体を再構成しています。このサンプルを凍結し、ペンシルベニア州立ハック生命科学研究所の最新鋭のクライオ電子顕微鏡施設を使って、クライオ電子顕微鏡による構造研究を行った。
◆研究チームは、一時停止した転写複合体の構造から、NusGが非鋳型DNA鎖を、NusGとRNAポリメラーゼのβローブというドメインの間の狭い空洞に挿入することによって相互作用していることを明らかにした。しかし、この相互作用はRNAポリメラーゼの活性部位から遠く離れた場所で起こっており、どのようにして転写を一時停止させているのだろうか。研究チームは、低温電子顕微鏡による構造研究によって得られた独自の情報を用いて、この疑問に迫った。
<関連情報>
- https://www.psu.edu/news/eberly-college-science/story/dna-stuck-gears-rna-production-machine/
- https://www.pnas.org/doi/10.1073/pnas.2218516120
NusG依存的なRNAポリメラーゼの休止による転写阻害のアロステリック機構 Allosteric mechanism of transcription inhibition by NusG-dependent pausing of RNA polymerase
Rishi K. Vishwakarma, M. Zuhaib Qayyum, Paul Babitzke and Katsuhiko S. Murakami
Proceedings of the National Academies of Science Published:February 6, 2023
DOI:https://doi.org/10.1073/pnas.2218516120
Significance
Transcription pausing by RNA polymerase (RNAP) regulates gene expression where it controls co-transcriptional RNA folding, synchronizes transcription with translation, and provides time for binding of regulatory factors. Transcription elongation factor NusG stimulates pausing in Gram+ bacteria including Bacillus subtilis and Mycobacterium tuberculosis by sequence-specific interaction with a conserved pause motif found in the non-template DNA (ntDNA) strand within the transcription bubble. Our structural and biochemical results revealed that part of the conserved TTNTTT motif in ntDNA is extruded and sandwiched between NusG and RNAP. Our results further demonstrate that an essential global conformational change in RNAP is directly linked to RNA synthesis and that the NusG–ntDNA interaction pauses RNA synthesis by interfering with this conformational change.
Abstract
NusG is a transcription elongation factor that stimulates transcription pausing in Gram+ bacteria including B. subtilis by sequence-specific interaction with a conserved pause-inducing –11TTNTTT-6 motif found in the non-template DNA (ntDNA) strand within the transcription bubble. To reveal the structural basis of NusG-dependent pausing, we determined a cryo-EM structure of a paused transcription complex (PTC) containing RNA polymerase (RNAP), NusG, and the TTNTTT motif in the ntDNA strand. The interaction of NusG with the ntDNA strand rearranges the transcription bubble by positioning three consecutive T residues in a cleft between NusG and the β-lobe domain of RNAP. We revealed that the RNAP swivel module rotation (swiveling), which widens (swiveled state) and narrows (non-swiveled state) a cleft between NusG and the β-lobe, is an intrinsic motion of RNAP and is directly linked to trigger loop (TL) folding, an essential conformational change of all cellular RNAPs for the RNA synthesis reaction. We also determined cryo-EM structures of RNAP escaping from the paused transcription state. These structures revealed the NusG-dependent pausing mechanism by which NusG-ntDNA interaction inhibits the transition from swiveled to non-swiveled states, thereby preventing TL folding and RNA synthesis allosterically. This motion is also reduced by the formation of an RNA hairpin within the RNA exit channel. Thus, the pause half-life can be modulated by the strength of the NusG-ntDNA interaction and/or the stability of the RNA hairpin. NusG residues that interact with the TTNTTT motif are widely conserved in bacteria, suggesting that NusG-dependent pausing is widespread.